
2019-08-27
QIIME全称是Quantitative Insights Into Microbial Ecology,是由微生物组领域大神Rob Knight领导团队开发的微生态测序数据分析流程,于2010年发表在《Nature Methods》期刊上。9年来,该软件已成为微生物组领域广泛使用的分析工具,引用量过万,更是在今年的6月17日,被《Nature》期刊评为近70年来人体微生物组研究的25个里程碑事件之一。

QIIME软件入选《Nature》评出的人体微生物组研究25大里程碑事件
(https://www.nature.com/immersive/d42859-019-00041-z/index.html)
9年之后,QIIME再次出发!为满足当前规模日益庞大的数据、以及分析可重复、可追溯的需求,QIIME论文一作、现北亚利桑那大学的Gregory Caporaso教授牵头,并与全世界79家单位的112名同行联合,从头开发了QIIME 2分析平台,论文于2019年7月24日在线发表于世界顶级学术期刊《Nature Biotechnology》!

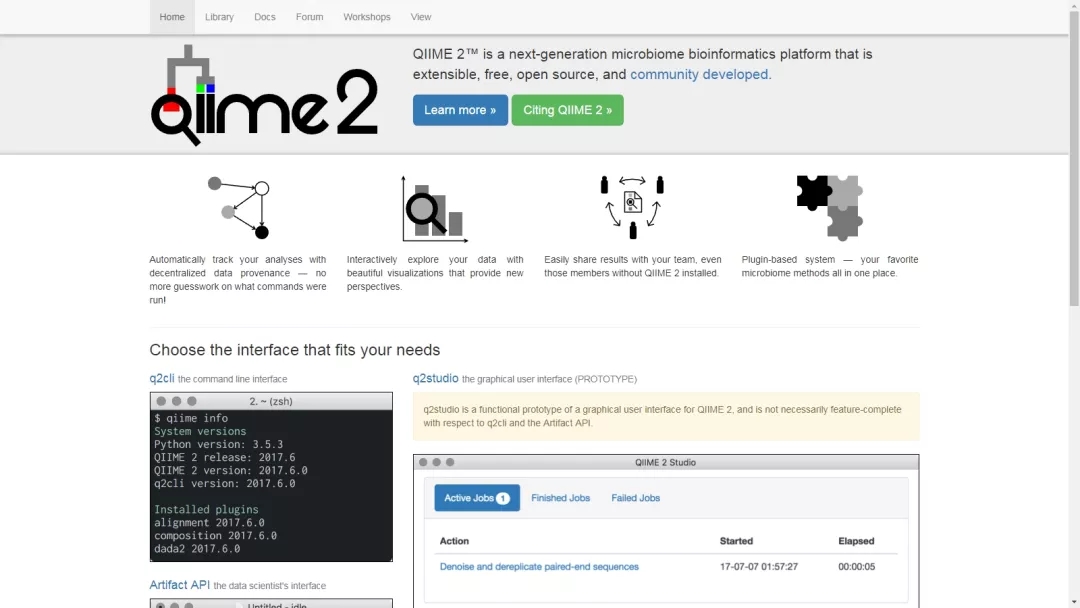
QIIME 2正式发表:可重复、可交互、适用范围广并且可扩展的微生物组数据科学
总体而言,QIIME 2对QIIME 1完全重新设计和重写,是全新的微生物组分析流程,不但继承了QIIME 1强大和广泛使用的诸多优点,同时也改进了先前版本中的许多不足和问题。一方面,QIIME 2可以整合多种分析流程、自动化追踪数据来源;同时,它也支持API、命令行、图形界面等多种用户界面。另外,QIIME 2还开发了“语义类型系统(Semantic types)”,自动识别输入文件类型;还可以通过插件系统,不断新增微生物组分析方法、扩展使用功能(插件的实质是软件包,比如dada2、q2-longitudinal等):QIIME 2制定了分析插件的标准化开发流程,每个人都可以开发,官方也鼓励第三方工具作为插件以为QIIME 2提供各种额外的分析功能,从而实现“去中心化”,使技术、方法得以快速部署、整合。
QIIME 2优势
1 每一步分析结果可追溯、可重复
2 插件系统赋予强大的可扩展性
3 全新可交互式图形系统,可视化功能更强大
4 安装更方便
5 使用方式更多样
6 合作共享更容易
7 完善的社区平台
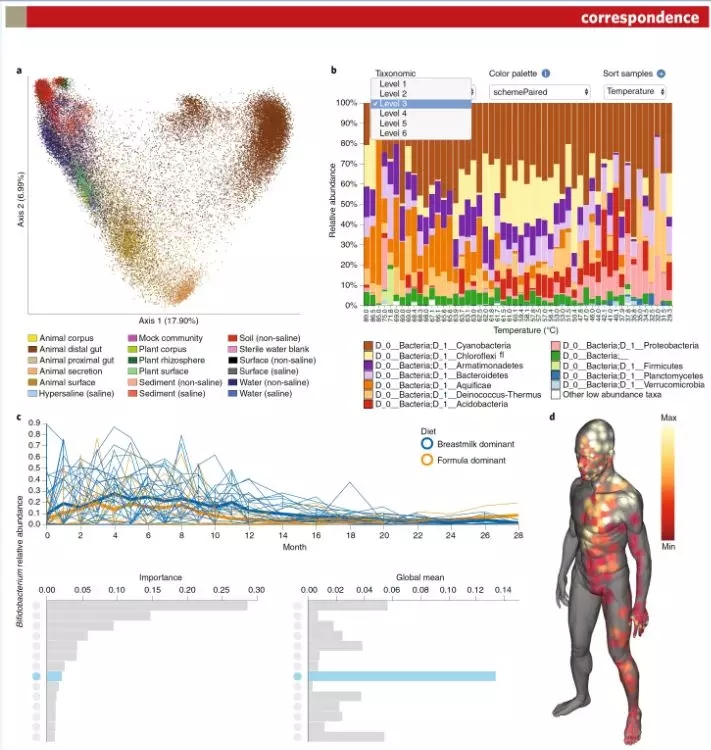
QIIME 2提供了多种多样的交互式可视化工具
(https://github.com/qiime2/paper1)
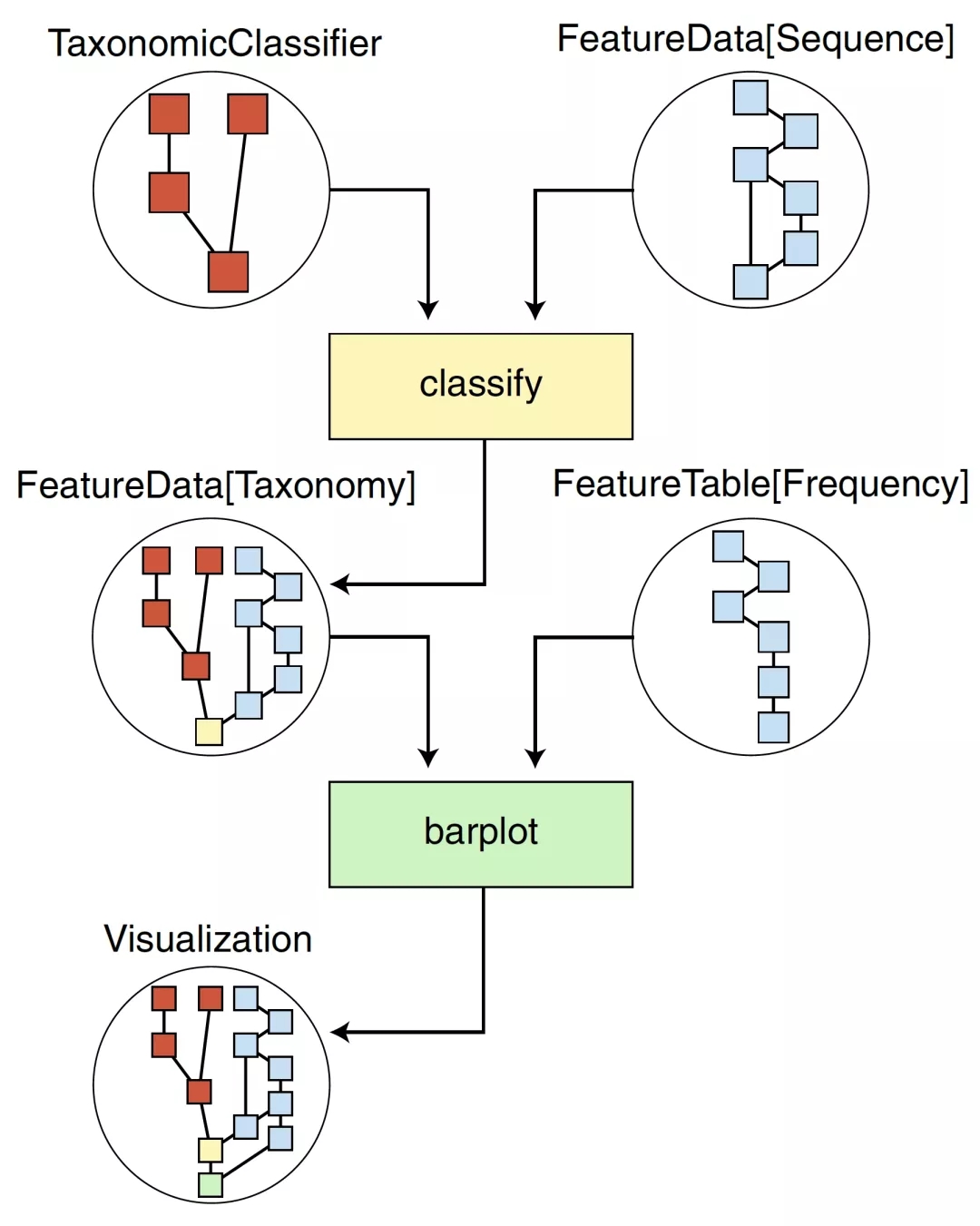
QIIME 2迭代记录数据来源,确保分析可追溯、可重复
QIIME 2从提出概念,到正式发表论文,已经经过了很长时间的迭代开发,目前已具备了完善的分析流程(详见https://docs.qiime2.org/2019.7/tutorials/overview/):
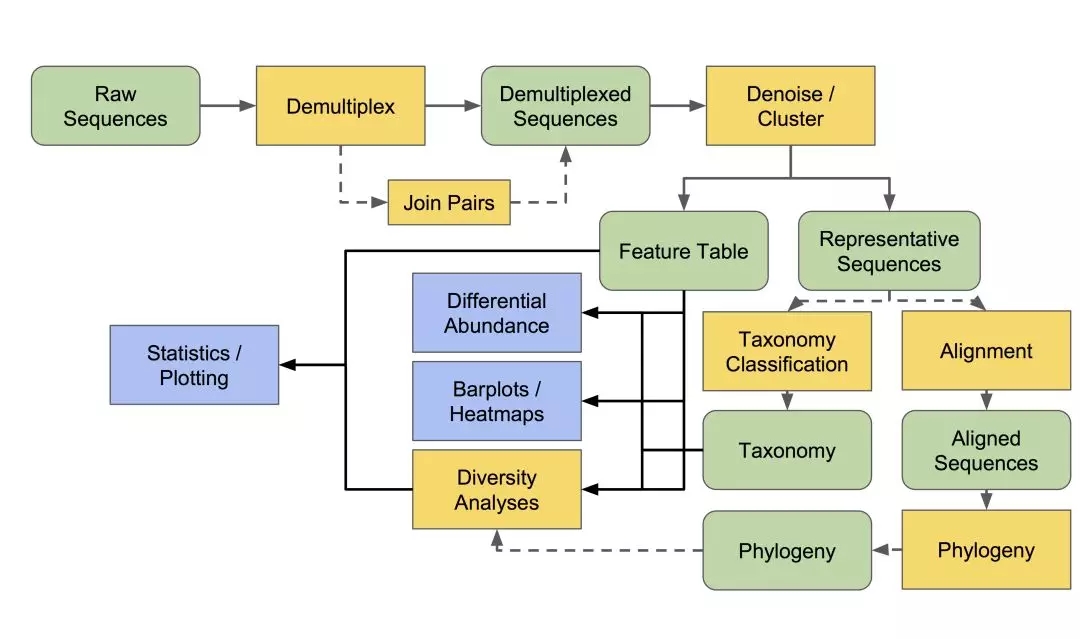
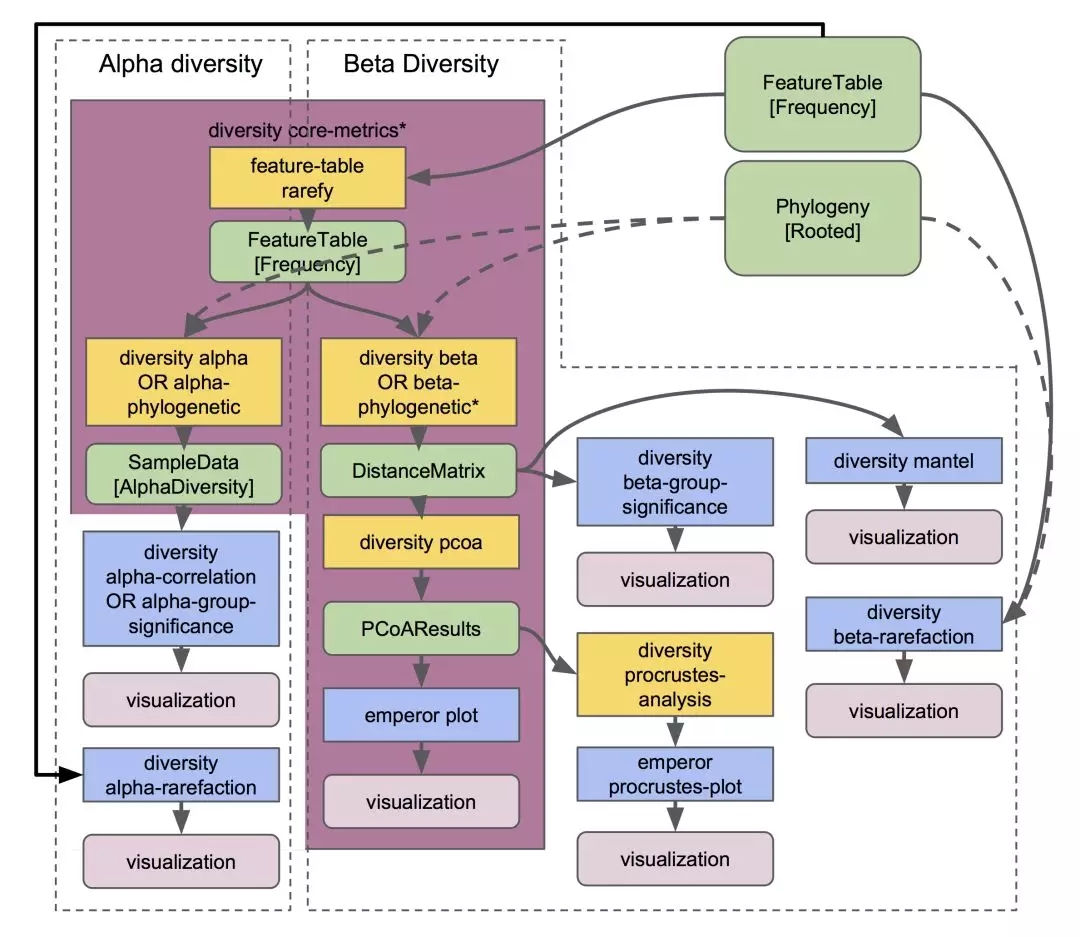
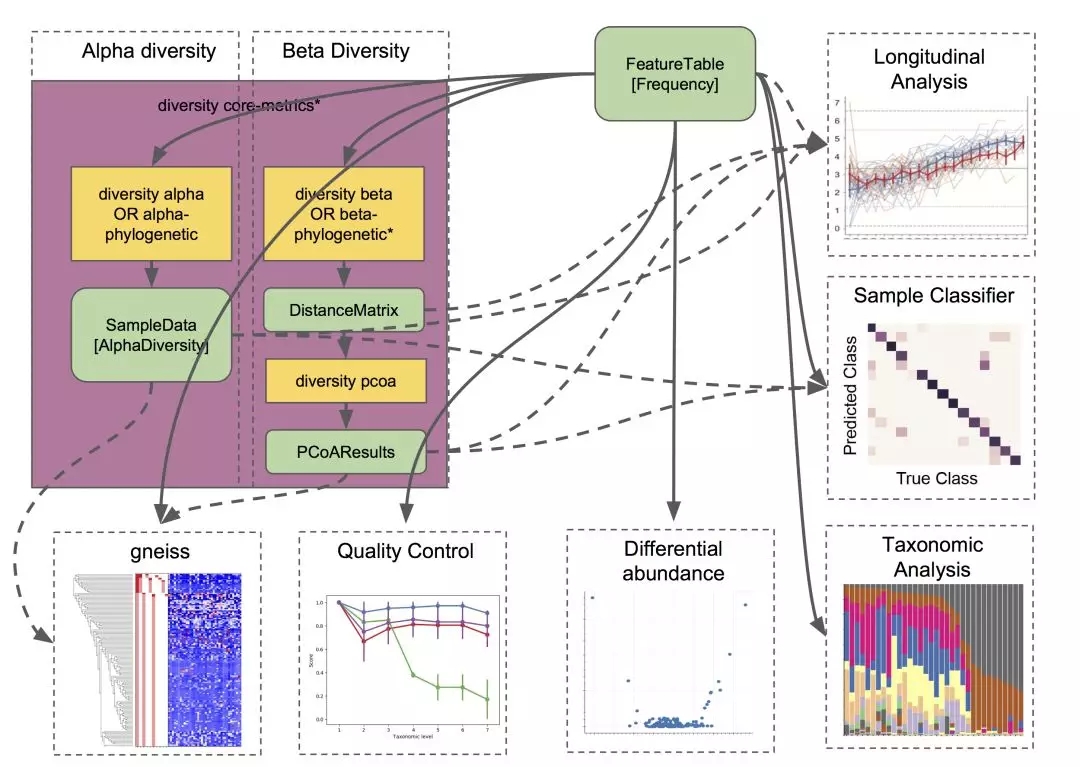
QIIME 2产生的数据类型,叫做对象(Artifacts),它可以是序列数据、表格数据、树文件、样本信息、参数信息等等。
在QIIME 2中,每一个特定的功能都是由一个插件完成的,而QIIME 2构建的基本思想,就是将这些插件的输入端和输出端统一为QZA和QZV的文件格式,进而可以衔接起来:
▶ qza文件的本质其实是一个文件压缩包,我们可以简单得将QZA文件理解为是Qiime Zipped Artifacts。QZA文件除了包含数据外,也包含了之前的分析过程、使用的方法命令、使用的数据等信息等,得实现分析步骤的可追溯、可重复;
▶ qzv文件末尾的v代表visual。我们可以简单得将QZV文件理解为是Qiime Zipped Visual。它与QZA文件类似,可追溯、可重复,唯一的区别就在于它是各个分析流程的终点,即无法再使用QZV文件作为输入文件在流程中继续分析。QZV文件包含的可视化结果有:统计表格、静态图片、交互式网页以及组合的可视化呈现。
QIIME 2 插件亮点
之DADA2
根据目前的QIIME 2官方的技术文档,目前QIIME 2已经包含20余种插件:
1. alignment: Plugin for generating and manipulating alignments
2. composition: Plugin for compositional data analysis
3. cutadapt: Plugin for removing adapter sequences, primers, and other unwanted sequence from sequence data
4. dada2: Plugin for sequence quality control with DADA2
5. deblur: Plugin for sequence quality control with Deblur
6. demux: Plugin for demultiplexing & viewing sequence quality
7. diversity: Plugin for exploring community diversity
8. emperor: Plugin for ordination plotting with Emperor
9. feature-classifier: Plugin for taxonomic classification
10. feature-table: Plugin for working with sample by feature tables
11. fragment-insertion: Plugin for extending phylogenies
12. gneiss: Plugin for building compositional models
13. longitudinal: Plugin for paired sample and time series analyses
14. metadata: Plugin for working with Metadata
15. phylogeny: Plugin for generating and manipulating phylogenies
16. quality-control: Plugin for quality control of feature and sequence data
17. quality-filter: Plugin for PHRED-based filtering and trimming
18. sample-classifier: Plugin for machine learning prediction of sample metadata
19. taxa: Plugin for working with feature taxonomy annotations
20. types: Plugin defining types for microbiome analysis
21. vsearch: Plugin for clustering and dereplicating with vsearch
由于篇幅所限,我们无法一一展示QIIME 2的插件系统的强大之处。我们在此重点讨论下DADA2这一插件。

DADA2可以实现Illumina扩增子测序数据的错误校正,去除引物、碱基质量控制、去噪(Denoise)、双端序列拼接和嵌合体去除,进而获得单碱基精度的代表序列。与传统的基于OTU的分析方法不同,DADA2不再以序列相似度进行聚类,只进行去重(Dereplication,相当于以100%相似度聚类),从而得到“扩增序列变体”ASVs(Amplicon sequence variants),或称为“特征序列”(对应于传统的OTU代表序列),而这些序列在样本中的丰度表称为“特征表”(对应于传统的OTU丰度矩阵表)。以DADA2为代表的去噪生成特征序列的方法,是目前主流分析平台(QIIME 2和USEARCH等)所力推的。
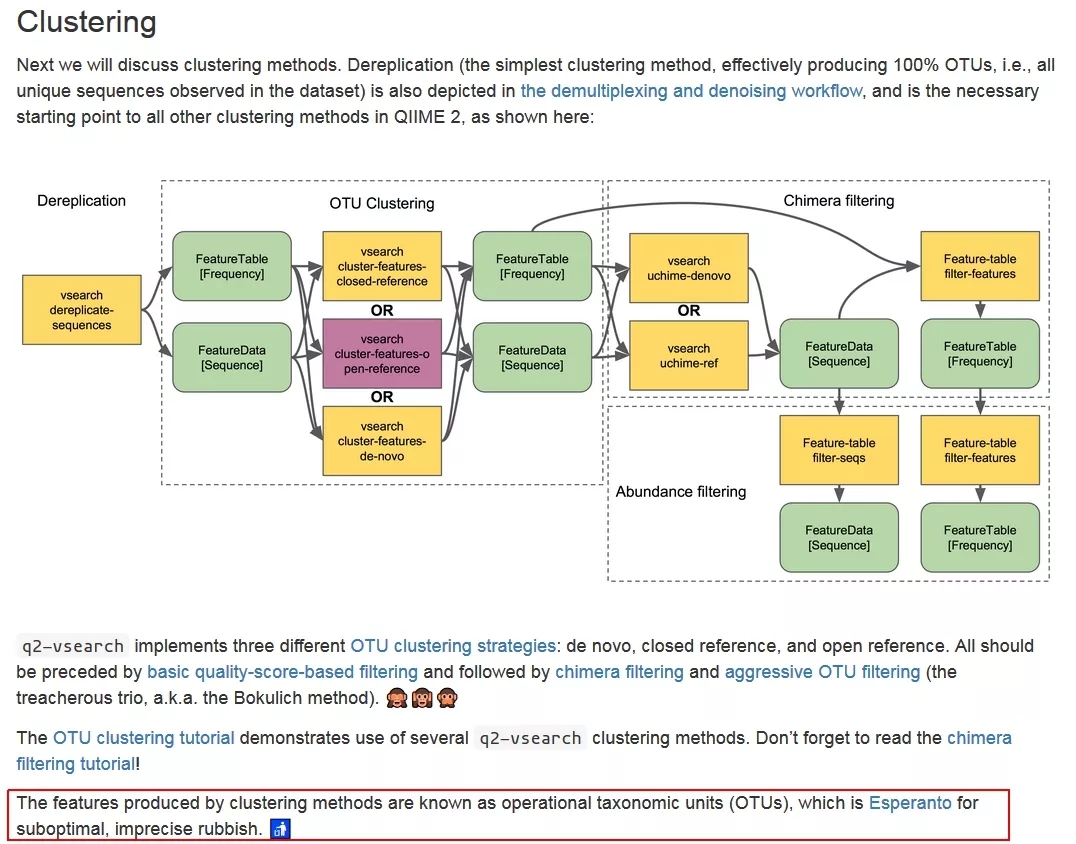
QIIME 2认为以OTUs聚类为基础建立的分析方法是不理想、不准确的
(https://docs.qiime2.org/2019.7/tutorials/overview/#denoising-and-clustering)
此外,QIIME 2还整合了新的条形UniFrac算法(Striped UniFrac),也大大提升了微生物组大数据的分析速度。
综上所述,我们纵览了QIIME 2的优点和诸多新特性。我们相信,QIIME 2的诞生,必将推动微生物组研究进入快速发展的新时期!作为微生物组大数据解析的一大利器,QIIME 2可追溯、可重复的“数据透明化”的特点,使其必将成为微生态领域研究人员熟知和接受度广的行业标准!未来,QIIME 2还将纳入宏基因组、宏转录组、宏蛋白组和代谢组等分析流程,可以预期,在不久的将来,QIIME 2将发展成为多组学整合分析平台!