2019-09-20

《QIIME 2:微生物组大数据挖掘的利器!》的科普介绍发布至今,很多老师都非常关注!
现在进一步的消息来了:即日起,派森诺微生物组事业部正式上线以QIIME2为的分析流程,喜大普奔!

新版的结果报告内容条理更加清晰紧凑,分析内容紧跟时代潮流,更加有利于对分析结果的理解和挖掘,基础分析+常规分析+个性化分析将助力您发表高分文章。
02、QIIME2数据处理流程 DADA2 / Vsearch + 物种注释等各种插件
我们已经介绍过,QIIME2具有强大的可扩展性,目前已经包含了众多插件,涵盖序列处理、物种注释、多样性计算等多方面。比如,在序列处理方面,有DADA2、Deblur、Vsearch等插件,其中DADA2不再以序列相似度进行聚类,而是通过“去重复”(Dereplication)等步骤,得到“扩增序列变体”ASV(Amplicon sequence variant),进而获得单碱基精度的代表序列,提高了数据准确度与物种分辨率。
另一方面,以DADA2为首的生成ASV的方法,目前尚不能与所有扩增子类型项目适配,所以我们同样保留了基于OTU聚类的Vsearch方法作为备选,该软件的聚类和去嵌合体准确率均优于USEARCH的uparse算法。
03、分析可视化效果进一步升级
包括物种组成柱形图、Graphlan、网络分析、热图分析、Beta多样性分析、差异分析等等,我们基于QIIME2进一步升级了可视化展示效果!同时,我们还可以通过QIIME2的可视化统一展示平台 https://view.qiime2.org/,“一步到位”,获取发表水准的酷炫图表!
04、多种展现形式
我们提供了多种出图形式,可供大家选择,比如多种Alpha/Beta多样性计算方法、多种排序图类型、多种热图展现形式(是否分组、是否聚类等等)、以及多种差异物种筛选方法!
05、功能潜能预测分析 —— PICRUSt 2.0
功能预测分析的PICRUSt目前已升级到2.0版本,等您来体验!与初代版本相比,PICRUSt 2.0不仅能对16S rRNA基因序列进行功能潜能预测,还能对真菌ITS序列和18S rRNA基因序列进行预测,同时新增了MetaCyc等功能数据库参考源,比初代版本的参考数据库更全、预测分析更准确,还可推断物种来源与预测到功能的对应关系,可谓增强了不止一点点!
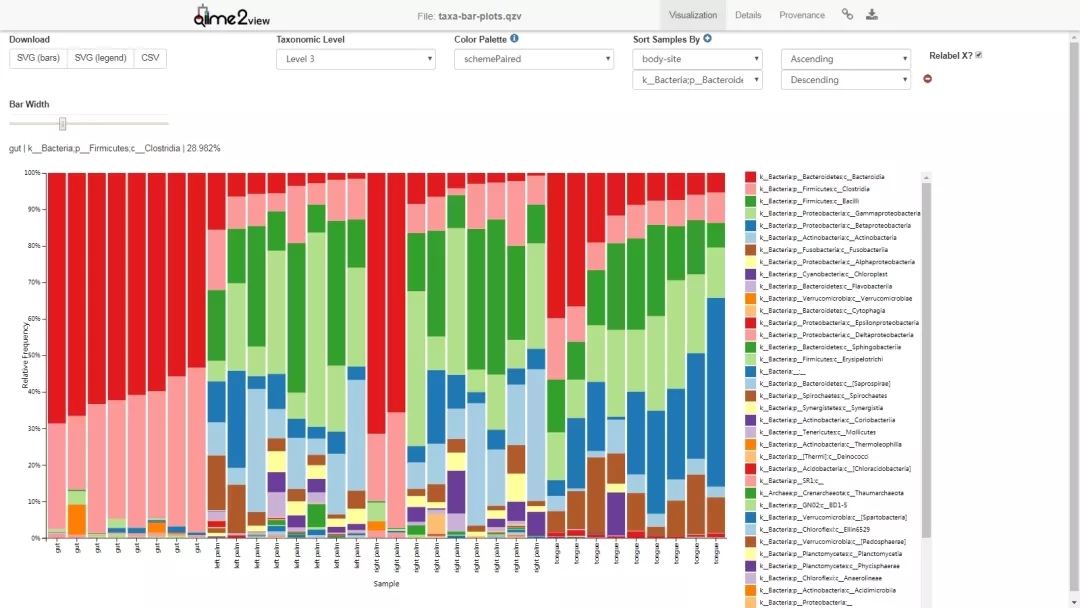
群落分类组成和物种丰度分布图
(通过 https://view.qiime2.org/ 展示)
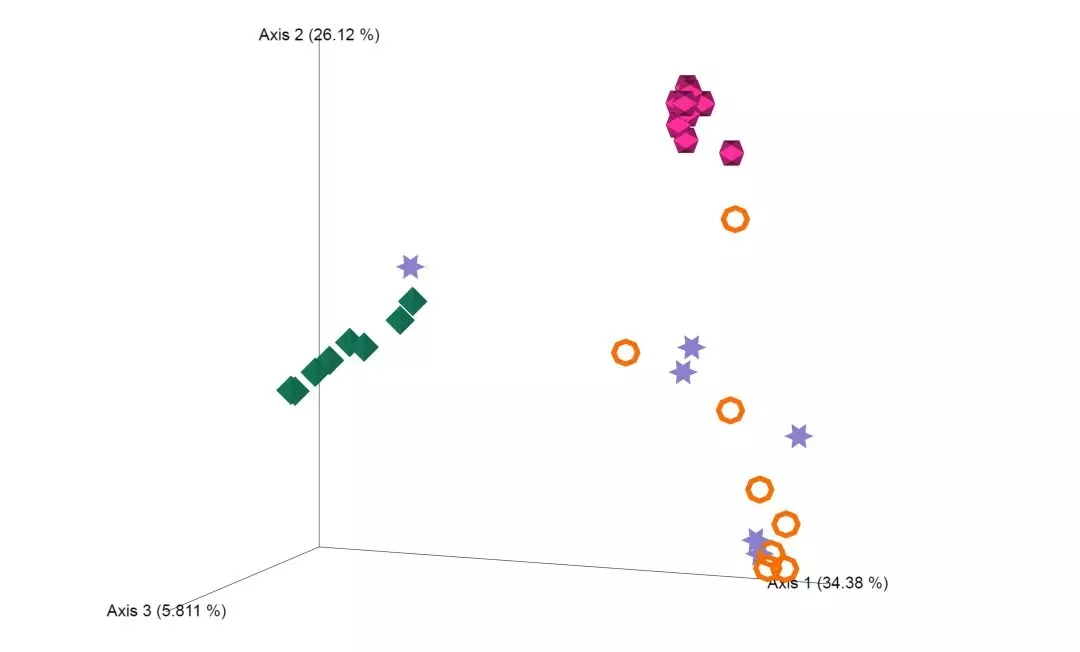
Beta 多样性分析
(通过 https://view.qiime2.org/ 展示)
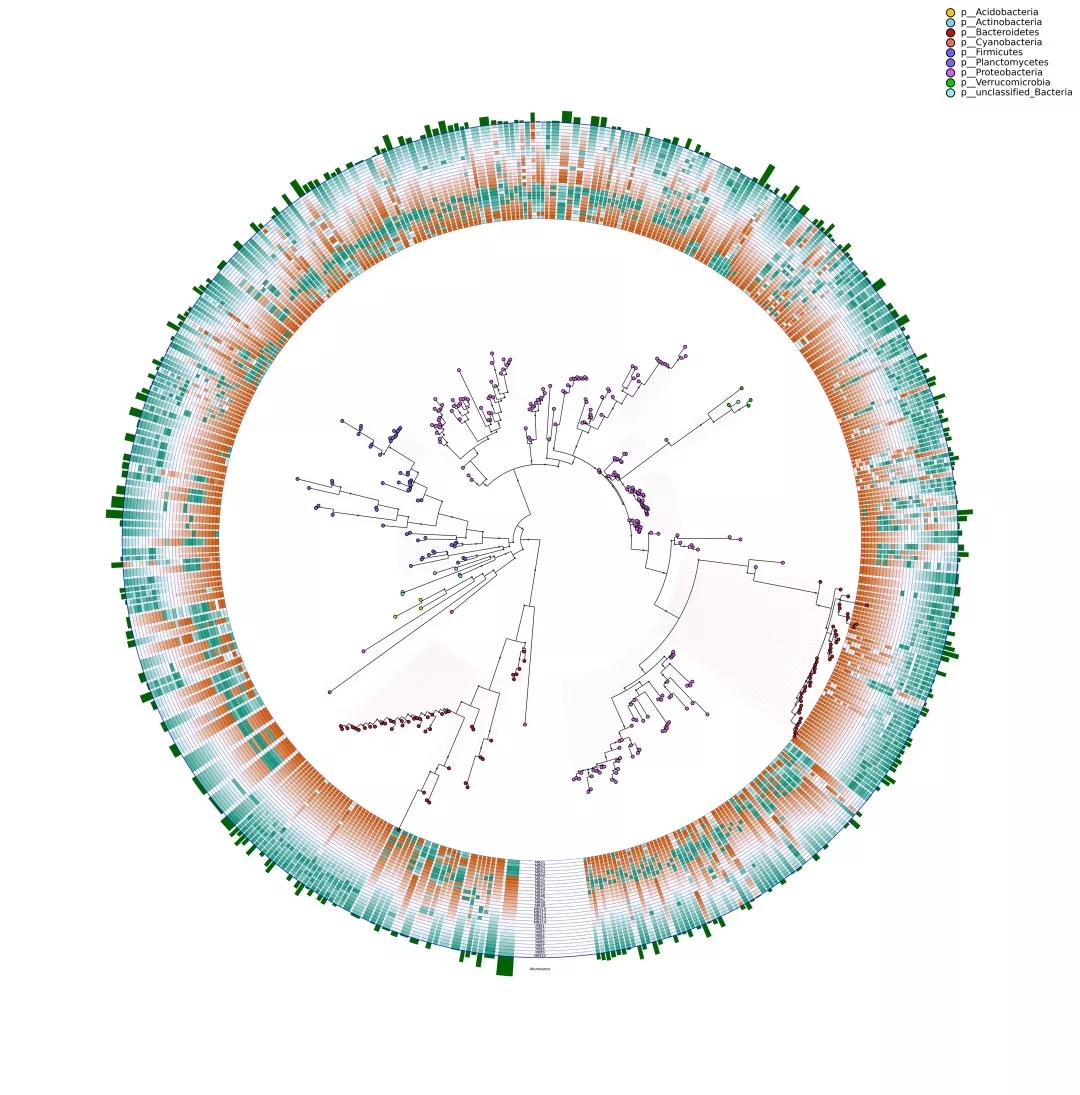
Graphlan 物种组成分析
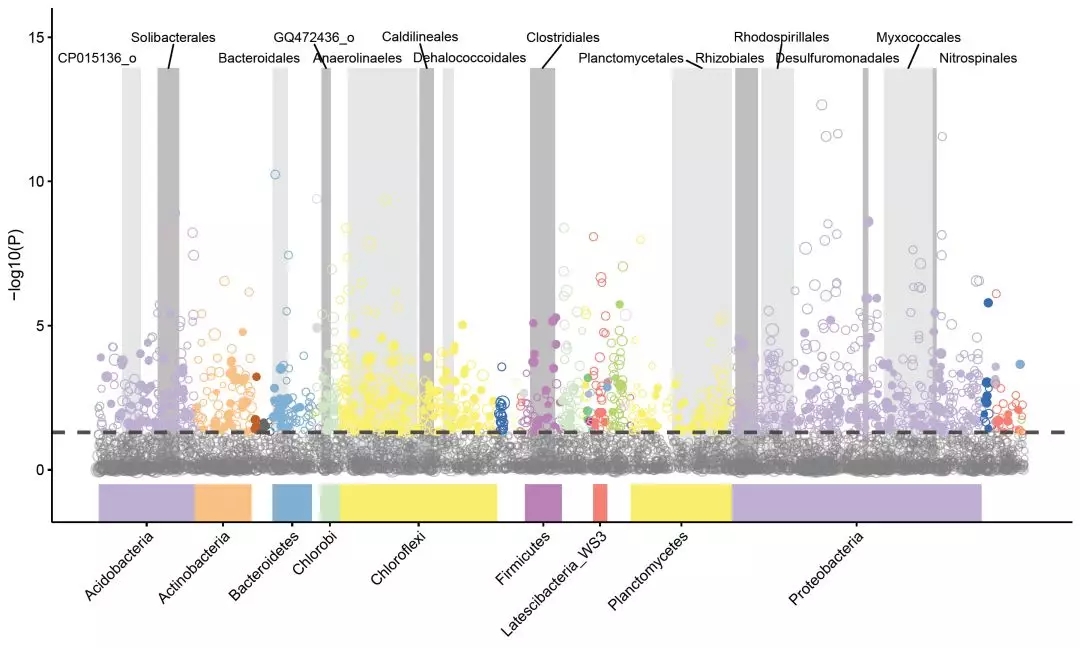
差异 ASV/OTU 的曼哈顿图
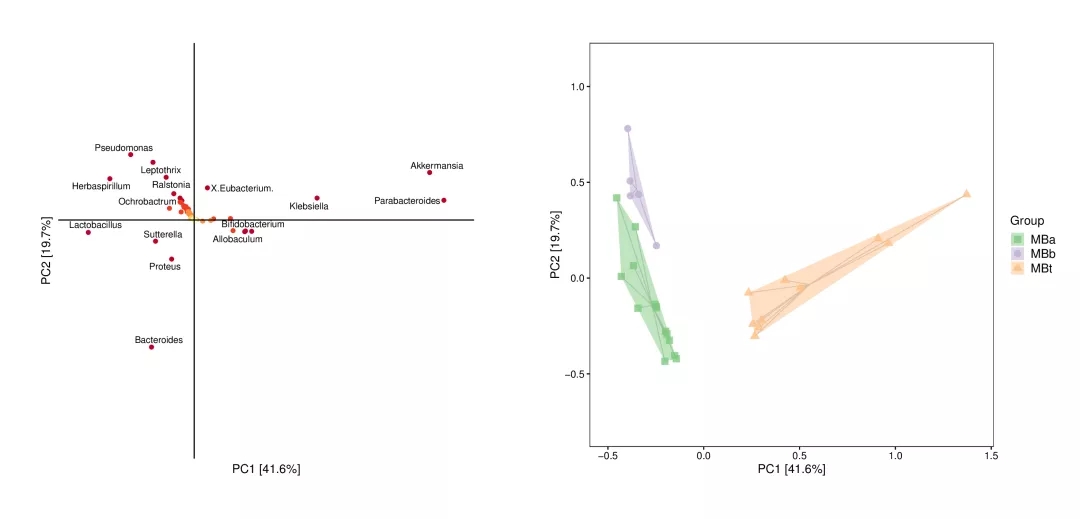
OPLS-DA 判别分析
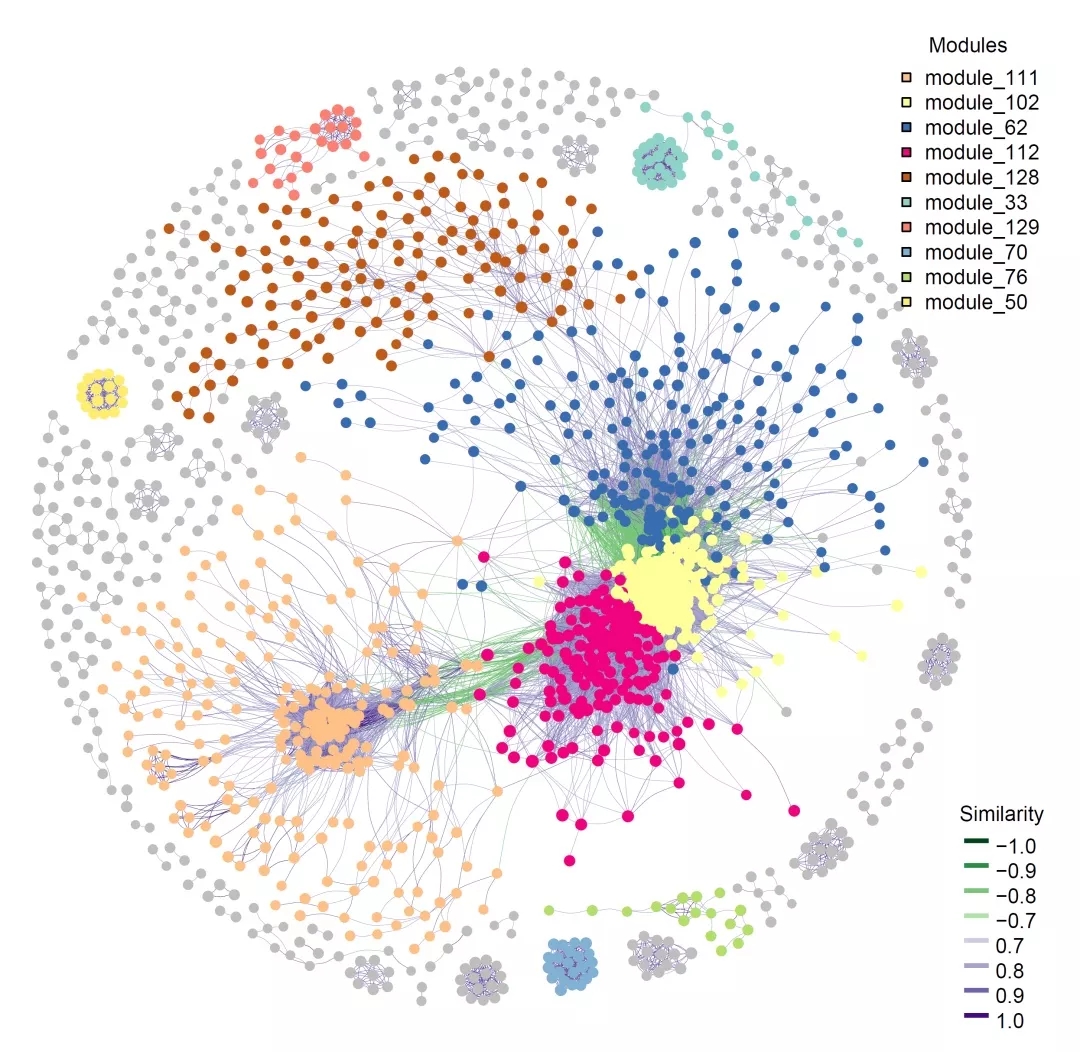
模块化物种组成关联网络图


