2020-02-02
新型冠状病毒感染的肺炎疫情自2019年12月在武汉爆发以来,科学界对这一病毒正进行争分夺秒的研究。我国此前已公布了新型冠状病毒的全基因组序列,由复旦大学生物医学研究院张永振教授领导的协作团队完成。
新型冠状病毒基因组序列有什么作用呢?
1、借助冠状病毒序列可进行溯源研究,研究宿主与病毒的关系
2020年1月23日,中国科学院武汉病毒研究所石正丽团队在bioRxiv预印版平台上发表文章Discovery of a novel coronavirus associated with the recent pneumonia outbreak in humans and its potential bat origin,报道了武汉新型冠状病毒nCoV-2019。在早期开始的时候,从5名病人体内获得了该病毒的全基因组,其中这5例基因组基本上一致,并于SARS-CoV的序列一致性有79.5%。重要的是,nCoV-2019与一种蝙蝠中的冠状病毒的序列一致性高达96%。
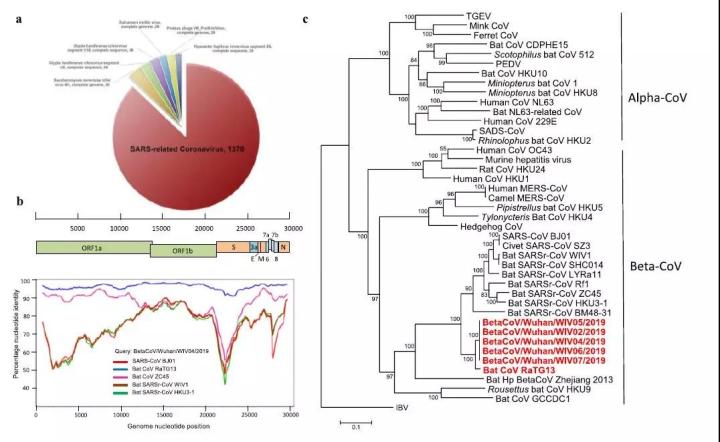
目前证据显示,SARS-CoV和MERS-CoV这两种病毒分别通过果子狸和骆驼传染给了人类。在感染人之前,病毒可能都源自蝙蝠,并分别在果子狸和骆驼群体内广泛传播、变异,最终形成了可以感染人类、在人与人之间传播的病毒。
虽然研究者在华南海鲜市场的环境样本中分离出了新冠病毒,但是,也有科学家强调,我们不能忽略第一批确诊的41个病例中,接近1/3的人与海鲜市场没有任何接触。
这次的疾病可能与SARS和MERS类似,也许病毒都源自蝙蝠,但也都不是蝙蝠直接将病毒传播给了人类,而是通过感染某种中间宿主(比如SARS-Cov通过果子狸,MERS-Cov通过骆驼),最终感染了人类。
2、冠状病毒序列有助于进行流行病学研究
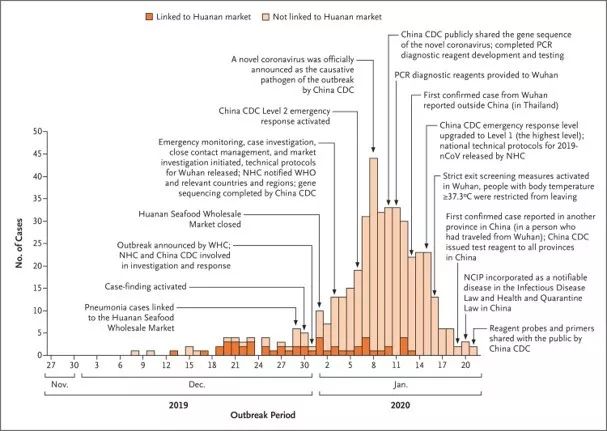
1月29日,来自中国多家多家疾控中心联合中国香港大学李嘉诚医学院公共卫生学院的联合署名作者在新英格兰杂志上发表了第一项包含425例2019-nCoV病毒感染相关肺炎的流行病学和传播动力学特征数据。
该报道收集了截至2020年1月22日已报告的实验室确诊NCIP(新型冠状病毒相关肺炎)病例的人口统计学特征,接触史和疾病时间表的信息。通过三种方法之一从患者的支气管肺泡灌洗液样本中鉴定出基因组:Sanger测序,Illumina测序或纳米孔测序。描述了病例的特征并估算了主要的流行病学时延分布。在指数增长的早期,我们估计了疫情的倍增时间和基本增殖速率。
结果在前425名确诊NCIP的患者中,中位年龄为59岁,男性为56%。到2020年1月1日之前发病的大多数病例(55%)与华南海产品批发市场有关,而随后的病例为8.6%。平均潜伏期为5.2天(95%置信区间[CI]为4.1至7.0),分布的第95个百分位数为12.5天。
在早期阶段,该流行病每7.4天增加一倍。平均连续间隔为7.5天(95%CI,5.3至19),基本增殖速率估计为2.2(95%CI,1.4至3.9)。
3、冠状病毒序列有助于进行诊断试剂盒、抗病毒药物的研发
对于一种新型病毒来说,拿到全基因组序列后,就相当于拿到了病毒的完整“大数据”,其鉴定依赖该病毒RNA序的高度保守序列,即使病毒再次发生变异,这些高度保守的序列在进化中基本保持不变,为试剂盒的研发奠定及疾病的快速诊断奠定了基础。
抗病毒药物的开发很大一部分是依托于病毒基因的,在病毒基因组上寻找药物突破的靶点。因此病毒的基因组研究对药物的开发和研究,都是有很大帮助的,可为医药企业提供完整的病毒数据,来确保抗病毒药物靶点的精准度。
作为一种新型冠状病毒,其基因组序列的破译有赖于高通量测序的支持,病毒的全基因组测序以及对应的生物信息学方法是研究病毒进化、毒力因子编译、疫病爆发之间的关系、疫病传播途径、不同遗传变异的分布模式,疫病发生地理区域的基础。
最后,每一场疫情之战,也是科学之战,是人类认识未知的艰辛跋涉。与病毒较量,拼的是知识、智慧和勇气,我们有信心,也有能力打赢这场疫情防控狙击战。
派森诺生物可提供病毒(DNA/RNA病毒)全基因组测序服务,基于高通量测序技术,获得病毒基因组的图谱信息,并在结构基因组学、比较基因组学层面通过差异分析、同源基因分析、共线性分析、物种进化分析等手段探究病毒的性质,为病毒的溯源和进化研究提供证据。
如果您想了解更多内容,欢迎在下方留言给我们,或发邮件到我们微生物基因组产品线邮箱:microsupport@personalbio.cn,我们期待您的咨询!
参考文献
1. 世界卫生组织,新型冠状病毒(2019-nCoV), https://www.who.int/zh/emergencies/diseases/novel-coronavirus-2019
2. Zhu N, Zhang D, Wang W, et al. A novel coronavirus from patients with pneumonia in China, 2019. N Engl J Med.
3. Xiang N, Havers F, Chen T, et al. Use of national pneumonia surveillance to describe influenza A(H7N9) virus epidemiology, China, 2004–2013. Emerg Infect Dis .
4. Munster VJ, Koopmans M, van Doremalen N, van Riel D, de Wit E. A novel coronavirus emerging in China — key questions for impact assessment. N Engl J Med.
5. Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus–Infected Pneumonia.
6. LI Q, GUAN X, WU P, et al. Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus–Infected Pneumonia[J]. New England Journal of Medicine.


