2020-03-03
之前的公众号文章给大家介绍了什么是翻译组学 转录组与蛋白组结果验证不上?——翻译组带你开启新世界的大门(点击查看),文献也有报道,从基因组生成整个蛋白质组需要有RNA生成调控(含表观遗传调控和转录调控)、RNA降解调控、蛋白质生成调控(即翻译调控)、蛋白质降解调控这四个主要调控阶段。其中,翻译调控占所有调控比例的一半以上,超过其他所有调控的总和,是细胞内最重要的调控方式。对于这么重要的调控方式,找到了开启研究大门的“钥匙”了吗?
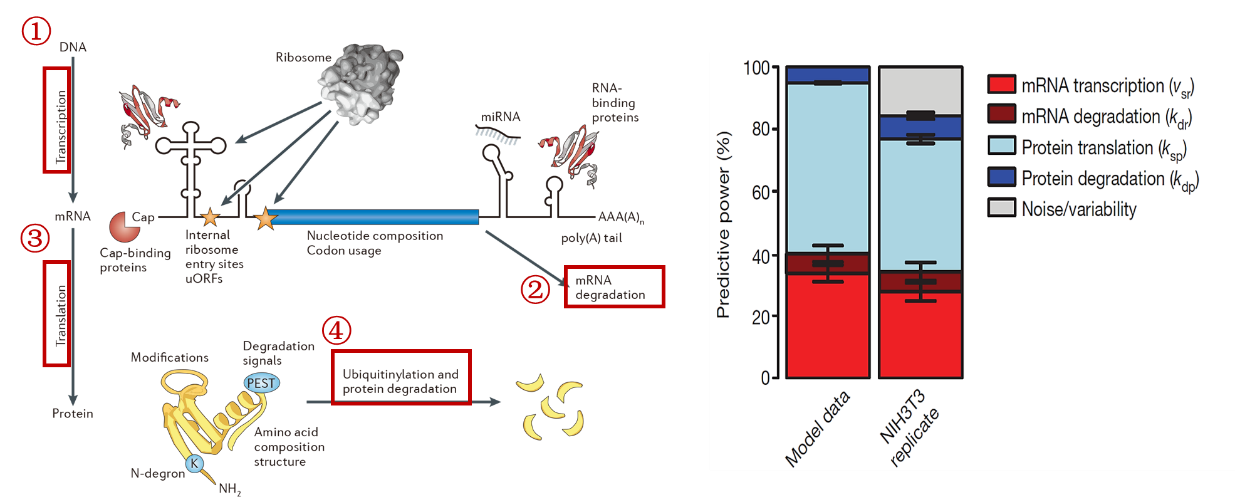
图1 翻译调控对蛋白质丰度影响占比
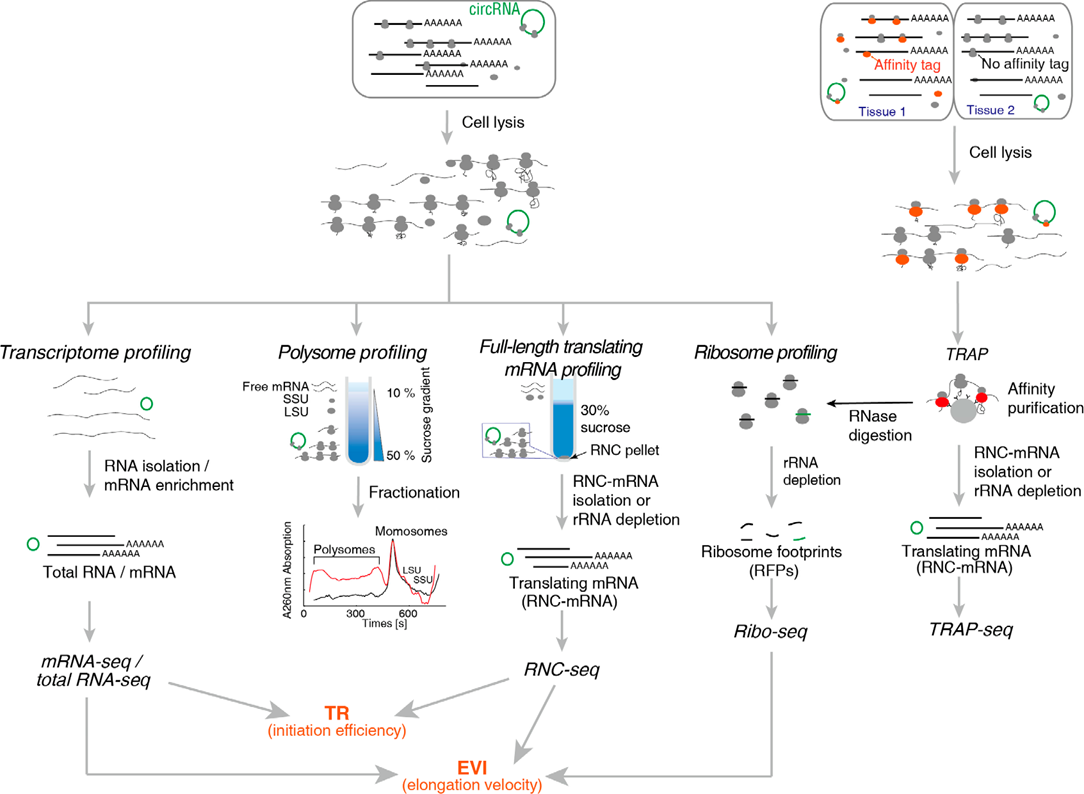
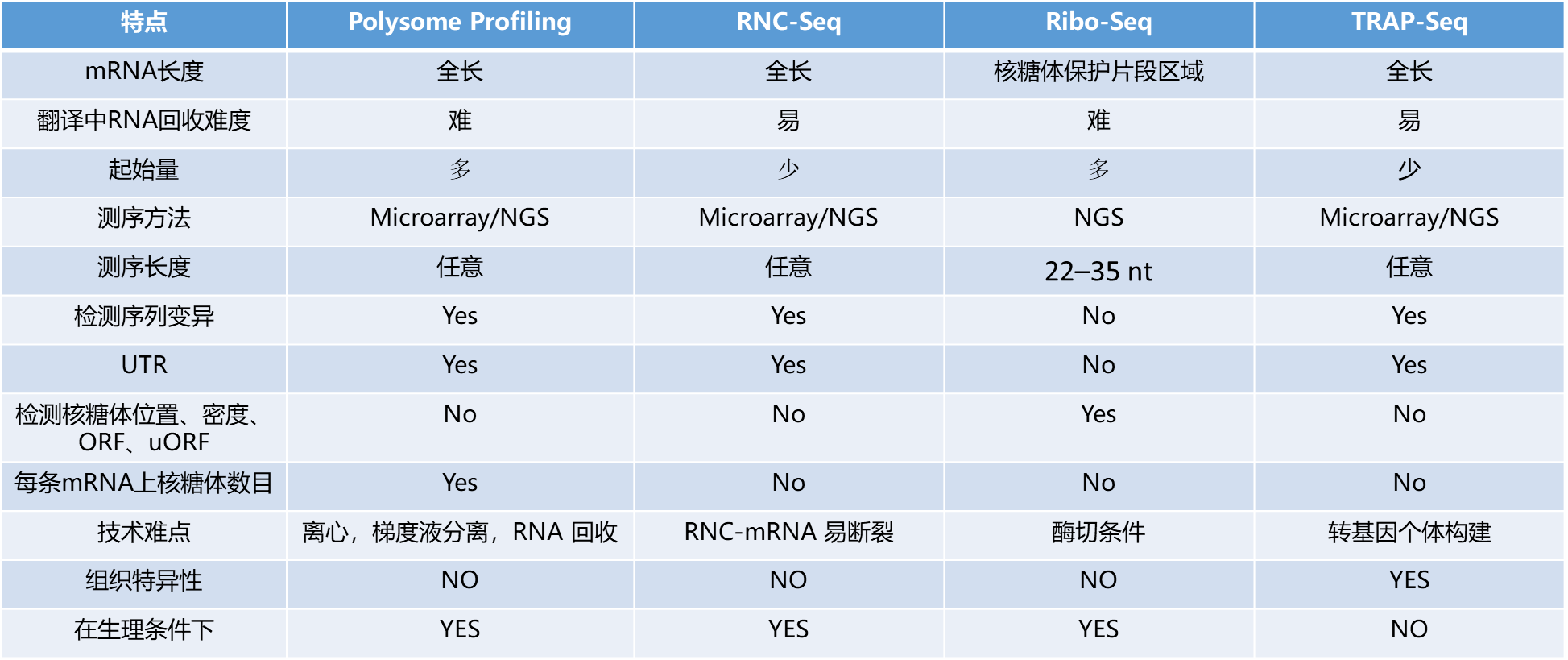
广义的翻译组(translatome)指直接参与翻译过程的所有元件,包括但不限于核糖体、正在翻译的mRNA(translating mRNA,又称RNC-mRNA)、tRNA、调控性RNA(如miRNA、lncRNA等)、新生肽链(nascent polypeptide chain)、各种翻译因子等;狭义的翻译组特指正在翻译的mRNA。由于参与翻译过程的元件包含RNA和蛋白质两大类,并由复杂而精细的大分子机器调控,兼具静态构成和动态变化的特性。针对不同的组分,翻译组学发展出了多种多样的研究方法。 1、Polysome profiling 多聚核糖体图谱技术 2、RNC-mRNA 翻译谱分析技术 3、Ribo-seq 核糖体足迹谱技术 4、TRAP-seq 核糖体亲和纯化技术 1、多聚核糖体复合物(polysome)是20世纪60年代在蔗糖梯度超离心法的基础上发展起来的。核糖体是大多数细胞中最大的高密度大分子结构。在蔗糖梯度下,由更多核糖体结合的mRNA分子沉积速度更快。polysome profling是利用核糖体沉降系数较大的特性,用蔗糖密度梯度离心的方法分离多聚核糖体。一条mRNA上结合的核糖体数量越多,在梯度离心时的沉降速率就会越快,因此结合有不同数量核糖体的mRNA通过离心就可以在溶液中分开。然后可以对分离出来的各组分中的mRNA进行分析。在环境胁迫时,翻译的全局状态常有非常明显的改变,如在高渗压力下单个核糖体的组分明显增加,而在氧化压力下单个mRNA上结合的核糖体数量呈明显增加。 2、RNC-Seq是在Polysome profling方法的基础上,在2013年暨南大学张弓教授课题组采用RNC-mRNA(RNC表示Ribosome-Nascent-chain-Complex, 核糖体-新生肽链复合物)直接分离技术,使用单一浓度蔗糖溶液作为缓冲液,通过超速冷冻离心将核糖体组分与游离mRNA以及其他细胞组份分离开来。由于所有的核糖体组分都沉淀在管底,抽提沉淀中的RNA,即可得到RNC-mRNA。然后结合高通量测序即可获得所有正在翻译的全长mRNA信息。 3、20 世纪60 年代人们就注意到蛋白质合成过程中核糖体保护的mRNA 片段可以免受RNA 酶的降解作用。2009 年Weissman 课题组首次将高通量测序技术应用于研究酿酒酵母核糖体保护的RNA 片段(Ribosome protected fragments,RPFs),即核糖体图谱技术。这是目前较为常用的检测正在翻译的mRNA的技术。首先使用低浓度RNase处理核糖体-新生肽链复合物,降解掉没有核糖体覆盖的mRNA片段,最后对获得的被核糖体保护的约22~30 bp的RNA小片段(称为Ribosome FootPrints, RFPs;或者称Ribosome Protected Fragments, RPFs)进行测序分析。 4、TRAP 是由Gerber 课题组于2002 年发表,最初是在核糖体大亚基的L25(RPL25p)蛋白质的C 端连接上亲和标签(如His、FLAG 和eGFP 等),后来又采用RPL16a 进行C 端标记,这些带有标签的核糖体蛋白基因可以被组织特异性启动子启动;通过再转化获得稳定的转化细胞系、动植物个体,再通过抗体将含有标签的核糖体进行分离,进一步得到标签核糖体上结合的mRNA。 图2 四种翻译组研究技术原理流程图 表1 翻译组研究技术对比 翻译调控比转录调控更快更灵敏,使得生物在面对逆境时可以做出快速的应对措施以保证生物体的存活,极大地提高了基因表达调控的灵活性与复杂性,目前翻译组学已经广泛应用于肿瘤方面研究,微生物抗逆机制,指导蛋白质组鉴定以及新蛋白的发掘等等。2020年了,请大家重点关注起翻译组。 参考文献: 1、Zhao, J. , Qin, B. , Nikolay, R. , Cmt, S. , & Zhang, G. . (0). Translatomics: the global view of translation.Int J Mol Sci.. 2、Vogel, C. , & Marcotte, E. M. . (2012). Insights into the regulation of protein abundance from proteomic and transcriptomic analyses. Nature Reviews Genetics, 13, 679. 3、Schwanhäusser B, Busse D, Li N, et al. Global quantification of mammalian gene expression control.Nature. 2011;473(7347):337–342.