2020-03-19
通过小派前两次对于翻译组的介绍,想必大家已经深刻了解了翻译组研究的必要性及其研究技术(请大家及时复习哦~转录组与蛋白组结果验证不上?——翻译组带你开启新世界的大门和翻译组4大研究技术该选一种?(点击查看))。简而言之,就是使用Ribosome profiling (Ribo-seq)技术了解翻译水平的基因表达丰度,简称翻译丰度。但是,除了这个,翻译组你还能给大家带来点什么新鲜事吗?


小派今天带领大家深入了解利用翻译组Ribo-seq技术揭示uORF在发育过程中的调控作用。怎么样,这个知识点听过没,想不想学,上硬菜咯~
发表期刊:PLOS Biology
影响因子:8.386
文献题目:Genome-wide maps of ribosomal occupancy provide insights into adaptive evolution and regulatory roles of uORFs during Drosophila development
真核细胞的mRNA由5’非翻译区(5’UTR)、编码蛋白的开放阅读框区(ORF)及3’非翻译区(3’UTR)构成。研究发现,5’UTR存在一些具有翻译能力的开放阅读框,被称为上游开放阅读框(uORF)。与之对应,5’UTR之后的生物信息学分析表明,uORF在动植物中广泛存在,人、小鼠、拟南芥、水稻、玉米中超过30%的mRNA含有预测的uORF。开放阅读框被称为主开放阅读框(pORF)。uORF通常能抑制下游pORF的翻译。不少人类疾病都与DNA突变引入新的uORF或者破坏已有的uORF有关。尽管大多数引入新uORF的突变都是有害的而且会在自然选择作用下从基因组中清除掉,我们仍不清楚基因组中为什么还是有很多比预期更保守的uORF。Ribo-seq能帮我们“短,平,快”地找到每个基因mRNA是否存在uORF,并且“真善美”的对uORF进行精准定位和保证其确确实实发生了翻译。
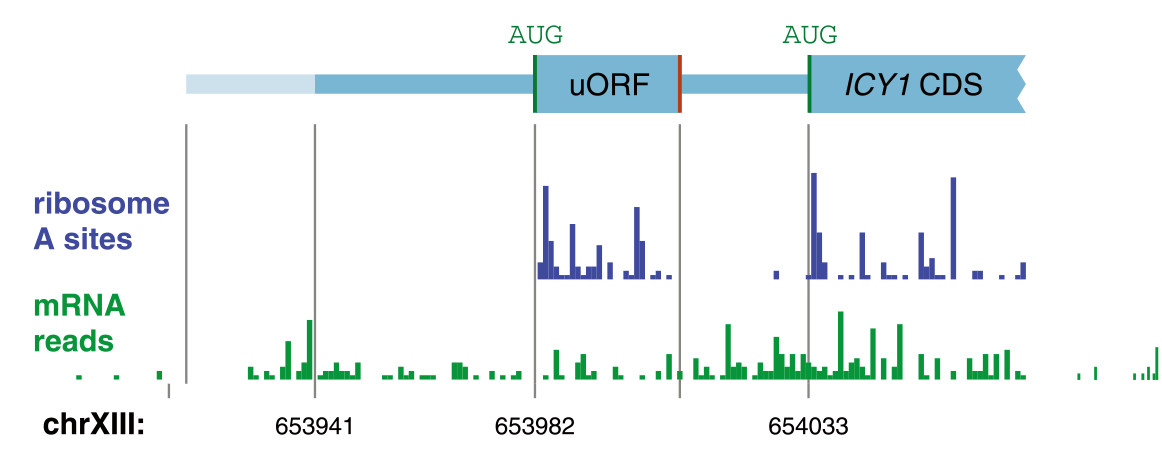
示例:ICY1基因的Ribo-seq reads图
Step1:对不同发育时期的果蝇进行Ribo-seq测序和转录组测序,获得其全基因组范围内的核糖体印记图谱。
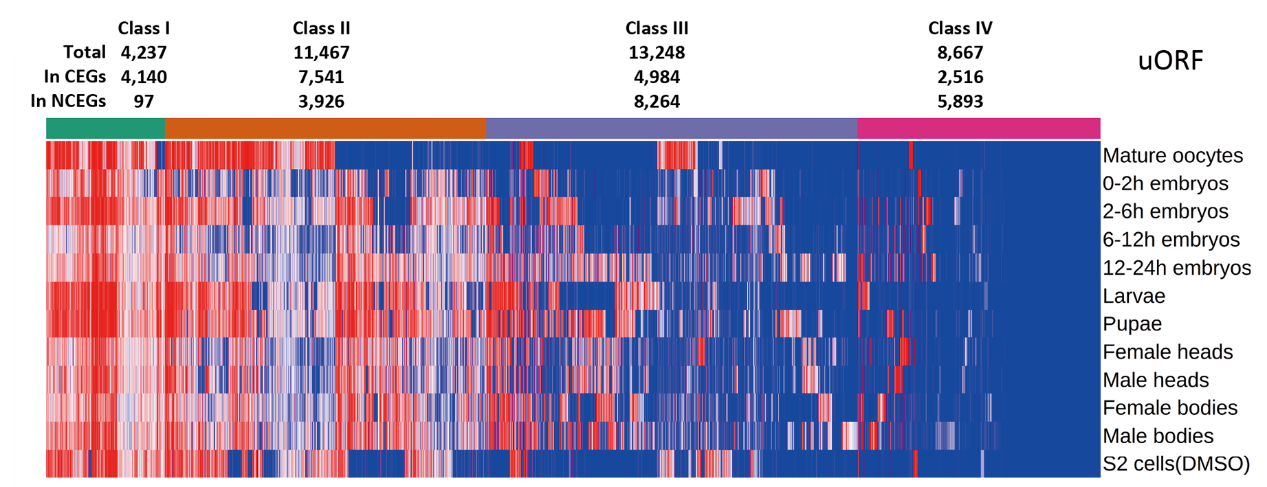
图1 果蝇基因组中全部uORF在不同发育时期的翻译效率图谱
Step2:通过分别计算uORF和下游CDs的ORF的翻译效率(TE),发现uORF整体上转译效率低于下游CDs的ORF的转译效率。
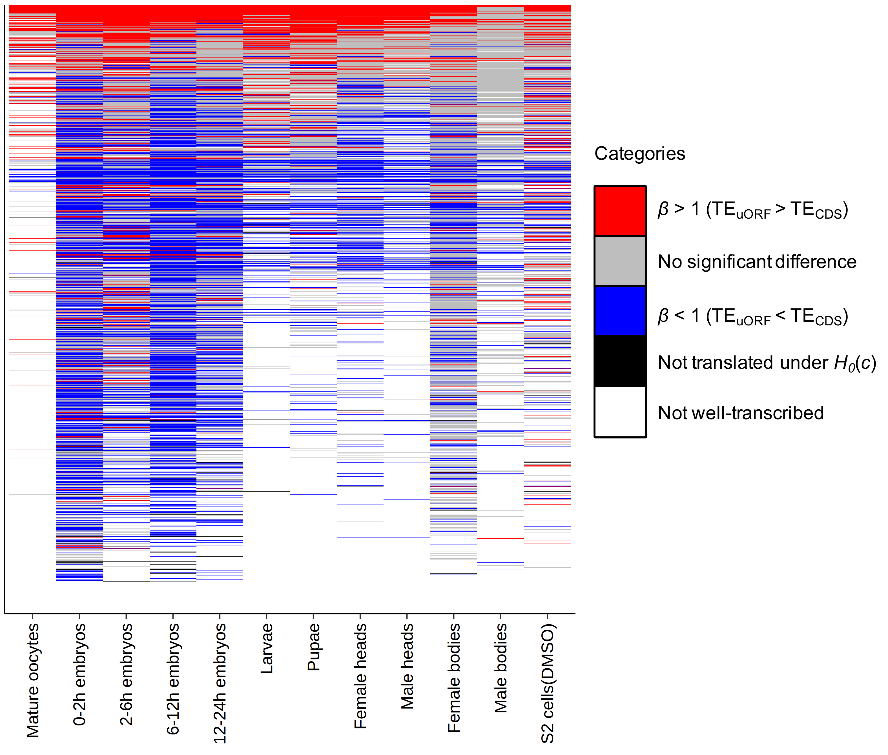
图2 uORF和下游CDs的ORF整体转译效率
Step3: 基因组序列特征(Kozak分数)影响uORF的转译效率,Kozak分数越高,uORF转译效率越高。
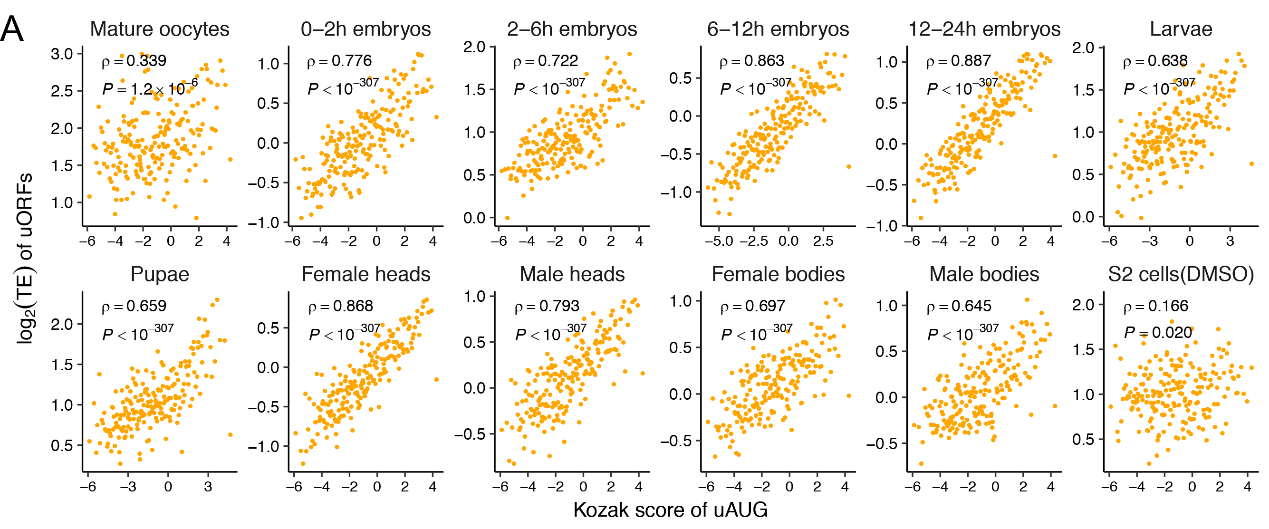
图3 Kozak分数与uORF转译效率相关性散点图
Step4: 5’非翻译区uORF的数目影响uORF对CDs的抑制强弱

图4 非翻译区uORF的数目与下游CDs区转移效率相关性统计图
Step5:5’非翻译区其他特征对CDs区转译效率的影响
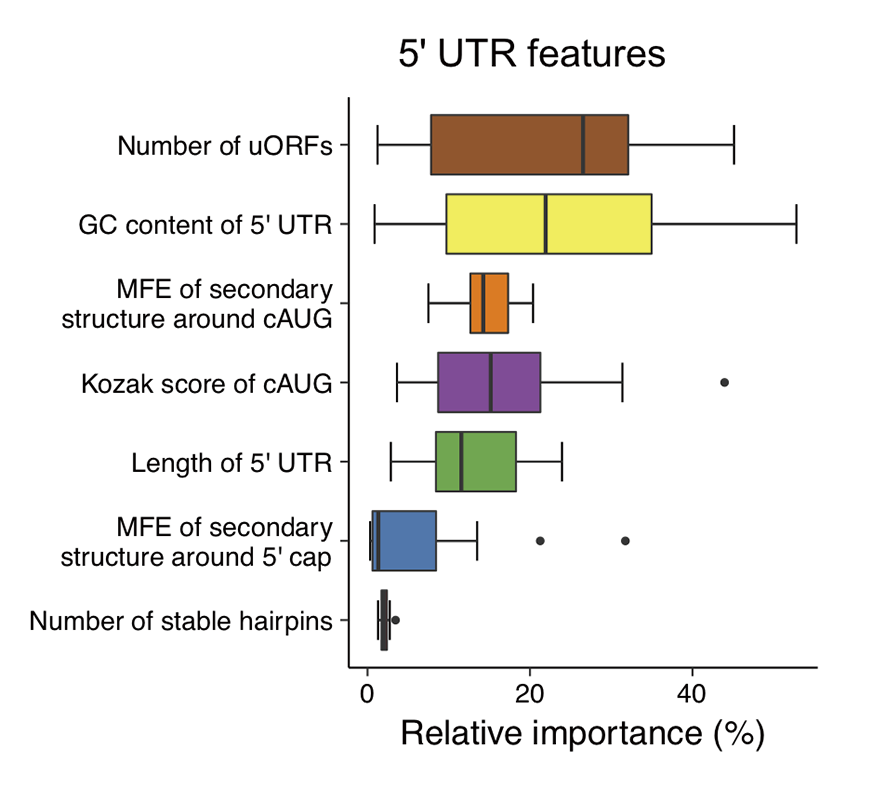
图5 5’UTR区对下游CDs区转译效率的影响重要性占比
今天的新知识点是不是有点多,理解起来有点困难,由于篇幅有限,如果想要深入了解本研究的思路和方法,欢迎大家有去查阅原文哦,当然,如果大家有任何问题,也可以在本公众号下咨询小派。
除了翻译丰度,除了uORF,翻译组,你还有什么?卖个关子,下期再告诉你。

参考文献:
1.Hong, Z. , Shengqian, D. , Feng, H. , Junjie, L. , Liping, W. , & Jian, L. , et al. (2018). Genome-wide maps of ribosomal occupancy provide insights into adaptive evolution and regulatory roles of uorfs during drosophila development. PLOS Biology, 16(7), e2003903-.
2.Ingolia, N. T. , Ghaemmaghami, S. , Newman, J. R. S. , & Weissman, J. S. . (2009). Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling.Science, 324(5924), 218-223


