
2020-03-31

这篇文章的研究背景、所使用的研究技术以及研究结论请看:干货 | Nature文章是怎么利用高通量测序做表观遗传的(一)。(点击查看)
重要提示
以下内容篇幅较长,详细介绍了整个研究过程与结论,推荐对ChIP-Seq以及DNA甲基化等表观遗传研究感兴趣的老师继续阅读。
PS:比大家直接阅读原文省力多了呢,英语大神除外(ಥ_ಥ)。如有与原文表述不符的地方欢迎大家留言指正!
1、为了描述组蛋白PTMs(翻译后修饰)在调节DNA甲基转移酶(DNMT)靶向和CpG甲基化中的作用,使用ChIP-seq在C3H10T1/2小鼠间充质干细胞(mMSCs)中分析了组蛋白PTMs (H3K36me3、H3K36me2、H3K27me3、H3K9me3、H3K4me1)和DNMT3A/B的全基因组分布模式。使用全基因组亚硫酸氢盐测序(WGBS)在分析CpG甲基化水平(测序深度45x)。结果发现H3K36me2和H3K36me3的富集区域非常接近,它们共同标定了Mb大小的区域,这些区域基本上不属于H3K9me3和H3K27me3所标定的区域。
H3K36me2水平与CpG甲基化呈正相关,与H3K27me3和H3K9me3一起,这些组蛋白PTMs分别将基因组分成CpG甲基化水平高(75%)、中(50%)和低(30%)的区域。此外,DNMT3A/B ChIP-seq reads主要出现在H3K36me2/3结构域中,这表明这些结构域中的CpG甲基化水平较高,至少部分原因是因为DNMT3A/B。与H3K36/CpGmehigh结构域相关的其他染色质特征,包括H3K27ac、H3K4me1水平的升高和基因表达的增加与先前将H3K36me2/3与活性基因转录联系起来的报道一致。此外,H3K36/CpGmehig结构域与小鼠成肌细胞的Hi-C研究中发现的区隔和拓扑相关结构域(TADs)相对应,这表明这些修饰共同定义了转录活性常染色质,它在空间上与构成性和兼性异染色质分离。观察放入结果与最近有关H3K36me3介导靶向DNMT3B活性的报道一致。然而,由于这种相互作用仅限于基因体,假设,一个额外的染色质反式调节通路可能会平行作用以促进常染色质区域CpG甲基化。在进一步的检测中,发现H3K36me3在基因体中表现出特征性的富集,而H3K36me2则表现出更为弥散的分布,包括基因区和基因间区。在活跃转录的基因中,H3K36me2通过第一个内含子覆盖了TSS下游的区域,然后在第一个剪接连接后切换到H3K36me3。值得注意的是,当DNMT3B在含有H3K36me3的基因体中富集时, DNMT3A的定位与H3K36me2的相仿,并跨越了较宽的基因间区域,而H3K36me3的水平并不明显。通过比较全基因组H3K36me2和H3K36me3的存在位置,可以校正de novo DNMTs的靶向谱。与H3K36me3相比,DNMT3A在H3K36me2高水平的基因组区域选择性富集DNMT3B。
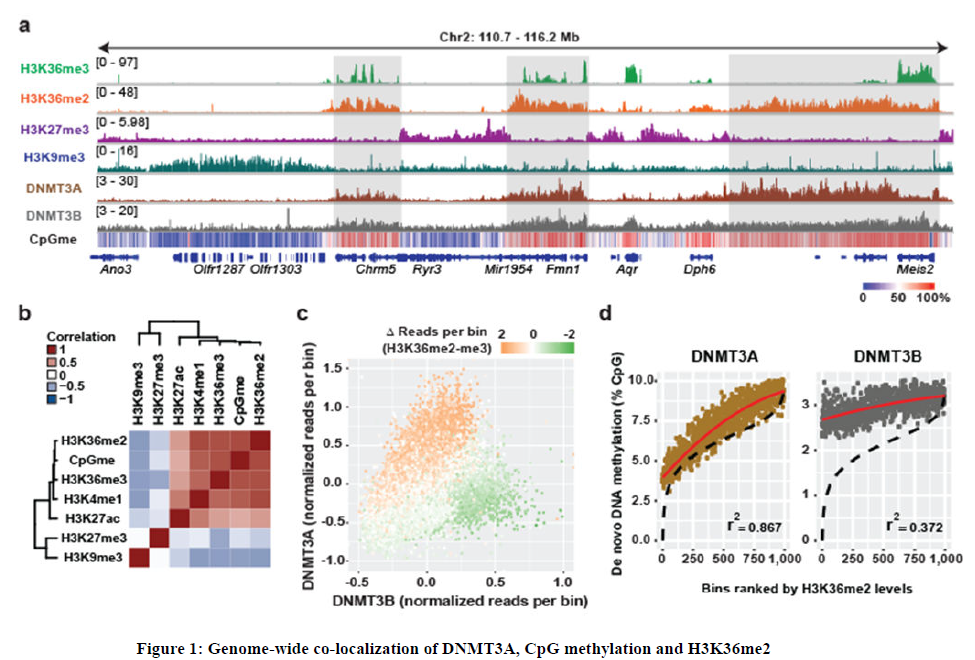
2、为了在另一个背景下证实上述结果,研究确定了H3K36me2是否在建立小鼠胚胎干细胞(mESCs)基因组甲基化过程中发挥了类似的重要作用。在亲本和DNMT3A缺陷(sgDnmt3a)的mESCs中稳定表达了主要DNMT3A亚型DNMT3A2 (HA)标记的血凝素。当表达接近生理水平时,当HA标记的DNMT3A的ChIP-seq显示其在亲代细胞和sgDnmt3a细胞间的全基因组定位模式相似。与H3K36me2/3的ChIP-seq谱相比,H3K36me2-DNMT3A与H3K36me3-DNMT3B之间的选择性共富集是明显的,但总体上,DNMT3A与DNMT3B在mESCs中的分布模式呈正相关。重要的是,DNMT3A的de novo甲基化活性是通过重新引入DNMT3A2到Dnmt1和Dnmt3a /b三者共敲除mESCs后的CpG甲基化水平来测量的,这些mESCs几乎不存在CpG甲基化,追踪H3K36me2的水平。这与Baubec等人观察到的DNMT3B的de novo活性与H3K36me3相关形成了对比,这表明DNMT3A的基因间靶向和DNMT3B的基因体靶向共同促进了在常染色质中CpG甲基化的建立。
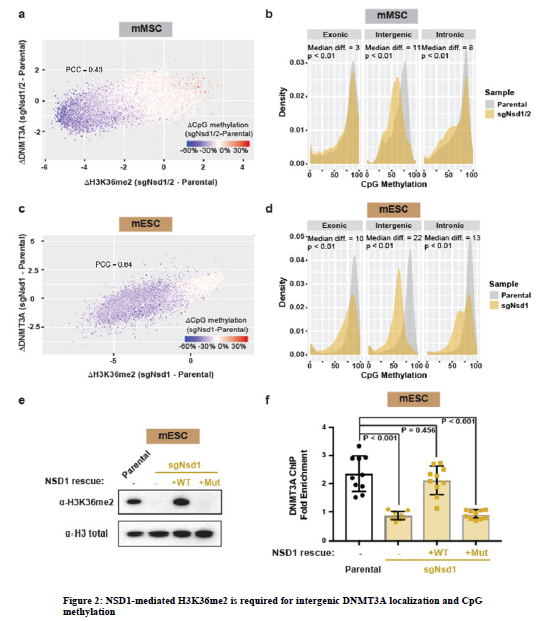
3、NSD家族酶已被证明可以在基因间区催化H3K36me2。在mESCs中,Nsd1是NSD的主要表达酶,而Nsd1和Nsd2均在mMSCs中表达。因此,研究使用CRISPR/Cas9对mMSCs中的Nsd1和Nsd2 (sgNsd1/2)或mESCs中单独对Nsd1 (sgNsd1)进行了基因基因切除。以Setd2被单独打断作为对照(sgSetd2)。sgNsd1/2和sgSetd2 mMSCs中H3K36me2和H3K36me3分别显著减少和特异性减少,表明这两种酶在染色质水平上的作用明显。利用添加了果蝇染色质的ChIP-seq作为外源性参考对照,被用来定量分析亲代细胞和H3K36me2 /3缺失细胞之间的全基因组变化。在sgNsd1/2 mMSCs和sgNsd1 mESCs中,H3K36me2全面降低,主要在基因间区。相比之下,H3K36me3水平未受干扰,基因H3K36me2的减少相对温和,这可能是由于NSD3或ASH1L等其他H3K36甲基转移酶的活性。
4、接下来,分析了HA标记的DNMT3A1(用于mMSCs)和DNMT3A2(用于mESCs)的全基因组结合模式,以及H3K36me2缺失细胞中的DNA甲基化。通过删除mESCs和mMSCs中的NSD家族酶消除了DNMT3A在基因间区域的招募。全基因组分析显示,靶向H3K36me2缺失区域的DNMT3A缺失与CpG甲基化减少相一致(图2a,c)。根据H3K36me2和CpG甲基化下降之间的共定位和H3K36me2基因间不平衡富集的预测,sgNsd1/2 mMSCs和sgNsd1 mESCs中的CpG低甲基化主要影响基因间区域(图2b,d)。重新引入野生型NSD1,但不是催化突变型(C2023A),能够恢复全面的和基因间的H3K36me2水平。重要的是,DNMT3A的基因间定位只能通过外接野生型NSD1来恢复,这表明NSD1催化的H3K36me2在将DNMT3A招募到非编码的常染色体区域中有特定的需求。因此,得出结论,NSD1在引导DNMT3A进入基因间区和维持CpG甲基化方面起着重要和特殊的作用。
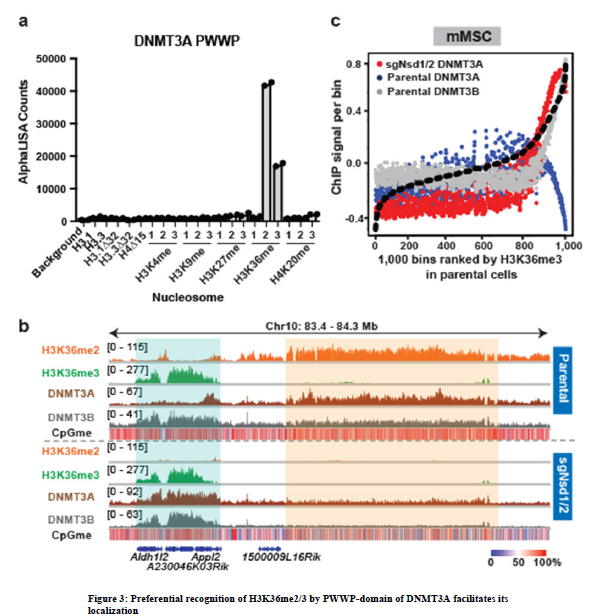
5、接下来,试图阐明H3K36me2介导的DNMT3A募集特异性背后的机制。含有PWWP‘reader’结构域的DNMT3A和DNMT3B都含有可以在体外与H3K36甲基化相互作用。为了测试DNMT3APWWP对不同价的H3K36甲基化的特异性,检测了纯化的DNMT3APWWP结构域和一组半合成核小体之间的相互作用。DNMT3APWWP与H3K36me2修饰的核小体的亲和力最高,其次是H3K36me3,但不与H3K4、H3K9、H3K27或H4K20的任何价态结合。定量等温滴定量热法(ITC)测定H3.1或H3.3 K36修饰肽段和DNMT3APWWP进一步支持其优先识别H3K36me2/3。这些结果表明DNMT3APWWP可以识别这两种甲基化状态,但对H3K36me2的亲和力更强。
6、研究推测,结合H3K36me2的相对丰度,这种结合偏好有助于DNMT3A在基因间区域的富集。因此,验证了DNMT3A在H3K36me2耗尽时与H3K36me3共定位的假设。事实上,注意到在sgNsd1/2 mMSCs中DNMT3A显著地重新分配到基因体中。这不能通过其与剩余基因H3K36me2的相互作用来解释,因为DNMT3A现在显示出与DNMT3B结合的轮廓,其中信号被富集到基因体的3 '端。此外,在亲代mMSCs中,DNMT3A在高H3K36me3区域被耗尽,而在sgNsd1/2细胞中,DNMT3A在相同区域被富集。sgNsd1/2细胞(TKO)中SETD2催化活性的中断破坏了DNMT3A在基因体中的定位,反映了DNMT3A基因组重新靶向中对H3K36me3的需求。在PWWP区域引入一个点突变(D333A),该突变削弱了其与H3K36me2/3的结合,终止了DNMT3A在sgNsd1/2细胞中重新定位到H3K36me3富集区。综上所述,这些结果表明PWWP域优先识别H3K36me2/3引导DNMT3A跨细胞染色质定位。
7、想知道DNMT3B是否也会发生类似的再分配机制。从其定位模式可以看出,DNMT3B的PWWP结构域在体外优先与H3K36me3重组核小体结合。在H3K36me3缺失的sgSetd2 mMSCs中DNMT3B的基因体定位缺失伴随着相对温和的向H3K36me2富集的基因间区重定向的增加。总的来说,这些数据支持H3K36me2/3比值是确定DNMT3A和DNMT3B靶向模式的关键决定因素,在一定程度上也支持H3K36me2/3比值是确定DNMT3A和DNMT3B靶向模式的关键决定因素。
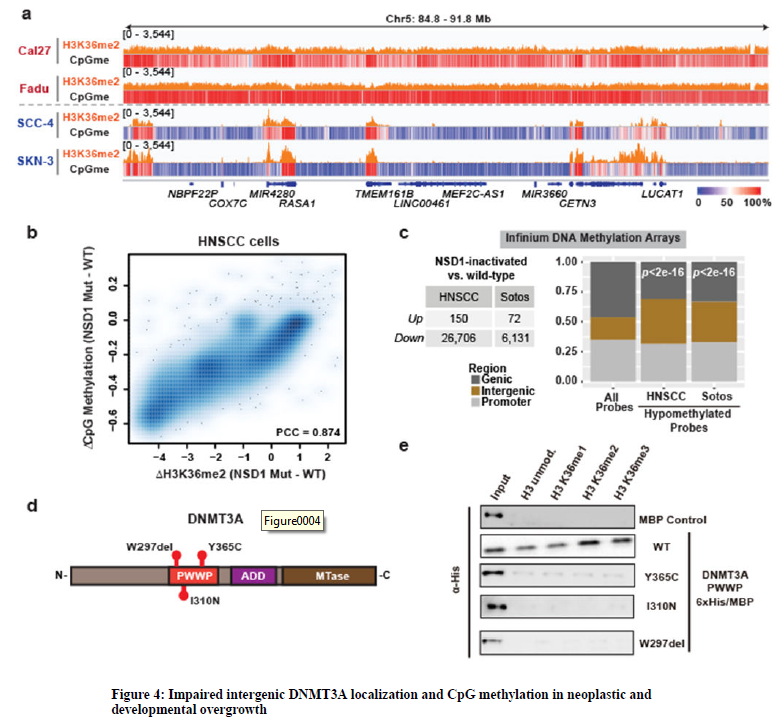
8、之前报道过,由于NSD1的基因或生化失活导致H3K36me2缺失的头颈部鳞状细胞癌(HNSCCs)有一个DNA低甲基化信号。同样,在Sotos患者的胚系NSD1单倍计量不足的血液样本中,DNA甲基化也显著降低。为了探索研究发现的疾病相关性,分析了携带野生型(Cal27和Fadu)或突变型(SCC-4和SKN-3)的患者来源的HNSCC细胞系中的H3K36me2和CpG甲基化。尽管有异质的遗传背景,与NSD1野生型细胞系相比,NSD1突变细胞系中的DNA低甲基化与H3K36me2的全基因组减少密切相关,且主要发生在基因间区域。进一步分析了公开的HNSCC或Sotos患者样本的DNA甲基化阵列。与NSD1野生型HNSCC肿瘤相比,NSD1灭活的低甲基化探针在基因间区显著富集。与对照组相比,索托斯综合征患者的基因间区也表现出低甲基化探针的富集。
9、DNMT3A的错义突变导致 TBRS,这是一种发育障碍,与Sotos有许多共同的临床特征,包括骨骼过度生长、面部畸形和智力残疾。在DNMT3A PWWP区域(W297del, I310N, Y365C)对TBRS的点突变进行了特征描述。携带这些突变的重组表达的DNMT3A PWWP结构域在体外表现出核小体结合减少,表明它们可能损害DNMT3A的染色质招募。事实上,W297del和I310N突变降低了DNMT3A与大量染色质的关联,最近报道的23个突变也伴随着蛋白水平的降低。进一步检测与染色质结合的野生型、W297del、I310N或Y365C突变DNMT3A,发现突变DNMT3A复合物中核小体上的H3K36me2水平显著降低。因此,ChIP-seq分析表明,I310N突变取消了DNMT3A对H3K36me2全基因组的靶向性。因此,DNMT3A募集障碍和H3K36me2富集的基因间区CpG甲基化减少是与TRBS和Sotos综合征相关的共同特征。
10、本研究发现为理解DNA甲基化在常染色体基因组中是如何建立和维持的提供了深刻见解。优先靶向DNMT3A和DNMT3B,在它们的PWWP染色质阅读框引导下,分别将CpG甲基化到H3K36me2富集的基因间区和H3K36me3富集的基因体。同时,H3K4me3在活性启动子上的存在破坏了DNMT3A/B的ADD结构域与组蛋白H3之间的相互作用,从而保护这些区域不受异位甲基化的影响。通过招募几个CXXC域蛋白,优先识别CPG密集的启动子,包括参与活性DNA去甲基化的TET家族酶和H3K36去甲基化酶KDM2B,可以进一步确保启动子上DNA甲基化的缺失。因此,认为CpG甲基化是通过DNMT3A/B的联合作用在广泛的常染色体区域建立的,而转录起始的局部位点是保留的。然而,该发现并不排除DNMT1活性也受H3K36甲基化调控的可能性。此外,与H3K36me2/3结合的DNMT3A/B的结构表征将是确定其PWWP域选择性识别的基础。
11、该研究表明,功能失调的NSD1-DNMT3A跨调节通路导致的基因间DNA低甲基化,代表了两个表型重叠的人类过度生长综合征之间的一种机制联系。这一途径也可能促进组织肿瘤过度生长。Nsd1或Dnmt3a的缺失在鳞状细胞癌小鼠模型中促进肿瘤的发展。
DNMT3A的体细胞突变在急性髓系白血病中很常见,恶性肿瘤中反复发生的将NSD1与NUP98结合导致NSD1异常定位的易位也被观察到。值得注意的是,包括EZH2和组蛋白H1在内的其他表观遗传调控因子的种系突变会导致类似于TBRS和Sotos的发育障碍。此外,DNMT3A的功能获得突变导致多梳抑制域的异位DNA高甲基化,最近在小鼠和人类中发现这种突变会导致生长延迟和微头盖癌。因此,未来的工作是有必要解析复杂的染色质调控网络控制的DNA甲基化图谱横跨广阔的常染色体区域在细胞分化和生长的控制机制。
参考文献:
Weinberg DN, Papillon-Cavanagh S, Chen H, et al. The histone mark H3K36me2 recruits DNMT3A and shapes the intergenic DNA methylation landscape. Nature. 2019;573(7773):281–286. doi:10.1038/s41586-019-1534-3.