
2020-06-19
长久以来,困扰着无数科研人员的难题之一就是样本总量不足。很多时候,有了好的想法,但样本的获得却受诸多条件的限制,比如样品珍贵、样品来源稀缺、组织大小本身受限、或者含量极少的微生物、细胞等等。对于此类样本,想进行基因组测序是非常困难的。
在保留传统测序文库构建优势的基础上,我们不断优化建库流程,力求用较少的样本获得较高的文库转化效率。以往常规全基因组PE建库一次投入的DNA总量需要500ng以上,随着实验方案的不断优化,现在我们能够做到以300ng起始建库。
但即便如此,仍然会有很多老师的样本无法满足建库要求。针对此种情况,我们进一步改进建库方案,目前派森诺已有两套完善有效的微量建库方案可以对最低5ng起始量的DNA进行常规PE文库构建。
本文就带大家了解下我们全新推出的两种微量建库方案:
一种是先通过全基因组扩增(whole genome amplification, WGA)无序列倾向性的富集基因组DNA,随后进行正常建库;另一种是对微量DNA直接建库测序。
1.1、微量扩增方案
全基因组扩增指的是对全基因组通过扩增的方法增加DNA的总量。目前有很多种可用于全基因组扩增的技术方法,总体有两类,一类是基于热循环PCR的扩增方式如LA- PCR(linker adaptor PCR)、T- PCR(Tagged PCR)、DOP- PCR(degenerate oligonucleotide- primed PCR,)等;另一类则是不依赖PCR扩增的方法如多重链置换扩增(Multiple Displacement Amplification, MDA)等。
第一类方法由于用到热循环PCR扩增,扩增过程中存在一定程度的偏好性,最终扩增结果对全基因组的覆盖度有限并造成部分区域相对增多,且这类方法常用到Taq DNA聚合酶,扩增长度很难超过3K、保真性有限。
多重置换扩增(MDA)是1998年由耶鲁大学Lizardi博士首次提出。这种恒温扩增的方法依赖于链置换扩增原理,利用噬菌体Phi29 DNA聚合酶,高度扩增DNA。该酶具有极强的模板结合能力,能克服很多DNA复杂结构如发卡环等,连续扩增100kb的DNA模板而不解离;同时这种酶具有极强的3’-5’外切酶活性,其保真度是Taq酶的1000倍。Phi29 DNA聚合酶强大的延伸活性和高保真度使得通过MDA可从极少量的DNA样本获取大量高质量的DNA(通常以5ng起始最终能得到5-20ug的DNA),MDA扩增产物可达到95%以上的覆盖度,扩增产物大小平均大于20 kb,可广泛适用于全基因组和外显子测序、大片段拷贝数变异分析、微卫星分析、qPCR分析、基因芯片分析,是目前为止对整个基因组覆盖最广、各位点扩增偏好最小的WGA方法。
MDA主要用到phi29 DNA聚合酶和硫代修饰的六碱基随机引物。在恒温条件下,随机引物在多个位点与模板退火,随后在phi29 DNA聚合酶作用下启动复制反应;沿模板合成的新链在延伸过程中从模板中置换出下游已复制的DNA链,被置换出的DNA链又作为新的模板,在phi29 DNA和随机引物的作用下继续合成新的DNA链直至引物和原料耗尽达到扩增反应的最大浓度(如图一所示)。最终得到的DNA可以直接用于后续分子实验。

图一 MDA反应示意图
1.2、全基因组DNA直接微量建库方案
MDA能得到高质量的可直接用于建库的DNA,但也有其局限性。第一,MDA对模板的灵敏度极高,提取到的DNA或者在MDA扩增中如果有任何轻微异源污染都会在后续扩增中成倍放大,所以其对整个实验阶段要求极高。第二,目前虽然有95%的覆盖率和高保真性,但和直接提取的DNA仍然有一定的区别。第三,MDA方案对DNA的完整性和纯度有极高的要求;如果完整度和纯度不足,扩增后会存在部分染色体错位或等位基因丢失的现象,这对提取的要求就会很高。通过优化MDA的实验方案能够在一定程度上改善这些问题,但直接将原始DNA用于后续建库测序实验显然是更有效的方案。
根据以往优化实验中积累的大量经验,我们开发了一套派森诺独有的直接以极低起始量DNA为模板进行建库的方案。经过大量实际上机测试,微量建库结果与高起始量的常规建库结果高度一致。
使用相同样品(水稻叶片)同时使用两种方案建库:300ng起始量和5ng起始量建库上机。我们分别从文库质量、下机数据质量、与参考基因组比对率几个方面进行比较两种建库方案。
1.2.1、文库质检
对文库分别用qubit(ng/ul)和qPCR(nM)两种方式定量,使用labchip对文库的大小检测(表一、图二、图三所示)。根据质检结果,两种方案最终文库质量基本一致,文库大小均集中在500bp左右。
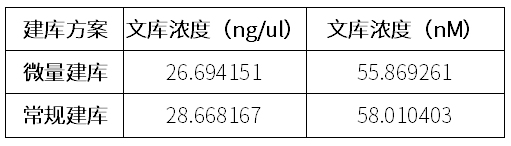
表一 文库浓度详表
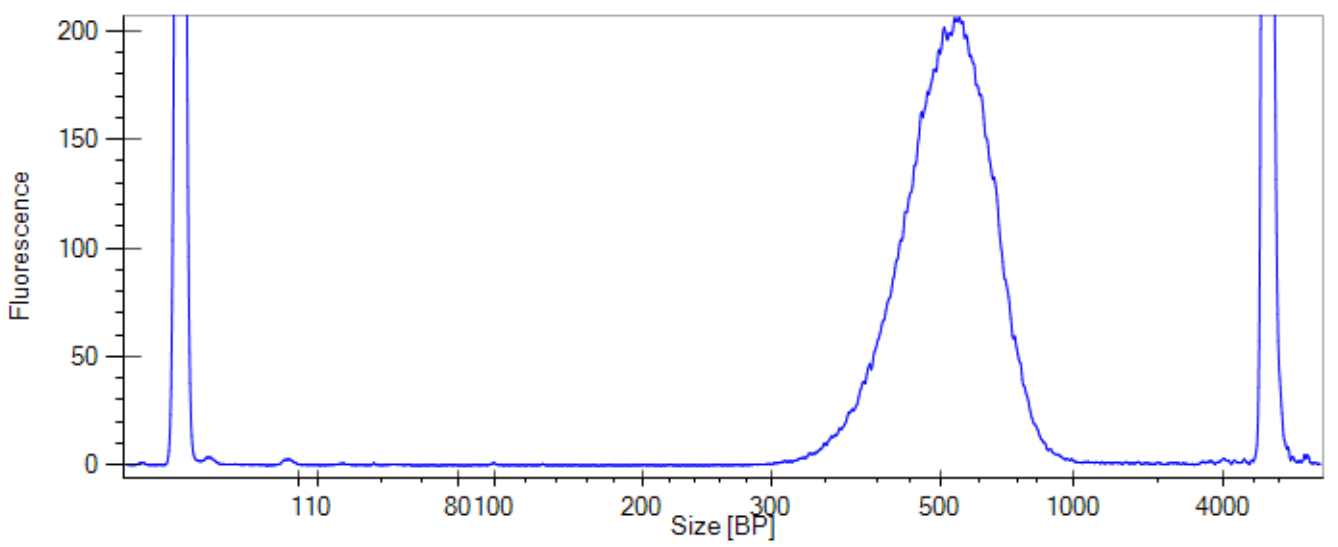
图二 微量文库labchip检测结果
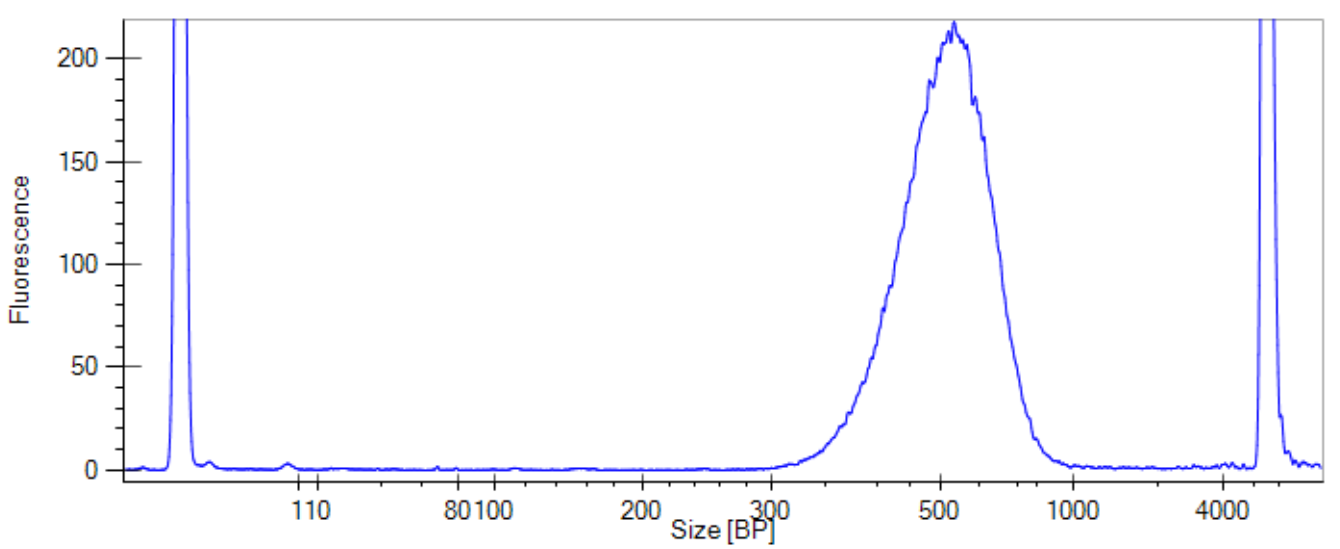
图三 常规文库labchip检测结果
1.2.2、下机数据质量比较
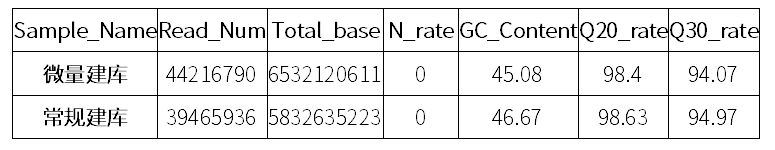
将两种文库同时测序,按照6G/样上机,上机模式为Novaseq PE150。分别对两个文库的下机数据进行统计,结果如表二所示。两个文库的下机数据量均在6G以上,N(%)比例、GC含量正常;Q30高,数据可信度高。
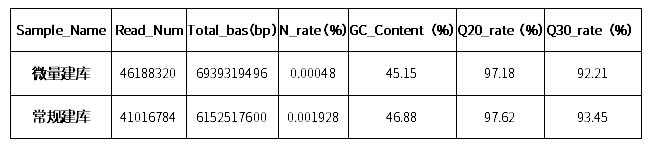
Sample:样品名;
Reads Num.:Reads 总数;
Total Bases(bp):碱基总数;
N(%):模糊碱基所占百分比;
GC(%):GC 含量;
Q20(%):碱基识别准确率在 99%以上的碱基所占百分比;
Q30(%):碱基识别准确率在 99.9%以上的碱基所占百分比。
表二 测序数据统计
1.2.3、高质量数据获取
将原始下机数据(raw data)过滤生成高质量序列(high quality data)。数据过滤的基本情况见表三。
Sample:样品名;
Reads Num.:Reads 总数;
Total Bases(bp):碱基总数;
N(%):模糊碱基所占百分比;
GC(%):GC 含量;
Q20(%):碱基识别准确率在 99%以上的碱基所占百分比;
Q30(%):碱基识别准确率在 99.9%以上的碱基所占百分比。
表三 测序数据过滤统计
1.2.4、序列比对
将过滤后得到的高质量数据比对到参考基因组上。序列比对结果统计见表四。
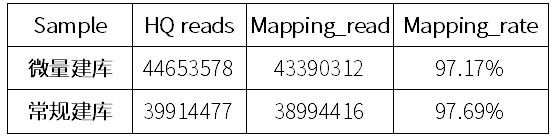
Sample:样本名;
HQ reads:过滤后的高质量 reads 数量;
Mapped reads:比对至参考基因组上的 reads 数量(包括单端比对和双端比对);
Mapping rate:比对率,比对至参考基因组上的 reads 数量占总 reads 数量的百分比。
表四 序列比对结果统计
根据下机数据结果对比显示,两种方案的文库质量、下机数据质量基本一致,过滤后的高质量数据在参考基因组上的比对率基本一致。除水稻等大型植物外,我们还对动物、微生物、环境等多种不同类型样本进行建库上机测试,微量方案和常规方案在文库质量、下机数据质量等方面都保持一致,在此就不一一赘述~
以上便是派森诺对于低起始量模板的两种建库方案。微量扩增方案对样品和操作环境要求高,需要最大可能避免外源样品的污染,这对取样、提取、建库环节都有着极高的要求。
全基因组DNA直接微量建库优势在于使用了提取后的原始DNA,能保证微量建库结果和正常高起始量建库测序结果一致。