2020-07-16
16S rRNA基因是细菌系统分类研究中最常用的分子标记,在进化上具有良好的时钟性质,其序列包含相互间隔的10个保守区和9个可变区,保守区物种间变化不大,而可变区具有属或种的特异性。利用高通量测序技术对16S rRNA基因可变区进行测序,能全面解析样本中的物种组成和对应的丰度信息,因此被广泛应用于微生物群落研究。
然而需要注意的是,当前基于高通量测序的微生物群落多样性研究,都是基于相对定量的方法,即把数据归一化到统一数据量,以每种菌reads数占总reads数的丰度比例来进行量化分析。这种定量方式存在的问题也是显而易见的,由于16S扩增子文库的构建有一个无法回避的过程,那就是PCR扩增。只要存在PCR扩增,就会有重复的产生,而扩增偏好性引发的重复的不均匀性,就是相对丰度不可靠的罪魁祸首。也许你会说,我们可以在生信分析端把重复都去掉,然而现实是这些重复的来源不同,代表的意义也不同,一刀切的去重同样也歪曲了事实,图一列举了微生物群落多样性研究的测序数据中重复的主要来源:
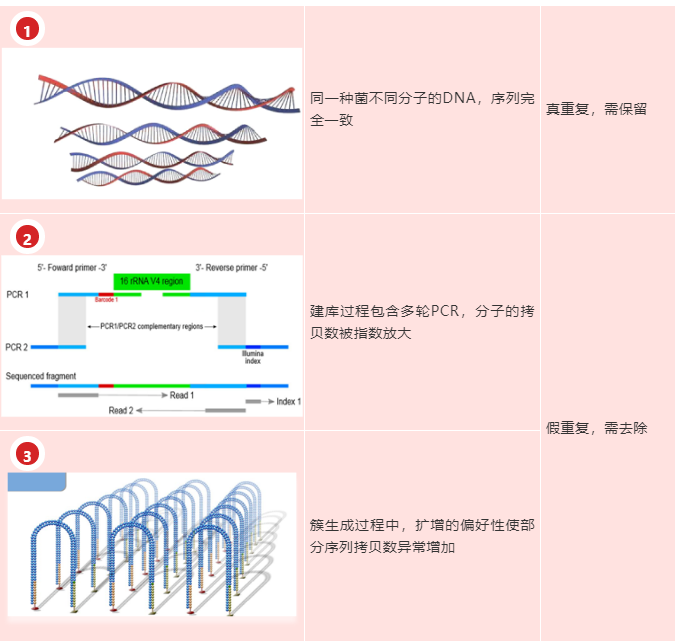
图一:测序数据中重复的主要来源
①中重复代表的是同种菌的不同分子,是真实物种丰度的呈现,这种重复需要被保留,而②和③这些重复是由扩增产生的假重复,他的数量不能代表PCR扩增前物种的原始组成,尤其当部分序列存在PCR扩增偏好时,Reads数被人为提高,从而导致定量不准,这些重复是需要去除的。
总的来说,PCR扩增之前的重复需要保留,PCR扩增之后的重复需要去除。怎么实现呢?UMI(Unique Molecular Identifier)数字标签技术这时候就派上用场了,只要在PCR扩增之前给每个分子加上一个特有的标签,之后无论经过多少个循环的扩增,这个标签都一直伴随着同步进行复制,最后可以通过UMI的种类对真重复和假重复进行区分,从而达到去除扩增重复的目的。下图分别展示了常规两步法建库和利用UMI数字标签建库的流程:
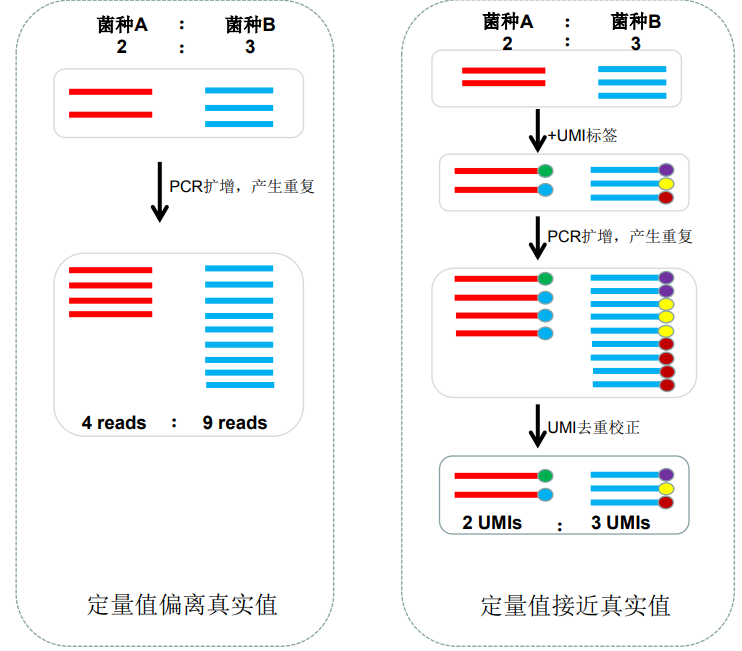
图二:16S扩增子建库流程 ,左,常规两步法建库流程,右,UMI数字标签建库的流程
从上图不难看出,既往基于测序reads数进行微生物群落多样性定量,定量值偏离真实值,在采用了UMI数字标签之后,根据UMI标签的种类进行定量,定量值更接近于真实值,校正了由PCR扩增偏好带来的影响。
UMI数字标签真就如此神奇吗?适用于哪些微生态场景的研究呢?咱们下期再来分享。


