
2020-07-21
群体进化前面两期带大家实战了基于群体SNP的PCA和进化树分析(点击查看),我们今天带大家进行实战群体遗传结构分析,依然含分析和绘图代码哦。
所谓群体遗传结构(Structure)是指遗传变异在物种或群体中的一种非随机分布。按照地理分布或其他划分标准可将一个群体划分为若干亚群,处于同一亚群内的不同个体亲缘关系较高,而亚群与亚群之间的个体则亲缘关系稍远。群体结构分析有助于理解进化过程,并且可以通过基因型和表型的关联研究确定个体所属的亚群。
群体进化研究的“三驾马车”中通过PCA和进化树分析可以直观了解群体中个体间的分类关系。但如果我们要探究:
目标群体划分为几个亚群更合理?
直接按照地域划分的群体分组是否合理?
群体间是否存在基因交流?
以及每个个体混血程度是多少?
这些疑问PCA图和进化树树形图都无法告诉我们答案,这时候就该高大上的Structure分析的遗传结构图出场啦!

图1 遗传结构分析结果图
虽然图形很高大上但看到花花绿绿的条形柱图你是不是也心存疑惑,这张图代表什么生物学意义呢?怎么解答前面的提出来疑问呢?接下来我们先帮大家一一解惑。
在进行Structure分析时我们只是获得了样本的基因型,并不知道这个群体实际包含几个亚群。把群体的亚群数称为K值,可以先预设群体亚群数等于1~n,即K=1~n,然后模拟在K=x的情况下,通过贝叶斯算法推算群体是如何分群以及每个个体的祖先来源。最后再根据每次模拟的最大似然值,找出划分这个群体的最佳K值。
具体解读以下面图2为例做详细的说明。
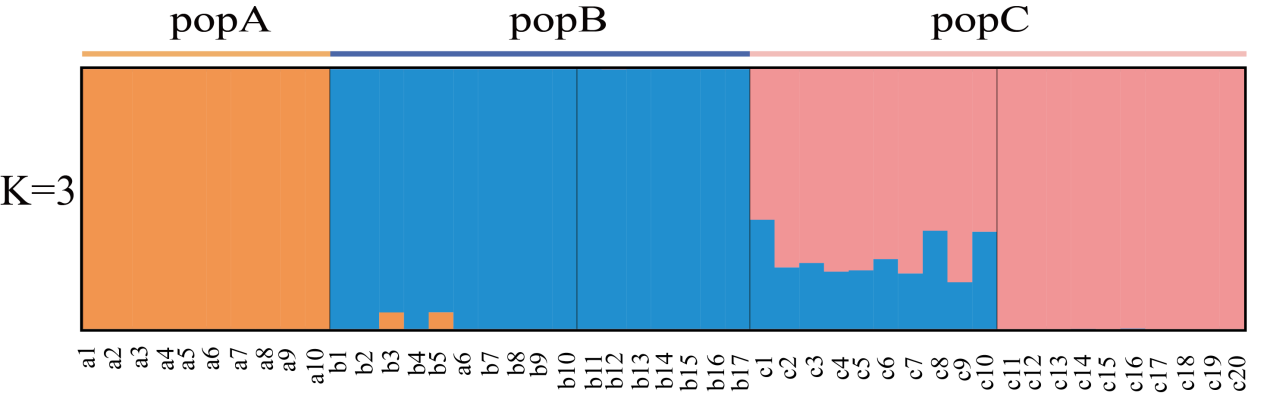
图2 K=3时群体遗传结构分布图
图2就是假设群体结构数的先验值K=3的模拟结果。每个直方柱子代表1个样本(这里popA包含10个样本,样本名a1~a10,popB包含17个样本,样本名b1~b17,popC包含20个样本,样本名c1~c20),柱子的颜色以及颜色比例代表这个样本属于哪个亚群以及祖先来源构成比例是多少,可以很直观的通过颜色就可以判断这47个个体是如何划分为3个亚群体(橙、蓝、粉)。同时也可以看出有些样本的祖先来源单一(仅有1种颜色),有些样本却由2个颜色组成。例如样本c1,它对应的直方柱子由两种颜色构成,大概是37%的蓝色和63%的粉色。说明这个个体很有可能是从两个祖先亚群杂交而来,且两个祖先亚群各占了37%和63%的血统。
但是每一个K值都对应着一个结果图,哪一个K值才是最准确的呢?这里就要引出下面图3的这张图啦。
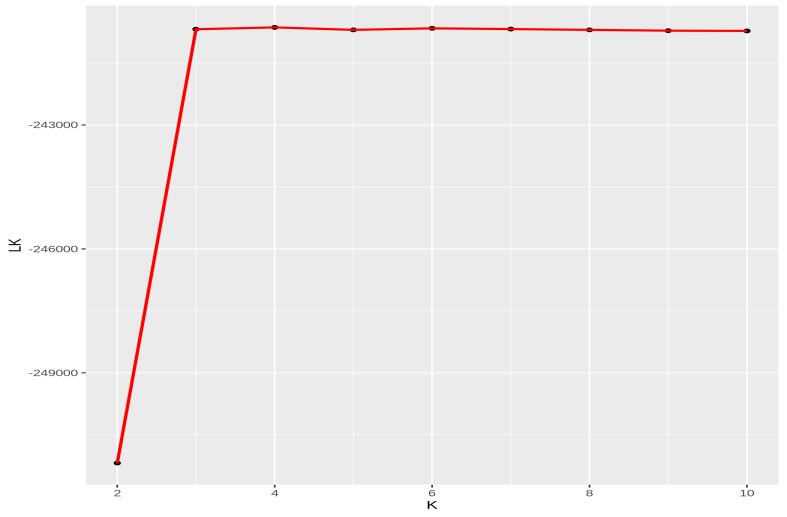
图3 LK分布图
每个K值基于贝叶斯模型的计算方法模拟的结果,都会产生对应的最大似然值(likelihood),LK值是取了自然对数后输出的(ln likelihood)。ln likelihood越大,说明K值越接近于真实情况,但一般随着K值升高,ln likelihood值也会进入平台期。最优K值就是进入平台期的那个拐点K值。
看到这里,大家是不是都想知道怎么才能实现群体遗传结构分析呢?接下来我们就带大家一起实战遗传结构分析。实战示例见以下链接:
链接:https://pan.baidu.com/s/1o1gNmyrQE2AA94pa-xC-8w
提取码:56bm
软件Structure、FastStructure、Admixture都可以进行遗传结构分析,Structure的运行速度较慢,Admixture使用的模型与Structure相同,但其凭借高速的运算速度逐渐成为群体遗传结构分析的主流软件,接下来就以软件Admixture的使用方法为例带大家逐步进行分析。
VCF文件格式转换
利用软件vcftools将群体SNP的vcf文件转换成Plink可以分析的格式,具体转换命令如下:
usage: vcftools --vcf test.vcf --plink --out test
##--vcf 输入vcf文件
##--plink 将数据转换为plink格式
##--out 输出文件前缀
得到的结果文件test.map和test.bed。
SNP过滤
在Structure分析过程中需要满足两点,其一,各个亚群内部个体应该符合哈代-温伯格平衡;其二,每个SNP位点是独立的不连锁的。一般基于重测序得到所有SNP作为Structure分析的输入文件是不对的,需要利用Plink进行SNP的过滤,生成.bed文件,命令如下:
plink --noweb --file test --maf 0.05 --hwe 0.0001 --make-bed --out filter
##--noweb 不连接网络
##--file 指定输入文件
##--maf 过滤掉低于次等位基因频率阈值的位点
##--hwe 过滤掉低于Hardy-Weinberg平衡检验p值低于阈值的位点
##--make-bed 数据转换为二进制格式
##--out 输出文件前缀
Admixture计算和K值的确定
K是假设的群体亚群或者祖先数,为确定最理想的K值,可以设定K=1~10,用软件Admixture进行计算,命令如下:
for i in {1..10};do admixture --cv filter.bed $i|tee out${i};done
从结果文件中提取出Cross-validation error值即不同K值的错误率,一般认为CV error最小值对应的K值为最佳K值。提取CV error命令如下:
grep -h CV out|awk -F ':' '{print NR"\t"$2}'|sed '1i\\K\tCV_error' > CV_for_plot.txt
然后将结果在R中可视化,最终得到CV error的分布图图4。具体代码如下:
library(ggplot2)
mydata<-read.table("CV_for_plot.txt",header=T,sep="\t")
qplot(x=K,y=CV_error,color=I('black'),data=mydata)+geom_line(color="red",lwd=1)

图4 CV error的分布图
根据结果显示K=3对应的CV error最小,故为最佳K值。
遗传结构图形绘制
根据K=3时软件Admixture计算的结果文件filter.3.Q利用R软件绘制遗传结构分布图。
具体画图代码如下:
mydata<-read.table("filter.3.Q")
barplot(t(as.matrix(mydata)),col=c("#E69F00","#56B4E9","#CC79A7"),border=NA)
然后就得到我们的目标结果啦!
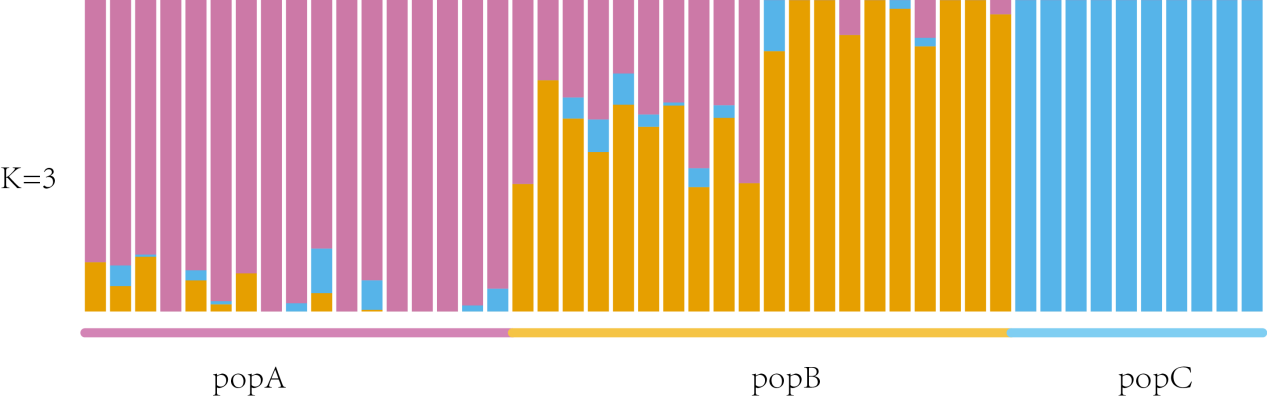
图5 K=3时遗传结构分布图
以上就是基于群体SNP进行遗传结构分析和图形绘制的方法,是不是没有想象的复杂呢,赶快用自己的数据试试吧!没有服务器资源的老师也可以用Structure软件的windows的Java图形界面版本试试,该软件包含三个主要参数length of burn-in period、Number of MCMC Reps after burn-in、admixture model,前两个参数一般设置为10000,第三个参数是该软件涉及的两种模型 no admixture model 和admixture model,前者假设亚群间不存在杂交,后者假设亚群间存在杂交,一般情况下选择admixture model更合理哦~
不管基于哪种软件如果大家实战分析的过程中有任何疑问,欢迎和我们一起探讨,可在文末留言或者邮件交流(genome_support@personalbio.cn)。
下面是动植物产品线的产品类型,如果对其他产品比较感兴趣的,也可以邮箱反馈我们,后续我们一一为大家安排。