2020-07-30
自2017年,“人类细胞图谱计划”启动以来,单细胞测序技术越来越受到科学家的青睐。
单细胞转录组测序相比于常规的整体的转录组测序,将视角聚焦到每一个单独的细胞,从转录组角度揭示了细胞之间的差别和联系,解析组织内部的异质性。仅从10x genomics平台的数据统计就可以看出,自2017到目前为止, 10x genomics单细胞转录组发表文献就有1015篇。
除了在单个细胞水平去解决组织异质性问题,细胞间的空间位置无疑是决定组织异质性的另一个重要因素。针对这一重要问题,10x genomics在2019年推出了革新的空间转录组技术,首次将目光聚焦到组织内部的精确空间范围内,揭示空间范围内的转录组信息,为科研工作者提供转录谱信息,有助于揭示精确空间范围内发生的信号传导通路和细胞行为,解析目标生物学过程的更深层次的分子机制。

既然空间转录组这么热,那么就让咱们带着几个问题去了解下空间转录组的相关信息,做个简单的入门:
为什么要做空间转录组?
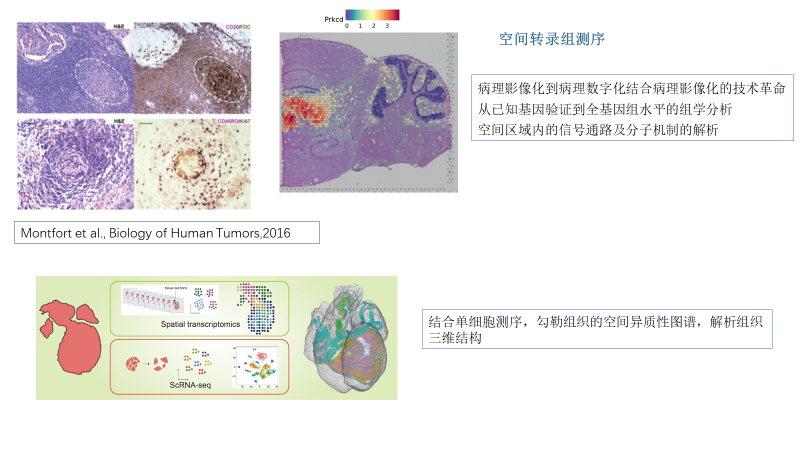
以肿瘤微环境为例,提出一系列的生物学问题。例如,哪一些细胞类型激活了怎样的信号通路在特定部位促进了肿瘤干细胞的产生?肿瘤细胞以及临近的肿瘤相关成纤维细胞(CAF)如何影响周围细胞,促进肿瘤发展?免疫细胞如何感知微环境的局部信号,招募更多的免疫细胞,促进免疫浸润以及在肿瘤附近形成三级淋巴结构,介导肿瘤免疫反应?肿瘤细胞如何抵制附近不同类别的免疫结构,完成肿瘤转移等等。以往的mRNAseq以一个肿瘤组织作为个体进行测序,无法揭示内部精确的信号传导过程,针对以上的问题,无法给出明确的答案。通过单细胞测序和空间转录组测序,则可以揭示非常精确全面的表达谱信息,帮助研究者分析特定空间位置、特定细胞类型、特定生物学过程中的信号传导过程,揭示其分子机理。
空间转录组测序是什么原理?
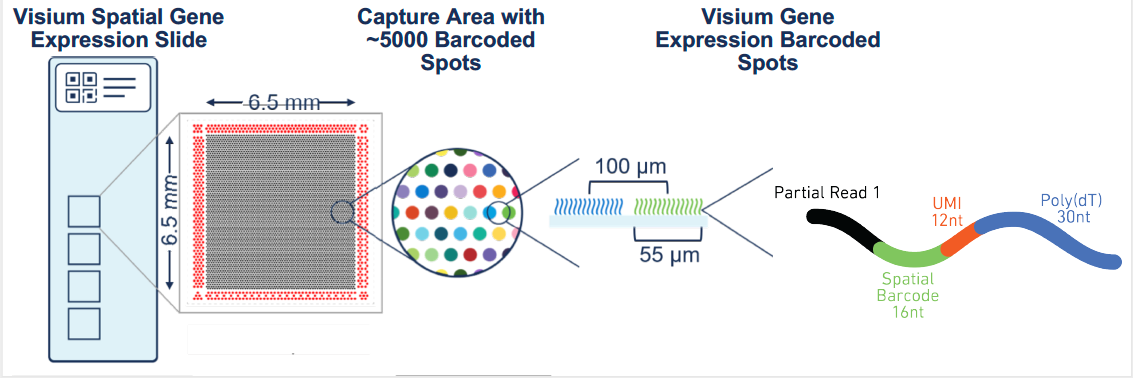
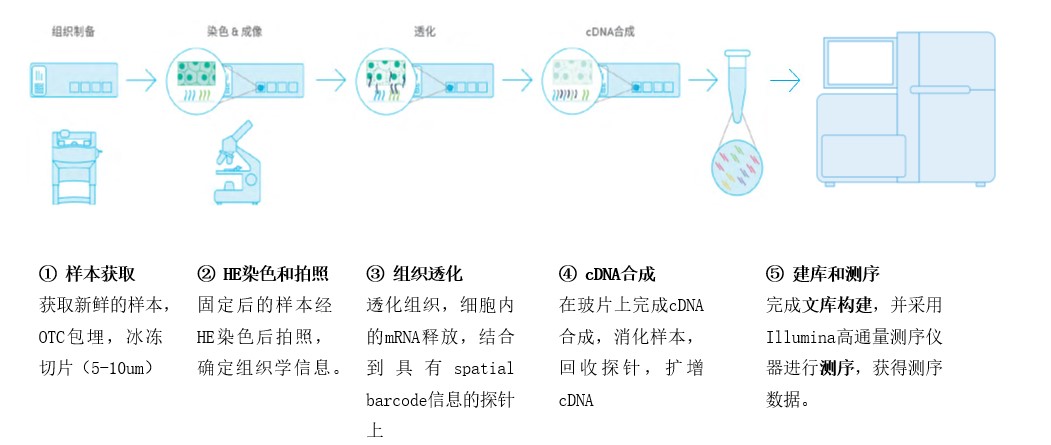
10X空间转录组测序主要在含有空间barcode的玻片上完成。冷冻切片的样本放在此玻片上,组织透化后样本的mRNA与玻片上的含有空间barcode的polyT捕获序列结合,不同的mRNA都标记上了各自的空间信息,针对这些mRNA进行cDNA合成、建库和测序。测序结果中除了基因表达的信息外,也包含每个基因的空间信息,将此结果重新mapping到HE染色的图片中,研究者可以非常直观地看到感兴趣地区域内表达了哪些基因,激活了哪些信号通路,可能与周围的区域发生怎么样的细胞间通讯等一系列过程;当然,研究者也可从另一方面进行分析,感兴趣的基因或信号传导过程,发生在哪个区域,这些区域可以成为研究对象揭示其它基因的表达以及其它信号通路的激活。有了空间转录组测序的结果,大大拓展了思维的广度,帮助提出和解决更多的、更深入的生物学问题。
这一切,研究者需要提供的只是一个OCT包埋的样本。

怎么做空间转录组测序?
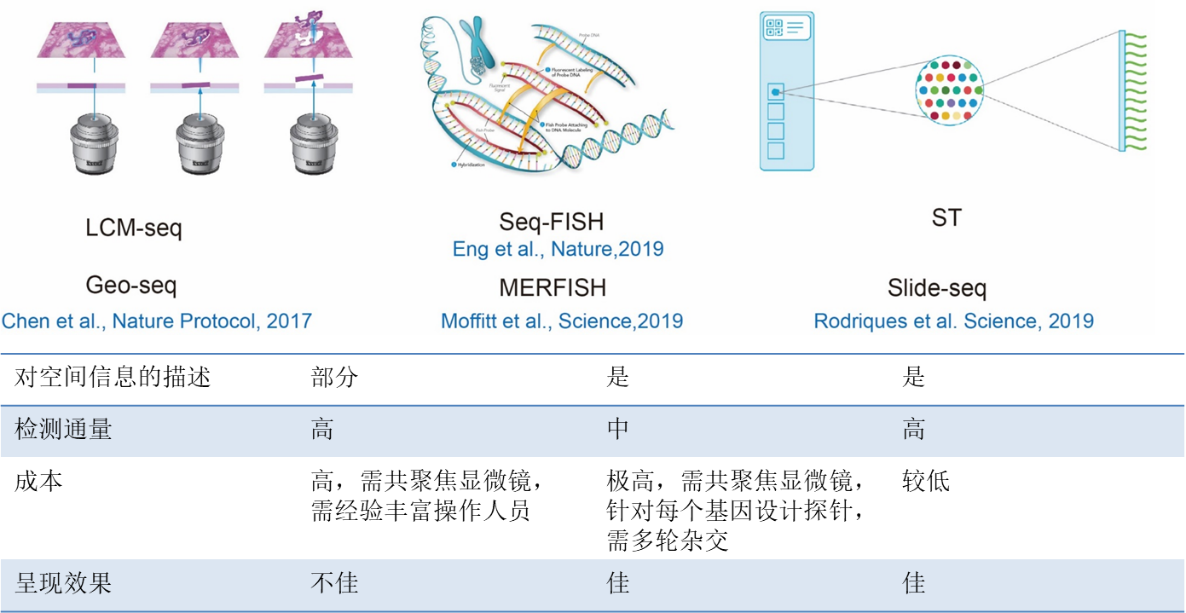
10X空间转录组测序的优势有哪些?
目前可揭示空间信息的分析方法还包括基于显微切割的RNA测序和免疫荧光等。然而,显微切割RNA测序无法给出测序样本内部的空间信息,即丢失了研究者关注的组织内部的空间信息,而免疫荧光只能针对已知基因进行表达模式的验证,对于未知基因的揭示则束手无策,此外,针对信号通路中的多个基因进行表达模式验证,免疫荧光则显得既耗时又烧钱。10X空间转录组测序解决了以上问题,在研究者感兴趣的区域内,揭示所有基因的表达模式。如果研究者的工作中涉及到多次免疫荧光,那么,可以考虑以下是否用空间转录组测序一步到位,解决所有问题。就像用RNA测序代替RT-PCR的解决方案一样。
10X空间转录组测序能发什么水平的文章?
目前阶段,10X空间转录组测序仍处于新技术的红利期,只对样本进行测序分析,不需要进行复杂的生物学验证,即可以发表CNS级别的文章。例如,2019年,science杂志发表了针对肌萎缩的小鼠和病人的脊髓样本进行空间转录组测序分析。2019年,cell杂志发表了针对人胚胎组织样本的心脏组织进行空间转录组测序分析。2020年,nature biotechnology杂志发表了针对胰腺癌样本进行的空间转录组测序分析。
以上文章通过新技术,结合简单的免疫荧光验证,解释了精确的生物学问题,获得了一流期刊的高度认可。
参考文献:
[1]Maniatis S, Äijö T, Vickovic S, Braine C, Kang K, Mollbrink A, Fagegaltier D, Andrusivová Z, Saarenpää S, Saiz-Castro G, Cuevas M, Watters A, Lundeberg J, Bonneau R, Phatnani H. (2019) Spatiotemporal dynamics of molecular pathology in amyotrophic lateral sclerosis. Science 364: 89-93.
[2]Asp M, Giacomello S, Larsson L, Wu C, Furth D, Qian X, Wardell E, Custodio J, Reimegard J, Salmen F, Osterholm C, Stahl P, Sundstrom E, Akesson E, Bergmann O, Bienko M, Mansson-Broberg A, Nilsson M, Sylven C, Lundeberg J. (2019) A Spatiotemporal Organ-Wide Gene Expression and Cell Atlas of the Developing Human Heart. Cell 179: 1647-1660.
[3]Moncada R, Barkley D , Wagner F, Chiodin M, Devlin J, Baron M, Hajdu C, Simeone D, Yanai T. (2020) Integrating microarray-based spatial transcriptomics and single-cell RNA-seq reveals tissue architecture in pancreatic ductal adenocarcinomas. Nature Biotechnology 38: 333-342.


