
2020-08-11
今年7月哥伦比亚大学、澳大利亚海洋科学研究所以及德克萨斯大学奥斯汀分校的研究者们在Science上联合发表了文章,他们通过对大堡礁中心约300km范围内一种造礁珊瑚进行群体遗传学研究,构建了一个可以预测珊瑚白化的统计模型,为全球珊瑚物种的保护提供了有利的工具。

项目背景
人为的全球变暖正在改变全球的生态系统,迫使物种迅速响应不断变化的环境。海水温度升高对海洋生态系统的影响尤其具有破坏性,使得全世界的珊瑚礁正以惊人的速度减少。珊瑚礁覆盖不到1%的海底,但却拥有者超过海洋1/4的生物多样性,对人类的重要性不言而喻。
升高的海水温度(甚至仅略高于长期最大值)也会导致珊瑚的白化现象,长期的白化最终可能导致珊瑚群落的死亡。面对迅速升高的海水温度,迫切需要采取新的保护策略,以防止珊瑚群落的大规模的损失。而这些工作的顺利开展,离不开对珊瑚白化遗传基础的清晰了解。
不同珊瑚物种和同种珊瑚内的不同个体对白化和对热胁迫的响应各有不同,存在表象差异;在印度洋-太平洋地区普遍分布的鹿角大珊瑚(Acropora millepora)中,研究表明其耐热性是部分可遗传的,因此,理论上个体间的白化差异应该可以从基因组数据中加以预测。
白化现象:温度或盐度变化带来的生态压力破坏了造礁珊瑚与其细胞内光合鞭毛藻(共生双鞭毛科)之间的共生关系,这种现象称为白化现象。
实验材料与方法
1 、基因组组装与注释
组装物种:A. millepora
测序策略:PacBio、10X Chromium barcoded单细胞测序法(illumina PE)
组装策略:pacbio数据组装contigs;10×单细胞数据连接scafflod和矫正,50×幼虫非共生基因组测序数据剔除共生藻类数据;简化(RAD)遗传图谱数据重排contigs;转录组数据预测基因及组装结果注释。
2、 样本收集及表型判定
样本选取: 从12个分布在大堡礁中心约300公里范围内的珊瑚礁中收集了253个鹿角珊瑚个体。
样本采样时间:2017.3.14~2017.4.1(白化高峰期)。
表型判定:白化等级判定,
1、视觉白化得分,Coral Color Reference Card ;
2、光合色素浓度(吸光度665、649、632nm);
3、共生体细胞密度,DC Protein Assay。
3、群体遗传学及微进化分析
测序策略:从12个群体中各选出4个个体进行>20x的高覆盖度重测序;
分析内容:连锁不平衡(LD)衰减分析,核苷酸多样性分析,历史有效种群大小(Ne)、有效迁移率、基因组单倍型 。
4、白化现象全基因组关联分析
测序策略:作者只对采集的珊瑚中48个个体进行了高覆盖度基因组测序,剩下的个体则是采用低覆盖度测序进行的。
分析方法:根据44个个体的重测序数据,用read-back phasing法构建了参考单倍型集合,并用此对低通量测序样本进行了基因型填补,排除了可能是物种鉴定错误、罕见的变异谱系、亲缘关系太近、以及表型数据缺失的异常值后,最终213个表型样本进行全基因组关联分析。
5、PGS(白化预测模型)的构建与验证
分析方法:基于LD的聚类和GWAS中估计的SNP效应量构建了多基因评分模型(PGS,或称育种值)。由于缺乏除了所研究样本之外的其他数据,用Jackknife交叉验证法评估了PGS的预测准确性。
结 果
1、参考基因组组装(物种:Acropora millepora(鹿角大珊瑚))
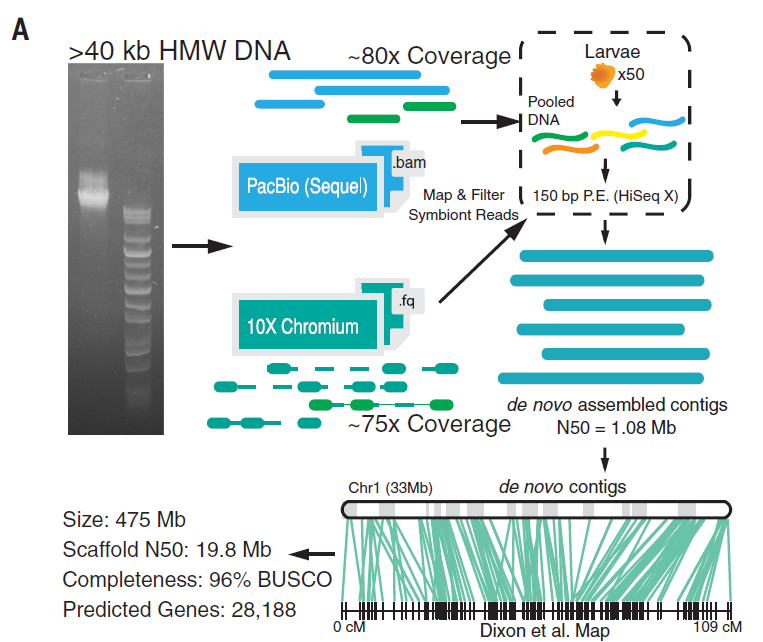
图1. 鹿角大珊瑚基因组组装及结果
最终获得了一个大小为475M的基因组,其scaffold N50值达到了19.8Mb。随后,用已发表的转录组数据,从得到的基因组中预测到了28,188个基因,其中超过96%的基因在真核生物中都存在单拷贝同源。
2、群体遗传学样品采集
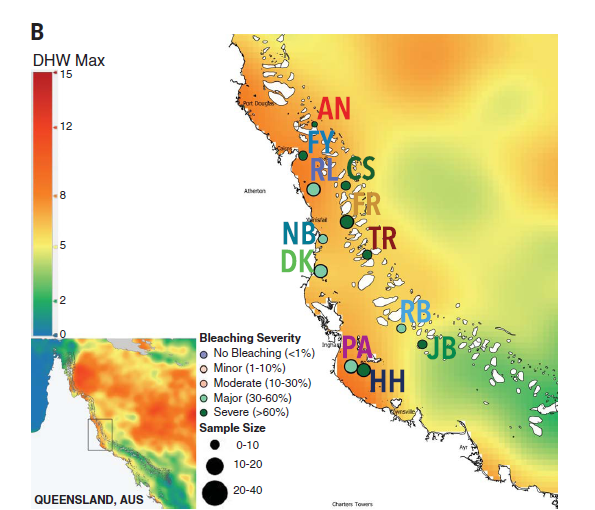
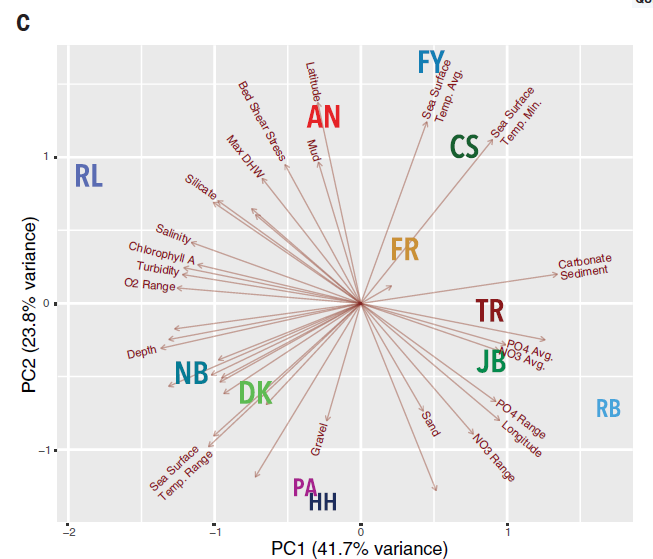
图2. 采样地分布图(B)及环境因子主成分分析(C)
为了研究与白化响应相关的遗传变异,2017年3月的一次白化高峰期间,研究者们从12个分布在大堡礁中心约300公里范围内的珊瑚礁中收集了253个鹿角珊瑚个体样本(图2B),并采集了这些珊瑚栖息地的生态环境数据。
将40个不同的环境空间参数进行整合分析,发现采集的珊瑚样本的分布深度介于1.2-7.2m之间,涵盖了鹿角大珊瑚在礁坪和礁上斜坡上的通常分布深度范围。采样地的生态条件则随经度和纬度不同而变化,12个采样地的月平均海水表层温度差异超过了0.5℃(图2C)。
3、白化表型评估
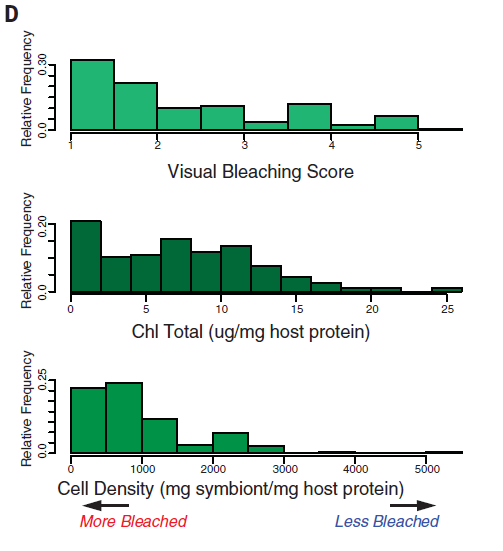
图3 白化程度表型评估
对这些珊瑚个体的视觉白化得分、光合色素浓度、共生体细胞密度这三个指标进行测定,用以衡量其白化程度。结果显示,这253个珊瑚个体的白化表型间存在较大的变异。
4、连锁不平衡衰减及历史种群大小分析
从12个群体中各选出4个个体进行>20x的高覆盖度重测序,获得了将近680万个高质量SNPs位点。经主成分分析,剔除掉了4个可能存在鉴定错误或罕见谱系分化的离群个体,保留了44个不存在亲缘关系的个体进行后续分析。

图4. 鹿角大珊瑚连锁不平衡(A)、核苷酸多态性(B)及历史种群大小(C)分析
从获得的680万个SNP位点中随机选出1%进行连锁不平衡(LD)衰减分析,结果显示当两个位点间的距离达到约15kb时,其平均r2值就降到了0.05以下(图3A)。对非重叠基因间区1 kb窗口内的核苷酸多样性(π,即每个碱基对的平均两两差异)进行计算,得到的平均π为0.363%(图3B)。如以每代每个碱基对的突变率为4×10e−9为标准计算,这个多样性值对应的长期有效种群大小(Ne)约为2.26×10e5。
对每个测序个体进行成对序列马可夫共祖(PSMC)分析,推断出了这些珊瑚群体在过去约一百万代内的Ne变化情况。结果显示,与在其他珊瑚中发现的类似,采集的多孔轴孔珊瑚的Ne在过去的一百万代内均呈稳定下降趋势,并且这些珊瑚群体的种群演变轨迹高度相似(图3C),表明这些多孔轴孔珊瑚的祖先群体很可能具有很大的种群大小和群体结构,而相似的种群动态历史则反映这12个采样地的珊瑚礁间可能存在高度的连通性。
5、群体结构分析
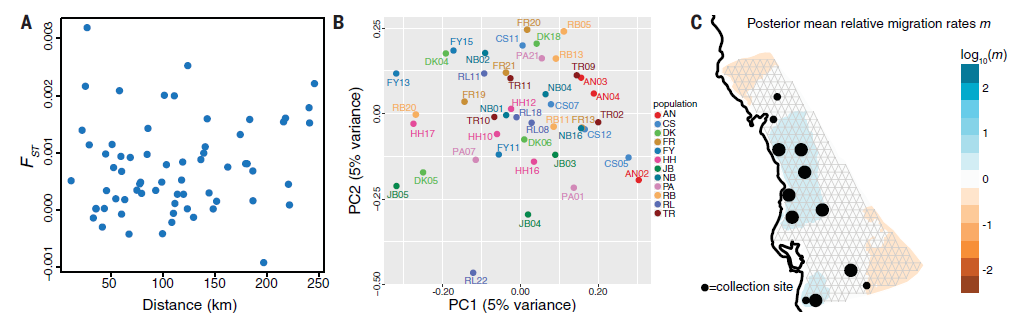
图5 鹿角大珊瑚群体结构分析
研究者们对这12个群体的遗传距离(FST)进行了计算,结果发现,尽管采样地间的环境差别很大,但这些珊瑚样本间的FST几乎均接近0,并且其FST也与地理距离无关(图5A)。PCA分析结果也显示这12个群体间不存在明显的群体结构(图5B)。对这些珊瑚群体的相对有效迁移率的评估结果也显示,取样的珊瑚间不存在明显的基因流障碍(图5C),证实了这些群体间的遗传分化程度确实很低!
分析其原因,这种跨越如此大地理范围的基因流模式,很可能是鹿角大珊瑚广泛产卵的繁殖方式造成的。
6、热休克共伴侣蛋白分析
尽管这12个群体几乎不存在遗传分化,但其分布范围内的环境条件尤其是热状况的差异却很大。自然状态下,成年珊瑚个体只与附近的个体繁殖,但珊瑚幼虫却可以扩散到更远的范围,因而每一代幼虫在不同的珊瑚礁定居下来后,都可能受到很强的选择。模型表明,在持续迁移中因强烈空间异质性受到的选择,会使生物基因组内一些局部有益的等位基因被长期保留下来。
为了寻找鹿角大珊瑚适应性相关的变异,作者们对基因组中多样性高的区域进行扫描,因为这些区域可能是长期平衡选择的结果。经扫描,发现整个基因组中多样性最高的区域位于7号染色体上,该区域包含一个sacsin的直系同源基因(图6A)。单倍型频率分析显示,该区域的h12统计值也很高,表明这个区域存在受选择的单倍型(图6B)。将基因组每10-kb窗口的序列进行PCA分析,发现与基因组其他部分相比,该区域在PCA结果中也显得格外突出,证实这种亲缘关系模式并不寻常。所有这些结果都指向一个异乎寻常的系谱关系——即在进化上,包含sacsin基因的两个单倍型的分化时间甚至比柔枝轴孔珊瑚(A. tenuis)和指形轴孔珊瑚(A. digitifera)的分化还要早(图6C)。
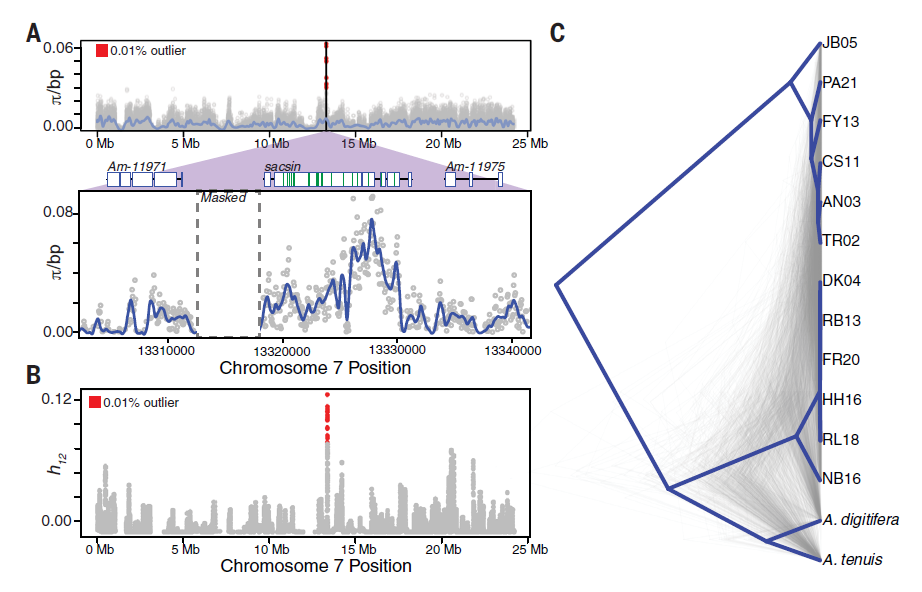
图6 全基因组多态性扫描及sacsin基因进化树分析
Sacsin是热休克蛋白Hsp70的共伴侣,包含多个与Hsp90同源的区域。轴孔珊瑚属(Acropora)和杯型珊瑚属(Pocillipora)中多种珊瑚的sacsin基因在实验室内高温下均显著上调表达,表明该基因的变异可能与自然种群的热响应有关。sacsin基因显示的高度多样性表明该基因是平衡选择的目标,位于该基因处的变异已经维持了相当长的时间(数百万代),因此在野外和实验室中都具有重要的功能。进一步对sacsin基因单倍型频率与环境因子的相关性分析显示,该单倍型的频率可能与共生藻的类型有关(P = 0.059),但与遗传或环境主成分之间均无相关性(P > 0.15),不过整体上仍不清楚具体是哪种因素造成sacsin基因受到强烈的平衡选择。
7、白化表型全基因组关联分析
用这213个个体的基因型数据再次对12个群体进行群体遗传学分析,结果仍显示这12个群体间几乎没有群体分层、存在高度基因流,并且在scasin基因处的多态性最高。
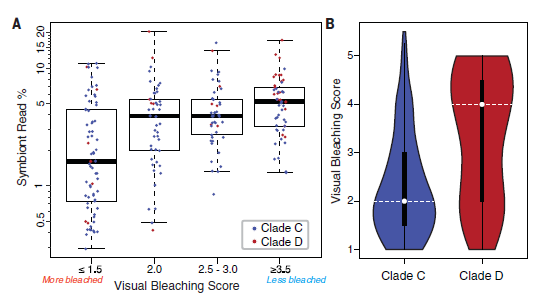
图7 共生藻组成和白化之间相关性分析
此外,测序也获得了共生在珊瑚体内共生藻的基因组数据。对每个珊瑚样本中的共生体种类进行分析,发现珊瑚样本中共生体reads的丰度随视觉白化得分这一表型的不同而存在显著差异(图7A),并且也与叶绿素总含量和共生体细胞密度相关。通过计算比对到珊瑚基因组上的reads的相对比例,进一步确定了共生体的组成类型,发现以Durusdinium为优势共生体的珊瑚拥有更高的白化视觉得分(图7B),这与先前研究发现的,野外环境中以Durusdinium为优势共生体的珊瑚通常具有更高的耐热性(但其生长速度较慢)相一致。整体上,这表明使用低覆盖度测序方法可以在对白化进行定量的同时,确定共生体的相对组成类型。
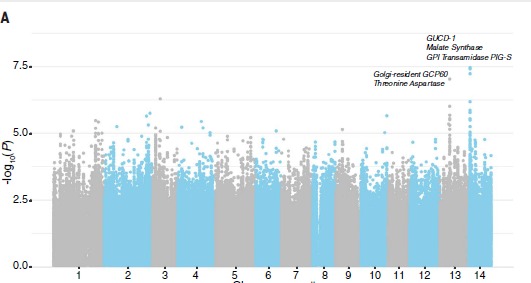
图8 视觉评分表型曼哈顿图
接下来,作者用线性混合模型(LMM)对3个白化表型指标与获得的基因型数据进行GWAS分析,并将环境变异主成分的前4维、遗传变异主成分的前两维、批次效应、采样深度、共生体reads中Durusdinium所占的比例作为协变量加入模型中。三个测定的白化表型间存在高度相关,但因叶绿素含量(n=190)和共生体细胞密度(n=172)的表型有缺失,作者们把重点放在了视觉得分(n=213)的GWAS结果上。结果在基因组上没有检测到与白化显著相关的SNP位点,表明不存在与白化变异相关的主效基因座(图8)。
8、自然珊瑚个体白化响应的预测模型构建
上述GWAS分析结果显示,鹿角大珊瑚白化的表型变异是受多基因控制的,因此难以鉴定致因位点,这和桌形轴孔珊瑚耐热性研究的结果一致。尽管如此,有了对基因组上每个SNP位点效应的评估结果,就可以帮助我们预测表型。因此,研究者们用基于LD的聚类和GWAS中估计的SNP效应量构建了多基因评分模型(PGS,或称育种值)。由于缺乏除了所研究样本之外的其他数据,他们用Jackknife交叉验证法评估了PGS的预测准确性。

图9 模型预测的准确性评估
为了增加模型的预测准确性,他们将批次效应和遗传效应主成分作为协变量加入模型中。此外,考虑到环境和遗传因素都有会影响白化的变异,因此将环境效应、优势共生体类型(以测得的Durusdinium的reads比例表示)这两个因素也加进来。经交叉验证后发现,在上述线性模型中加入这些预测因子后,模型的预测精度明显提高(图9B)。尤其地,后两者的加入使得模型的准确性得到了极大提升。总的来说,这个模型对珊瑚样本视觉白化得分这一表型变异的解释率达到了62%左右。不过,进一步讲,这一模型的准确性还需使用另一个不同的样本集进行验证,仅仅靠交叉验证还不够。
在制定保护策略时,白化预测模型可以帮助区分不同栖息地中更有可能耐受的个体,为当前的空间保护策略和对珊瑚礁未来的预测提供信息。使用上述完整的预测模型和交叉验证程序,预测得到的表型值最高的1/4个体的平均视觉得分明显高于最低1/4的个体(图9C)。该方法也可以帮助识别最耐受的个体,用于管理移种和选择育种,以辅助基于进化的保护策略。在这点上,多孔轴孔珊瑚在如此大地理距离上缺乏种群结构的特点,使其成为一个理想的系统,可以从多个地方繁殖具有高预测特征值的珊瑚。此外,该模型的预测还可用来增加这些珊瑚在本地种群中的数量,而不需要通过辅助基因流来转移它们,从而降低基因渗入风险和伦理问题。
主要结论
1、为2017年白化高峰期在澳大利亚大堡礁中部12个珊瑚礁那里收集的237个样本进行了染色体尺度的基因组组装,并获得它们的全基因组序列 ;
2、用规模适中的参考单倍型panel(reference haplotype panel)可靠地推算低覆盖率测序数据中的基因型,从而为未来大规模的全基因组测序工作提供了一种具有成本效益的方法;
3、群体结构分析表明在被采样的珊瑚礁上检测到的种群结构非常少,推测可能是由于鹿角大珊瑚的广播式产卵繁殖模式造成的;
4、在这种基因组背景下,他们在热休克辅助伴侣蛋白(heat-shock co-chaperone)sabsin中检测到异常古老的变异,这与作用于该基因的长期平衡选择是相一致的;
5、他们的基因组测序方法同时提供了对白化的定量测量,并确定了珊瑚宿主内存在的共生物种的组成;
6、对白化现象进行了全基因组关联分析,没有找到明显的关联基因组,这表明白化反应的变异并不是由具有较大效应的共同位点造成的;
7、整合了根据全基因组关联数据、相对共生物种组成的基因组数据、环境变量估计的遗传效应的模型可预测个体白化表型。
本文研究高温和白化的遗传学对于预测珊瑚适应气候变化和珊瑚礁生态系统的未来至关重要。这些知识也支持常规的管理方法和新的干预措施的开发。这项研究提供了对白化反应的遗传结构的新见解,并作为在保护工作中使用基因组方法的原则证明。这些研究人员发现,一种基于环境因素、共生物种的基因组数据和珊瑚宿主全基因组关联数据的模型,可以帮助区分最耐受白化的个体和最容易受白化影响的个体。因此,这些结果为从基因组角度预测鹿角大珊瑚和其他珊瑚物种的白化反应奠定了基础。
本文部分内容参考了公众号“AGCT与鱼”,若有侵权,请联系删除。
文章索引:
Zachary L. Fuller*, Veronique J. L. Mocellin, Luke A. Morris et al. Population genetics of the coral Acropora millepora: Toward genomic prediction of bleaching. Science 369, 268 (2020)