2020-08-13
遗传图谱又称连锁图谱、遗传连锁图谱。如果说物理图谱指的是碱基在DNA上的线性排列,那么遗传图谱则指基因或DNA分子标记在染色体上的排列顺序了,根据选定的基因或分子标记,以及这些标记之间的重组率为“图距”确定不同多态性标记位点在每条连锁群上排列的顺序和遗传距离所得到的图谱就称为遗传图谱。
遗传图谱的单位为厘摩(cM),即基因或DNA片段在染色体交换过程中的重组频率。距离越远,发生重组的概率越高。
遗传图谱主要有四个方向的应用:数量性状位点(Quantitative trait loci , QTLs)定位、辅助基因组组装、比较基因组分析、分子标记辅助育种等[1]。遗传图谱构通过遗传重组分析将各类标记根据重组率定位在染色体上。
因此,开发分子标记在遗传图谱的构建中是非常重要的环节。传统的遗传标记通过表型间接的反映基因的差异,而分子标记以DNA分子的多态性为基础,能够更加直接的反映DNA水平的遗传变异[1]。
随着分子标记的不断发展,目前已经有几十种不同的分子标记技术,这些标记技术各有优缺点,随着标记技术的不断发展,目前常用的标记主要有:RFLP (Restriction Fragment Length Polymorphism)即限制性片段长度多态性、RAPD(Random Amplified Polymorphism DNA)即随机扩增多态性 DNA、 AFLP(Amplified Fragment Length Polymorphism)即扩增片段长度多态性 、 SCAR(Sequence Characterized Amplified Regions)即序列特异扩增区域、CAPs(Cleaved Amplified Polymorohic Sequence)即切断扩增多态性序列、SSR(Simple Sequence Repeat)即简单重复序列或微卫星 DNA 标记和SNP(Single Nucleotide Polymorphism)即单个核苷酸多态性标记等[1,2]。
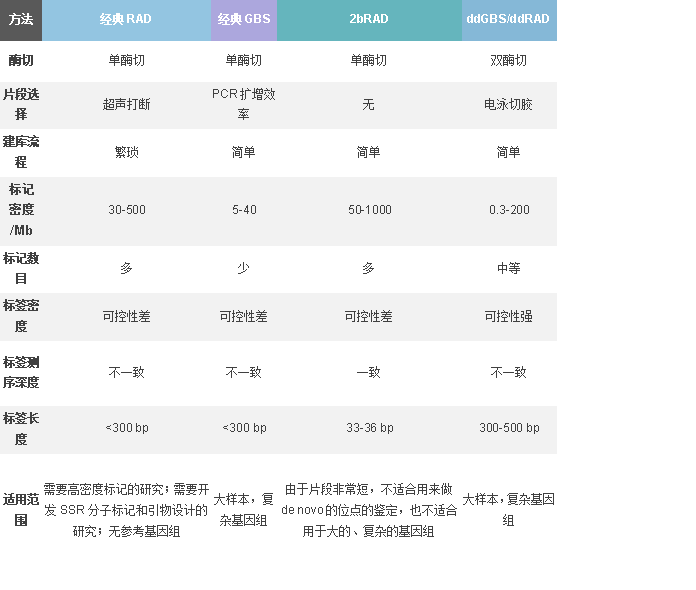
重复碱基类的分子标记 当前,测序技术不断发展,将高通量测序技术应用于分子标记的开发,能够更加快速的得到更加丰富的分子标记。将高通量测序技术用于分子标记开发能够节约大量的时间和成本,常用于开发SSR、SNP等标记方法。 真核生物基因组包含大量由1 - 6 bp的重复 DNA 片段构成的特定序列,长度在1000bp以内,这段序列就被称为SSR序列[4]。SSR序列的突变率高,且对于不同的等位基因,其重复单元的重复次数差异很大;但其两端一般为高度保守的单拷贝序列。在DNA修复或复制时,DNA序列发生错配错位导致重复序列数目发生变化;在减数分裂、有丝分裂中姐妹染色单体交换的不对等性也会产生SSR序列[5]。我们可以根据其两端保守序列设计引物扩增,其重复区域拷贝数不同导致最终片段长度也不同,根据分离片段的大小判断基因型,并计算等位基因发生频率[6]。 SSR是PCR技术类的分子标记代表,其被广泛应用与各类物种的遗传作图研究中[5,7,8,9]。此外,还有报道SSR在种间也有良好的通用性[10]。 根据SSR的特点,其优点是显而易见的: 共显性标记检,测单一的多等位基因位点; 鉴别杂合子和纯合子; 检测结果重复性好,稳定可靠; 所需 DNA 量少。 早期测序手段欠缺,所以SSR标记的前期开发成本较高,原因是我们必须得到扩增SSR序列两端的保守序列;而很多时候,序列本身或其旁侧序列并不清楚,这极大地限制了该技术的应用[11,12]。 传统的开发的方法有很多,主要可分为以下三类:利用数据库/相关文献、利用近缘物的序列扩增或从gDNA、cDNA、EST中筛选。现在,测序技术不断发展,我们利用高通量技术,通过构建一个SSR文库(图一),一次测序就能得到大量的SSR序列并统计这些序列的多态性(图二)。通过高通量测序能够极大地节约开发成本和时间,且有无参考基因组均可,适用性较广。 图一 SSR分子标记开发流程 图二 派森诺SSR分子标记开发结果展示 单碱基的分子标记 单个核苷酸多态性标记(Single Nucleotide Polymorphism,SNP),指DNA上单个核苷酸的变异(如突变、插入和缺失等)导致的DNA序列多态性[13,14]。 SNP位点普遍存在于生物的基因组中,目前已经发展成为最广泛的标记[15,16]。在不同样本之前开发SNP标记,目前常用的测序策略就是简化基因组测序和全基因组测序两种方案。 以往,出于成本考虑,研究者通常采取对全基因组的一部分进行测序的方案(即简化基因组测序,Simplified genome sequencing) ,其核心策略就是利用限制性内切酶来得到全基因组的一部分,对该部分测序以达到测相同数据量获得更高测序深度的目的。目前简化基因组的测序方案多种多样,常用的方法主要为RAD(Restriction site Associated DNA)和GBS(Genotyping-By-Sequencing),其余如2b-RAD,dd-RAD,SLAF等方案都是以RAD和GBS为基础的改良方案,各简化方案详细比较如表一所示。RAD和GBS的主要区别在于GBS仅使用限制性内切酶对DNA片段化并测序,而RAD先用限制性内切酶酶切DNA,随后经行超声波随机打断。目前常用的简化基因组建库方案中最常用的就是RAD和dd-RAD方案了,图二是两种方案的流程,可以明显发现RAD方案比dd-RAD方案更加繁琐,成本更高,但是RAD方案更加适合无参考基因组的物种,所以当我们确定实验方案时还是要根据需要来选择合适的方案。 以往很多遗传图谱基于简化基因组测序,遗传图谱的饱和度相对不足;另一方面,简化基因组测序由于其自身特点导致测序结果在全基因组中覆盖度较低,对后续实验有一定的影响。相比于简化基因组测序,全基因组测序可以更快更准确的开发大量适于遗传图谱构建的分子标记并构建出高密度的遗传图谱。随着测序成本的大幅下降、对遗传图谱的密度要求越来越高,在遗传图谱构建方面,全基因组测序已经比简化基因组测序更具优势了。 表一 各简化方案比较 图二 RAD和dd-RAD方案实验流程 1.克拉克,顾红雅,瞿礼嘉.植物分子生物学——实验手册[M].北京:高等教育出版社,1998. 2.周延清,杨清香,张改娜.生物遗传标记与应用[M].化学工业出版社,2008.6 3.徐礼羿. 茶树SNP高密度遗传连锁图谱构建与数量性状候选基因挖掘[D]. 2019. 4.Grover A, Aishwarya V, Sharma PC. Searching microsatellites in DNA sequences:approaches used and tools developed. Physiology and Molecular Biology of Plants,2012, 18: 11-19. 5.缪体云. 结球甘蓝遗传图谱的构建及主要农艺性状的QTL定位[D]. 中国农业科学院, 2007. 6.罗文永,胡骏,李晓方.微卫星序列及其应用[J].遗传,2003.25(5):615-619 7.Hearnden PR, Eckermann PJ, Mcmichael GL, Hayden MJ, Eglinton JK, Chalmers KJ.A genetic map of 1,000 SSR and DAr T markers in a wide barley cross. Theoreticaland Applied Genetics, 2007, 115: 383. 8.Isobe SN, Hirakawa H, Sato S, Maeda F, Ishikawa M, Mori T, Yamamoto Y,Shirasawa K, Kimura M, Fukami M. Construction of an integrated high densitysimple sequence repeat linkage map in cultivated strawberry (Fragaria× ananassa)and its applicability. DNA research, 2012, 20: 79-92. 9.陈冬明, 刘长清, 朱连发,等. 基于SSR分子标记的鸭茅指纹图谱构建及遗传多样性分析[J]. 草学, 2020(3). 10.Zhou CP, He XD, Li FG, et al. 2014. Development of 240 novel EST-SSRs in Eucalyptus L'Hérit. Molecular Breeding, 33(1):221-225. 11.车永和.几种代表性分子标记技术[J].江苏农业科学,2003,2:3-5.E523 12.曹仪植.植物分子生物学[M].北京:高等教育出版社,2002 13.Sachidanandam R , Weissman D , Schmidt S C , et al. A map of human genome sequence variation containing 1.42 million single nucleotide polymorphisms[J]. 2001, 409(6822):928-933. 14.Mammadov J, Aggarwal R, Buyyarapu R, Kumpatla S. SNP markers and their impact on plant breeding. International journal of plant genomics, 2012, 3: 728398. 15.Rafalski, Antoni J. Novel genetic mapping tools in plants: SNPs and LD-based approaches. Plant Science, 2002, 162: 329-333. 16.Jiang Z, Wang H, Michal JJ, Zhou X, Liu B, Woods LCS, Fuchs RA. Genome wide sampling sequencing for SNP genotyping: methods, challenges and future development. International journal of biological sciences, 2016, 12: 100-108.