2017-08-31
发表期刊 Nature Genetics
影响因子 27.96
在GWAS分析中找不到Peak SNP肿么办?如果你也有这样的苦恼,我们一起来看一看高大上的文献是怎么做到的?最近在Nature Genetics上刊登了一篇陆地棉选择消除分析和GWAS相结合的文章,该研究利用基因组重测序技术对陆地棉在驯化过程中受到的选择区域进行鉴定和验证,并对控制皮棉产量和棉纤维质量等多个重要农艺性状相关的基因进行快速定位,而且定位效果非常不错哦,部分性状直接到单基因水平!
研究背景
陆地棉是世界上最重要的棉花栽培品种,占全球棉花种植面积的90%以上。然而其栽培种的遗传多样性以及在改良过程中一些遗传特性的变化仍然未知。
研究方法
对318株来自世界各地的陆地棉样本进行重测序,包括35个地方种,258个改良种、13个优质栽培种以及12个近缘外群品种。
测序平台Illumina HiSeq2500,测序深度5X,PE文库
研究结果
1. 遗传变异检测及群体结构分析
通过对318株陆地棉进行重测序,与参考基因组TM-1比对,共鉴定到8,621,073个SNPs,其中A、D亚基因组特有SNPs分别占31.2%和20.5%。进化树分析结果表明陆地棉栽培种的遗传多样性较低,大多来源于同一祖先。地方种和改良种之间的Fst值仅有0.04,群体分化程度较低。
陆地棉在驯化过程中经历了比较弱的选择作用,在地方种和改良种的基因组中共检测到25个信号较弱的受选择区域,其中只有3个报道过,说明这些基因座可能经历了二次选择作用。
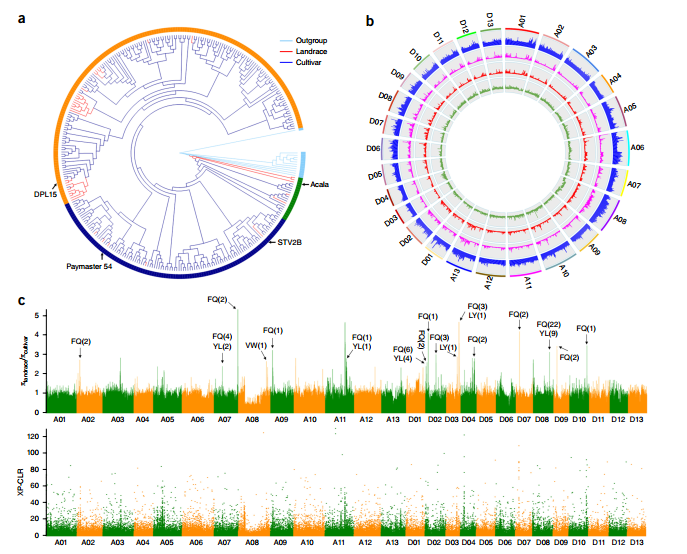
2. 全基因组关联分析
利用1,871,401个高质量SNPs的表型数据进行GWAS分析,共鉴定到119个性状相关位点,其中71个与皮棉产量相关,45个与纤维质量相关,另外3个与黄萎病抗性相关。进一步分析发现皮棉产量和纤维质量相关位点在A亚基因组上的数量要多于D亚基因组,然而,3个与黄萎病抗性相关位点全部位于D亚基因组上。结合GWAS分析和选择清除分析,在4个受选择区域周围发现了控制棉花衣分、纤维长度和强度的基因座。
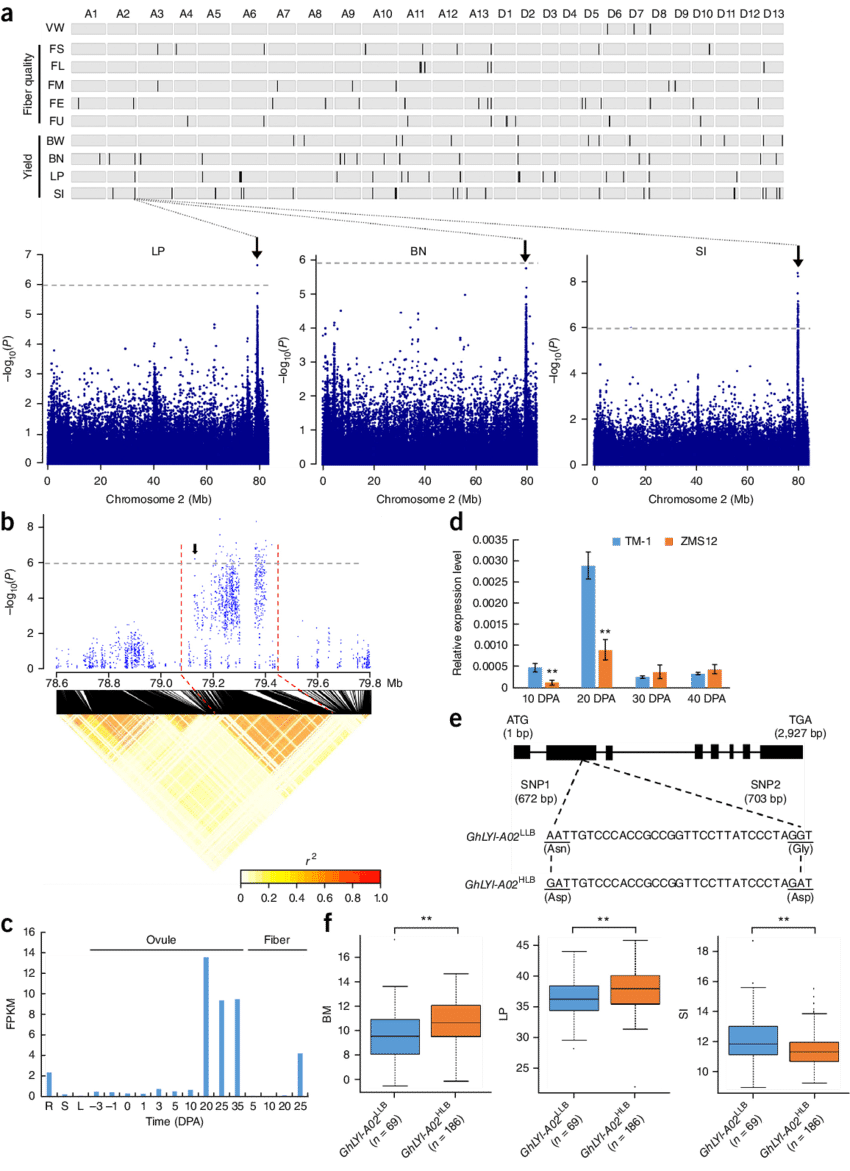
3. 增加皮棉产量的两个候选基因
通过多种分析方法相结合,鉴定到了与多个农艺性状相关联的两个基因座,它们与棉花衣分、株铃数的增加和籽指降低有显著相关性。在位于A02染色体上的基因座附近,鉴定到了候选基因AIL6,该基因与拟南芥ERF型转录调控因子同源,转录组数据表明AIL6在花后20至25天高表达。qRT-PCR分析结果表明AIL6在TM-1中的表达量是在改良种ZMS12中的10倍;另一个是位于D08染色体2.8-3.0Mb区段内的EIL基因,该基因在TM-1和ZMS12中差异表达,qRT-PCR分析也再次验证了该结果。
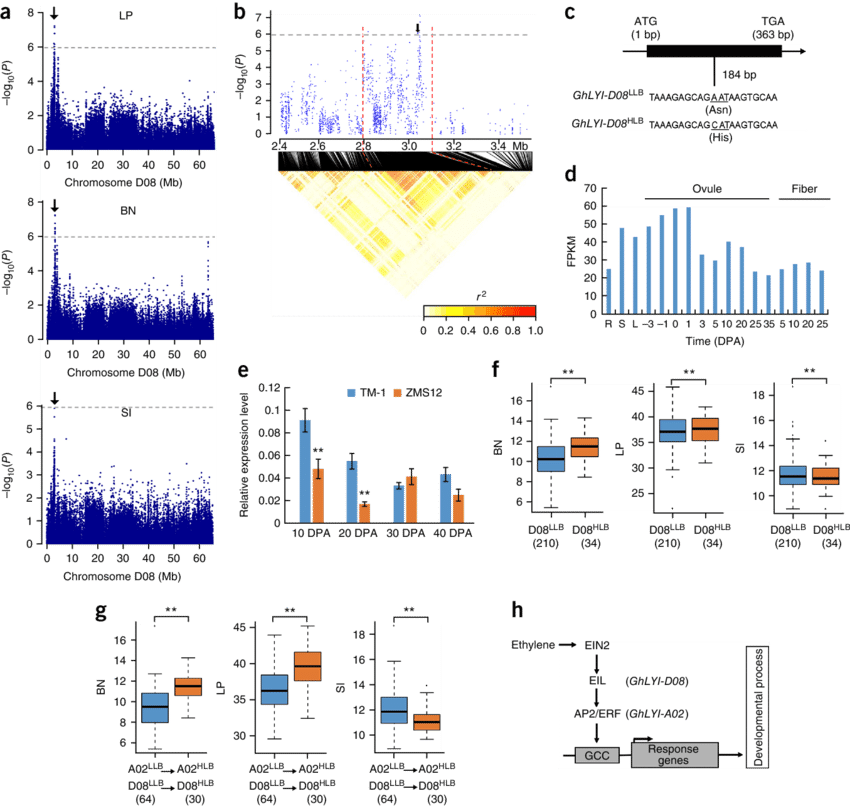
4. 与其他农艺性状相关的候选基因
在75.90-76.05Mb的LD连锁区域内鉴定到一个候选基因——GhXIK,该基因编码XI-K肌球蛋白家族的一个成员,该基因在纤维伸长和次生细胞壁合成过程中高表达。非同义突变导致1,011位的丙氨酸突变为缬氨酸,这与棉纤维长度、纤维强度的增加以及棉纤维的整齐度提高显著相关。在3个与黄萎病抗性相关基因座附近,鉴定到一个编码疾病抗性相关蛋白的候选基因。
结论
该研究将GWAS分析、转录组数据、GBA以及拟南芥直系同源基因的功能注释进行了有效整合,在陆地棉中快速鉴定到多个与重要农艺性状相关的候选基因,并进行了功能验证。这对于陆地棉栽培种的改良以及多倍体作物的进化分析具有重要的遗传学意义。
文章索引
Fang L, Wang Q, Hu Y, et al. Genomic analyses in cotton identify signatures of selection and loci associated with fiber quality and yield traits[J]. Nature Genetics, 2017, 49(7):1089.


