2020-09-03
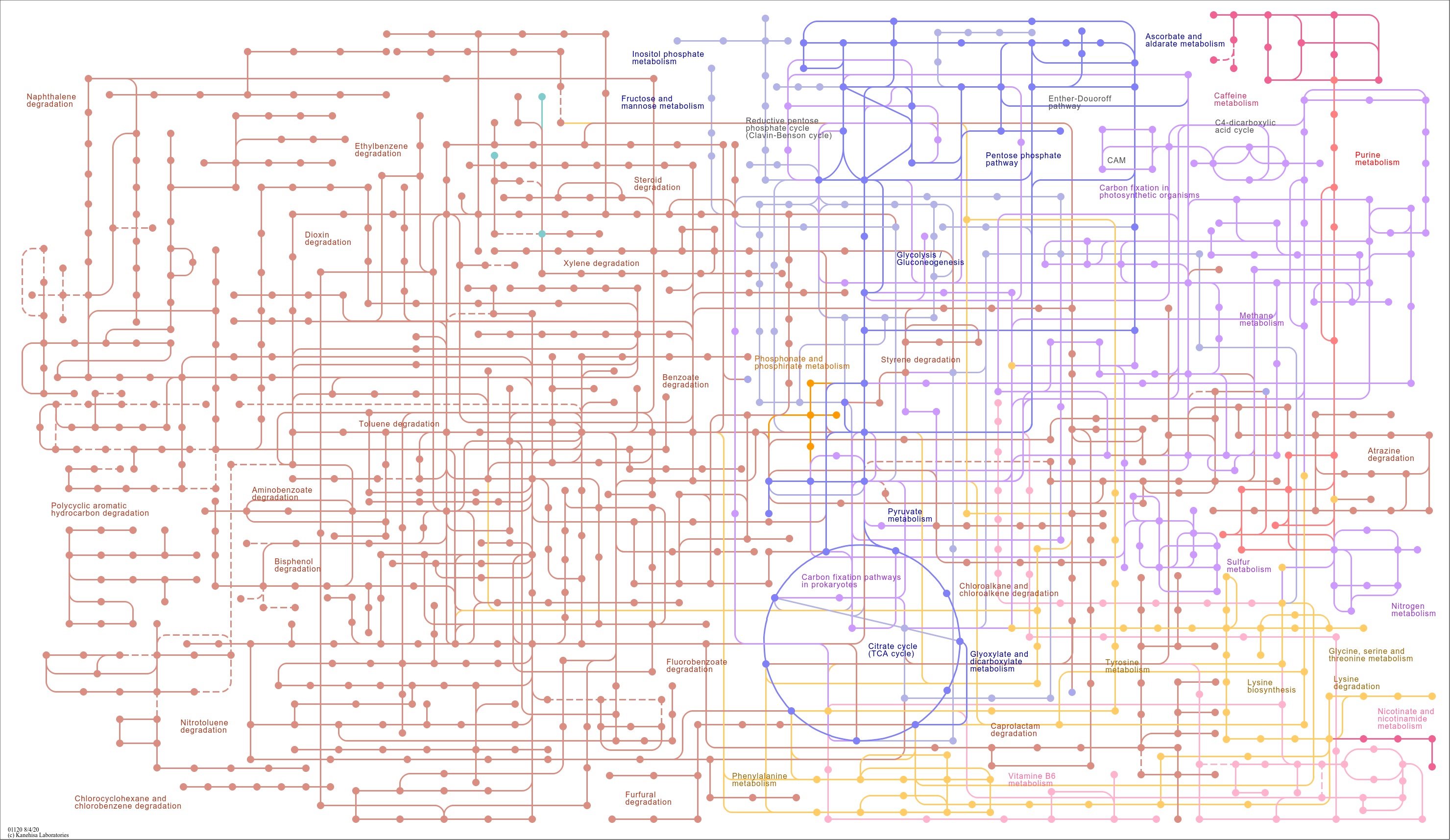
高通量测序技术的发展,不仅让我们对微生物的群落结构有了更全面的了解,更是大大加速了我们对微生物群落功能代谢的研究进程,也让越来越多的研究人员注意到了PICRUSt2等功能潜能预测方法与宏基因组学等研究技术,并在这两大利器的加持下,更深入地挖掘微生物群落代谢功能的相关信息。无论是PICRUSt2功能潜能预测还是宏基因组测序分析,都会得到许多的KO numbers,这可是个好东西!这些KO数据,可以在KEGG数据库中进一步找到它们所涉及的代谢通路图,从而获得更深层的微生物功能特征信息。
KEGG数据库,是一个能系统分析基因功能、并将基因与功能信息相联系的大型知识库,它还将功能通路进行了归类与进一步的等级划分,如下图所示↓↓↓

有意思的是,KEGG还具有强大的作图功能,能利用图形来展示众多的功能途径以及各途径之间错综复杂的相互关系,比如刚才咱们提到的功能通路图!那么,这个功能通路图要怎么做呢?为了方便大家使用,我们特地整理了一套简单的KEGG功能通路图的在线制作流程。
话不多说,首先在浏览器中打开下方链接
https://www.genome.jp/kegg/pathway.html
然后我们就看到下面这个有着花花绿绿Logo的KEGG数据库啦,数据库页面就展示了各种1级通路的信息与部分2级通路的信息。
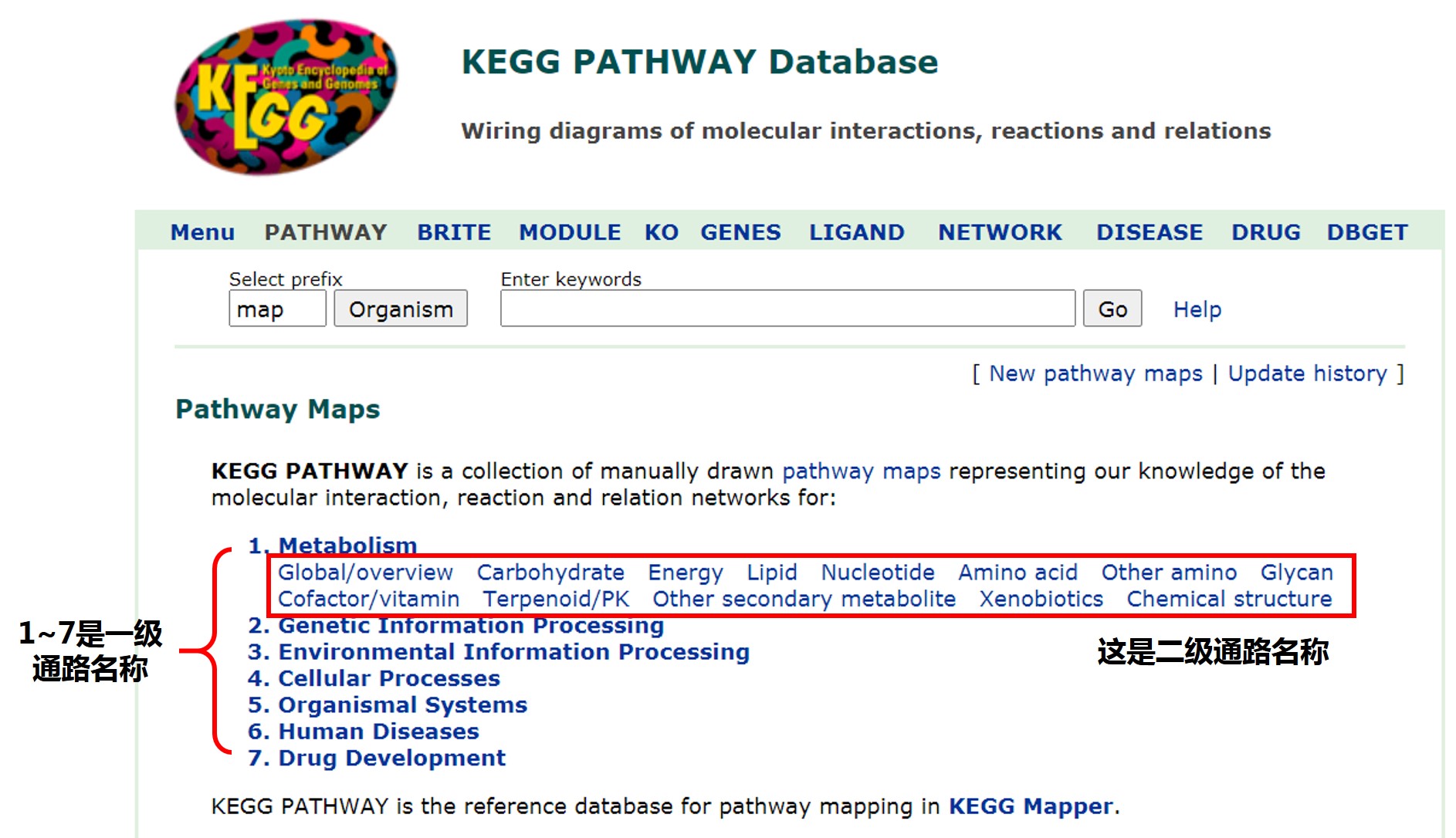
点击需要的1级通路信息,就可以看到相应的2级和3级通路信息,就像下面这样:

然后找找看,有没有您关注的通路信息哦~找到了?好嘞,点击进入!
新页面上,已经自动生成了对应通路的通路图:

以上通路,只是数据库中的参考通路信息,我们还需要把功能通路和我们关注的基因功能信息,结合起来。那么,如果我们需要了解测序分析结果中的KO信息在所关注通路的分布情况,该怎么办呢?别急!下面是绘制方法~
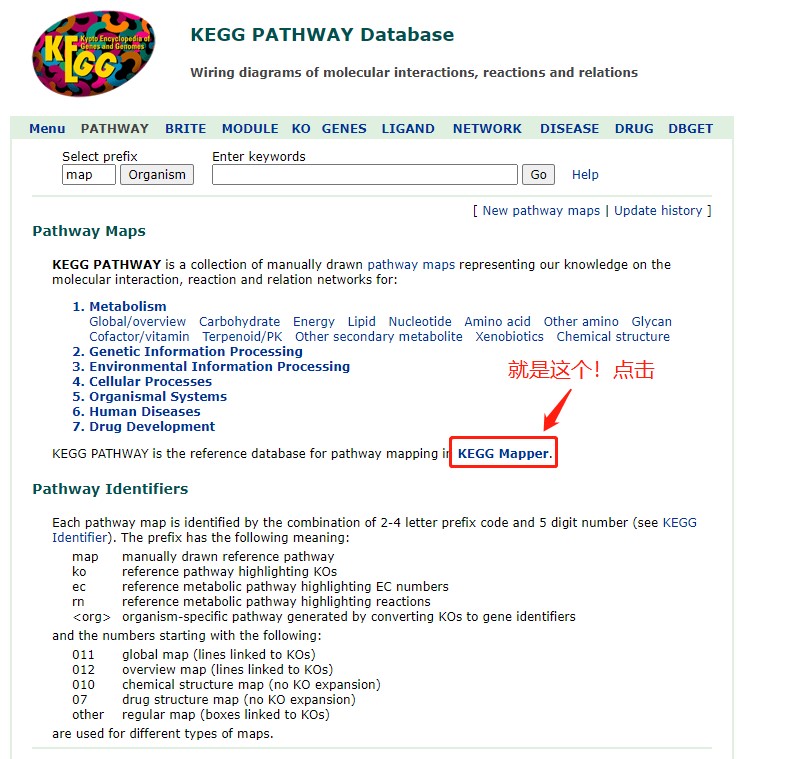
返回到KEGG pathway主页,点击网页中的KEGG Mapper。
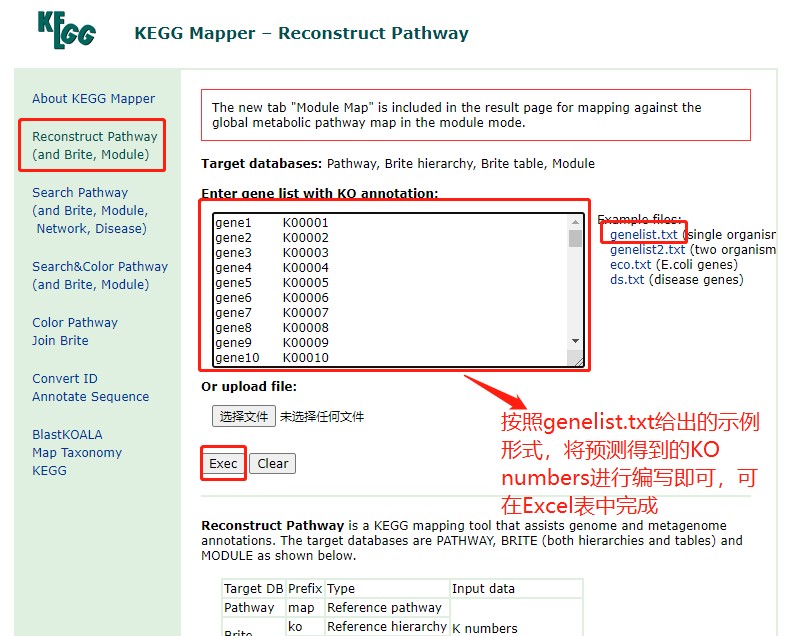
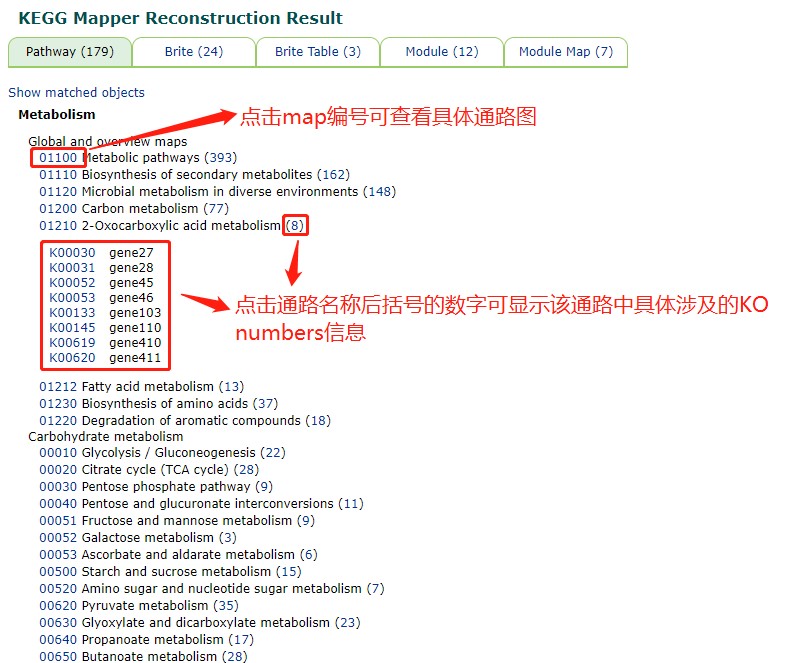
进入网页后,点击左侧目录中的“Reconstruct Pathway”,可进入通路图绘制页面。在对话框“Enter gene list with KO annotation”中需要输入我们预测到的KO numbers的信息(这个信息可以在PICRUSt分析结果C07_picrust文件夹中的predicted_metagenomes_kegg.txt表格里找到;或PICRUSt2分析结果2.7_picrust2\2.7.1_normalized_data文件夹中的KO_pred_metagenome_unstrat_norm.tsv表格里找到),输入格式可参考对话框右侧“genelist.txt”的内容;
就像这样

点击Exec,稍等一会儿,即可进入根据我们提供的KO numbers信息重构后的功能通路界面,选择自己关注的通路进行查看即可:
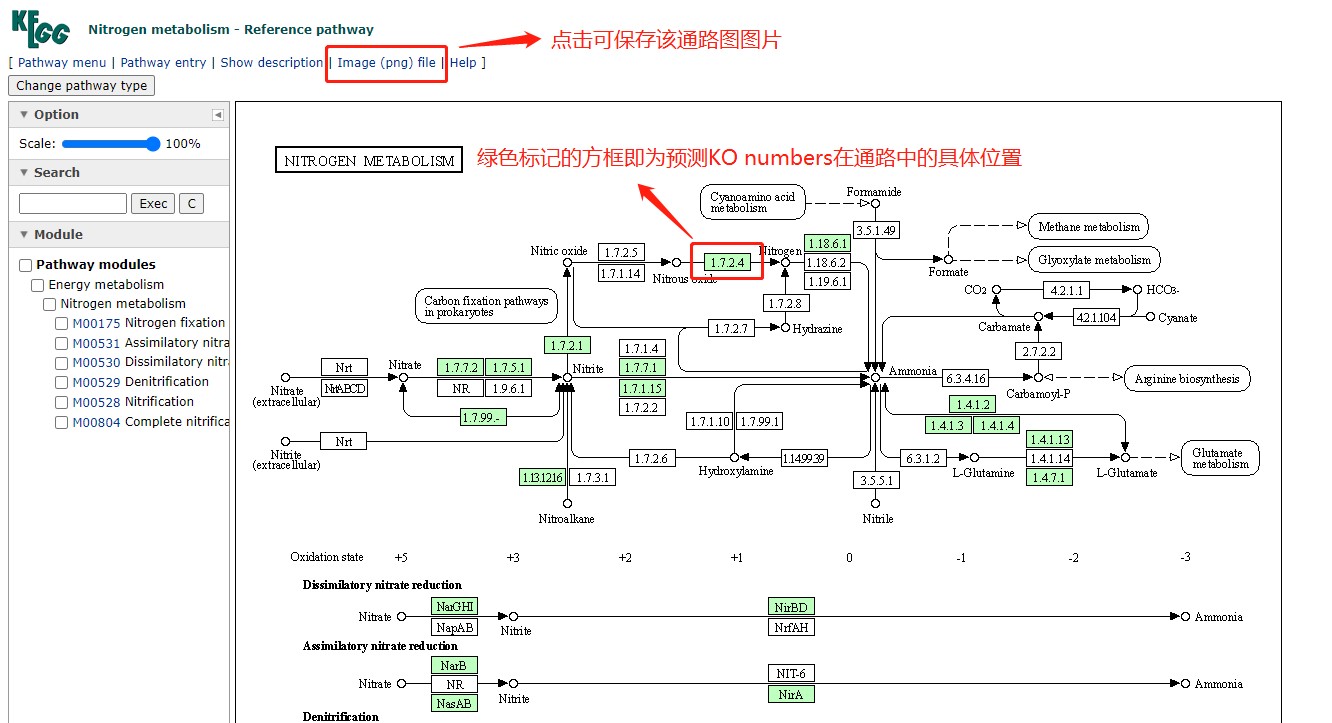
最后,点击Image (png) file即可把查到的代谢通路图保存下来啦~
到这里,整个功能通路图就完成啦,是不是很方便呢?小编偷偷告诉大家,其实KEGG的用途远不止这些,它还可以生成功能通路富集分析的结果、差异比较的结果等等……如果想要了解更多KEGG分析的方法,请不要犹豫,第一时间联系我们派森诺哦,我们将为您提供全方位的服务和解答!


