
2020-09-08
哈喽,又见面啦!BSA项目得到了候选区间,后续如何缩小候选区间,如何对候选基因进行验证呢?
今天小编就选取了几篇典型的BSA文章为大家进行验证方法的总结。

该文献研究的是玉米籽粒大小的性状,该项目bsa选取了极端表型植株共100株(野生型与突变型混池各50株)进行测序,采用△snp-Index进行分析,最终定位区间为7号染色体上0~2M的区间,包含64个基因。通过分析突变类型,找到了8号染色体有一个snp(在Zm00001d018624的第8个外显子上)发现突变,导致提前出现终止密码子,转录水平显著降低。并通过构建突变体的BC1F1群体,对候选区间进行了进一步缩小与确认。
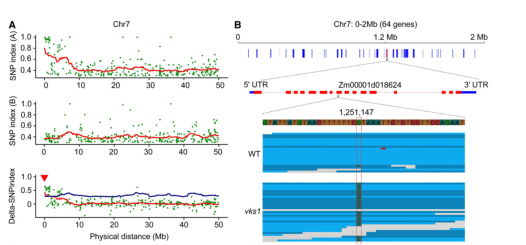
候选基因已经确定了,如何验证呢?本文采用了多种方式对这一基因进行了验证。
验证方法1:突变数据库反向筛选
通过在突变数据库的筛选,找到了2个产生相同性状的突变类型的突变株,将这些基因克隆,其中vks1-2等位基因是由Zm00001d018624第12个内含子的剪接受体位点突变引起的,导致该内含子的RNA剪接缺陷。vks1-3等位基因来自于Zm00001d018624的59未翻译区(UTR)的Mu插入。将vks1-2自花授粉后子代发生性状分离,种子中观察到vks1-1的类似表型。vks1-3种子也远小于野生型(W22),但在纯合vks1-3穗上没有明显的种子大小变异,说明vks1表型部分依赖于遗传背景。为了进行等位性检验,我们在纯合子vks1-1和杂合子vks1-2/+植株间进行杂交。得到的穗与vks1和野生型种子的分离比例接近1:1。
验证方法2:CRISPR/CAS9编辑技术
我们利用CRISPR/CAS9基因编辑技术,产生更多的vks1零等位基因。gRNA被设计用于靶向Zm00001d018624的第8个外显子。得到3个C缺失、T插入和127-bp缺失的独立转基因株系,分别命名为vks1-4、vks1-5和vks1-6。自花授粉的vks1-4/+穗(作为这些CRISPR/ cas9诱导的无突变体的代表)也分离出了不同大小的小种子,如图invks1-1和vks1-2所示。因此,Zm00001d018624是vks1表型的基因。

本文中,作者结合传统基因定位方法和BSA法,利用155个F6代重组自交系个体,定位了IL52的霜霉病与白粉病抗性候选基因,并揭示了两者之间的连锁关系。
从155个RILS中选取16株抗病,20株感病材料混池测序后,做BSA定位分析,结果定位到5号染色体23.21-25.70 Mb的2.49M区间内,区间内包含27个△SNPindex = 1的SNP位点,其中非同义突变位点3个,3’UTR位点1个,分布在4个基因上。
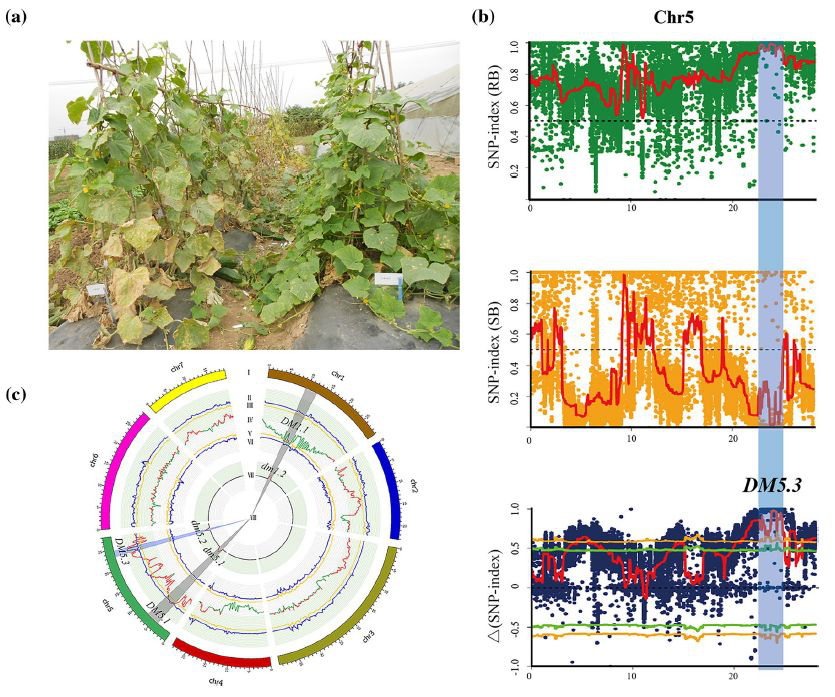
通过连锁图谱和RILS表型数据,PM基因被定位在InDel73 (23.68 Mb) 和InDel82 (24.94 Mb)之间,与BSA结果相符。后基于BSA结果开发的Indel、SNP、SSR分子标记与193个F2个体表型综合分析,pm基因被定位到17ID185和17ID211之间, 遗传距离 0.5 cM,物理距离468 kb (24,550,703-25,018,946 bp),区间内包含3个基因。同时发现基因Csa5M622830.1的3’UTR上的 SNP6,与PM完全连锁。

该文献BSA定位未取得较好的区间定位结果,但却为我们提供了一种新的候选基因确定的思路。
通过SNP和indel call,在MF-和MSbulk之间确定了2,594,935个SNP和405,428个indel。为了绘制雄性不育图谱,应用ED算法测量等位基因分离,并根据两大群体间的snp和indels确定连接基因组位点。通常,ED在统计上显著的峰值被认为是一个候选范围。然而,在我们的研究中,12条染色体都没有发现明显的峰。为了克服上述问题,我们采用了更严格的过滤,规则如下:(1)ED值必须大于0.5;(2) SNP必须引起非同义突变;(3) indel必须导致移码突变。最后,9个snp和4个indels通过筛选,发现它们位于6条染色体上的12个注释基因上,被认为是mcs-1位点的候选基因。
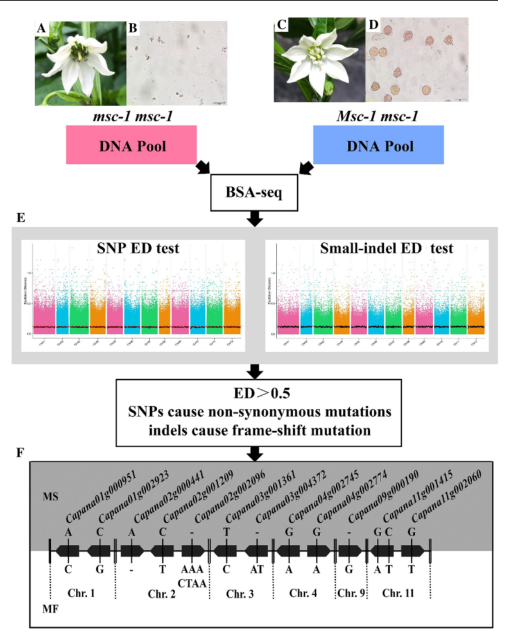

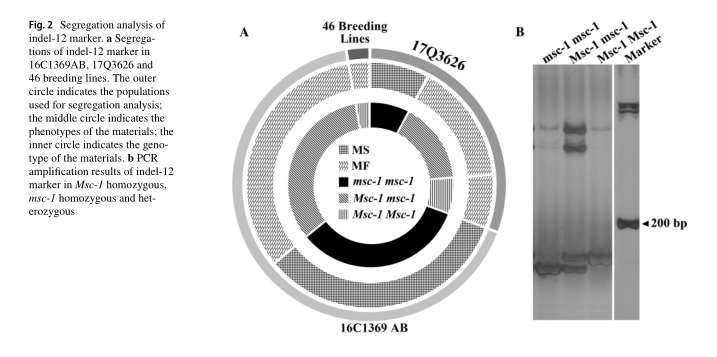
候选基因选定了,该文献老师采用的是根据已有文献报道的相关基因,锁定了最终的候选基因,为了验证这一基因,老师开发了indel标记,利用两个群体NILs群体验证了indel-12标记与雄性不育性状的共混关系,结果如下图所示,表明在验证群体中,msc-1基因组呈现完全的共分离现象,表明Capana02g002096可能是msc-1基因座的强大候选基因。
篇幅有限,小编就不多赘述了,总结下来。
当候选区间较大时,缩小候选区间的方法如下:
1、构建遗传图谱,结合表型数据,采用QTL分析进行定位,一般选取与BSA分析的交集作为候选区间;
2、开发分子标记,通过验证群体性状与分子标记的一致性缩小候选区间;
3、结合已经报道的性状相关基因或者相关突变体库,锁定候选基因;
4、结合转录组的相关基因表达水平分析选取具有明显表达水平差异的基因作为候选基因。
候选基因主要的验证方法有:
1、将候选SNPs转化为CAPs或者indel等标记进行验证;
2、基于转录组数据的差异表达分析,看基因是否有显著表达差异;
3、功能缺失型实验验证,如CRISPR、基因敲除,过表达等;
4、RT-PCR验证候选基因的表达。
好啦,BSA定位后续分析方法总结就到这啦,不知道老师们有没有学会呢?其实从以上文章大家应该也能发现,BSA项目中,定位得到候选区间只是故事的开始,候选基因的确定及基因功能的验证才是文章画龙点睛的那一笔,研究者们每一篇文章的背后,都是固有思维与大开脑洞的碰撞,也许这就是科学的魅力吧!
参考文献:
1. Yongcai Huang, Haihai Wang, Xing Huang et al. Maize VKS1 Regulates Mitosis and Cytokinesis During Early Endosperm Development. Plant Cell. 2019 , 31(6):1238-1256. doi: 10.1105/tpc.18.00966.
2. Kaijing Zhang, Xing Wang, Wenwei Zhu et al. Complete resistance to powdery mildew and partial resistance to downy mildew in a Cucumis hystrix introgression line of cucumber were controlled by a co-localized locus. Theor Appl Genet. 2018 , 131(10):2229-2243.doi: 10.1007/s00122-018-3150-2
3. Qing Cheng, Peng Wang, Jinqiu Liu et al. Identification of candidate genes underlying genic male-sterile msc-1 locus via genome resequencing in Capsicum annuum L. Theor Appl Genet, 2018, 131(9):1861-1872. doi: 10.1007/s00122-018-3119-1.