2020-09-15

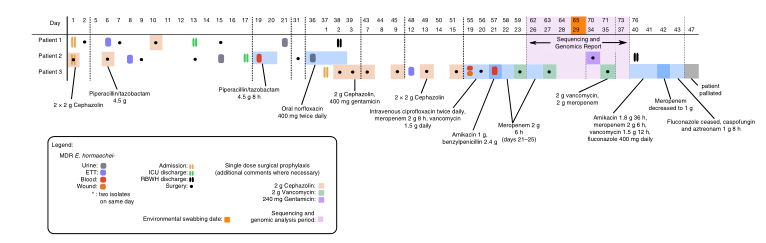
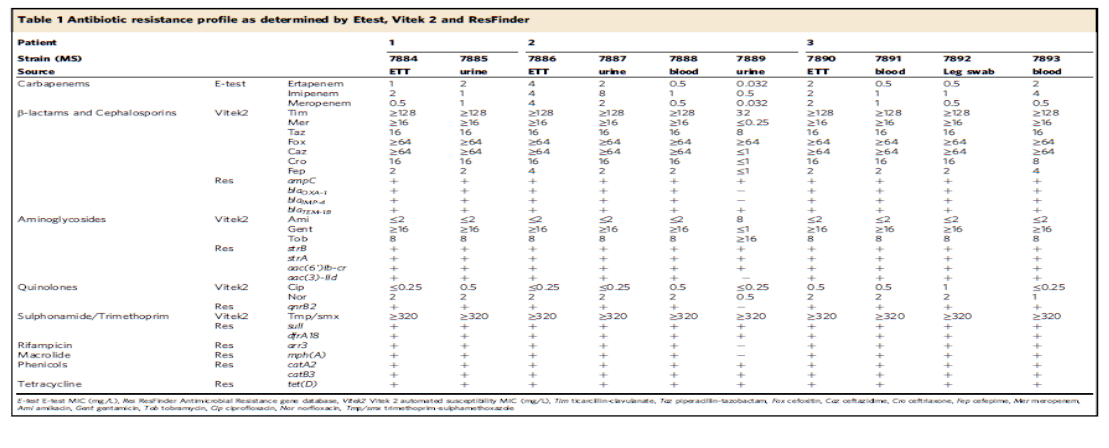
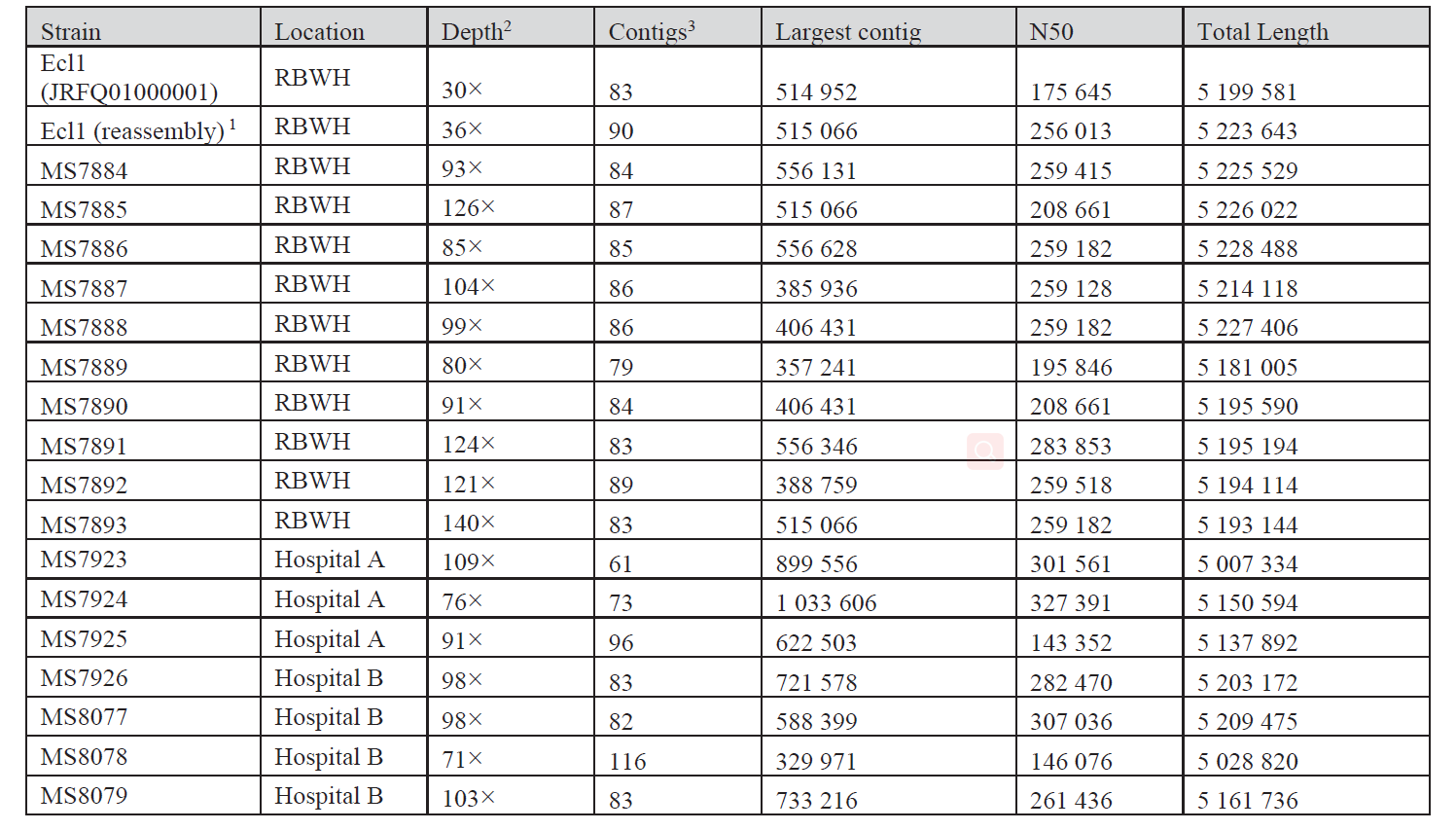
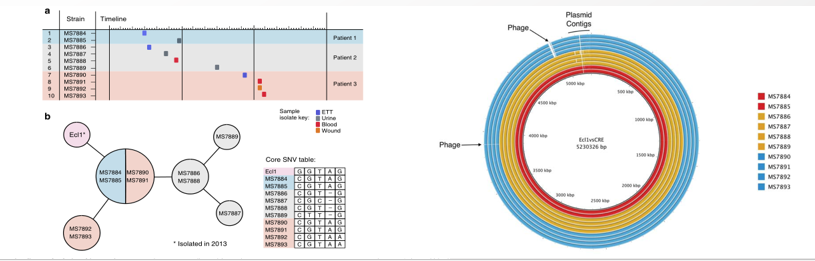
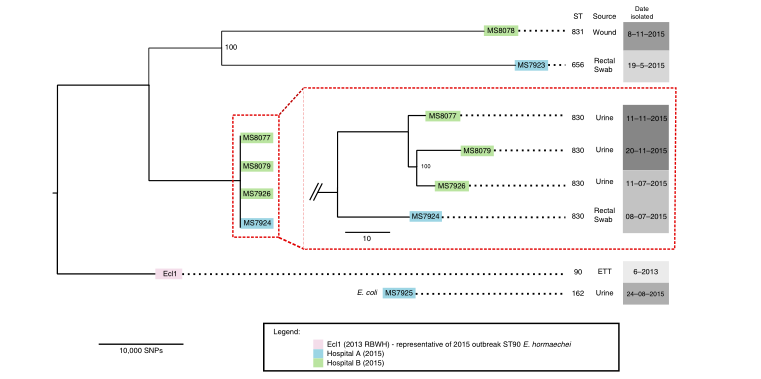
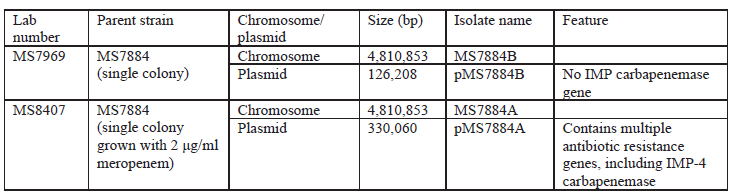
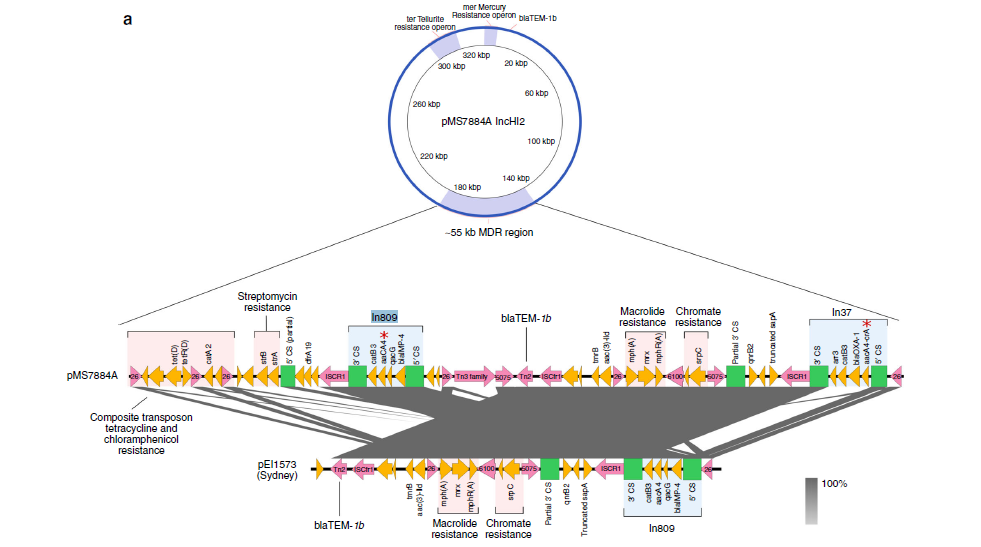
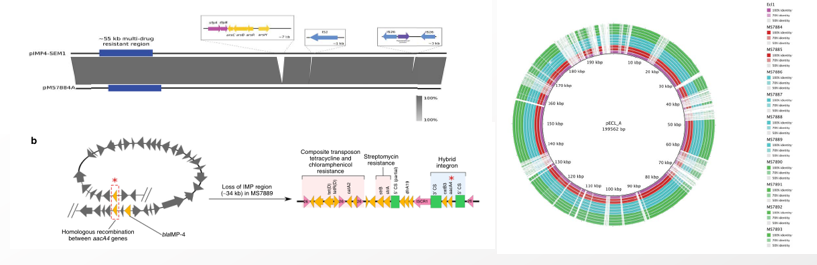
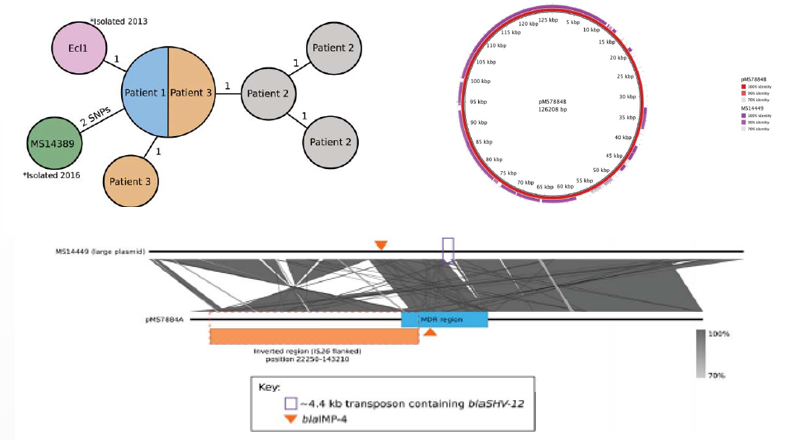
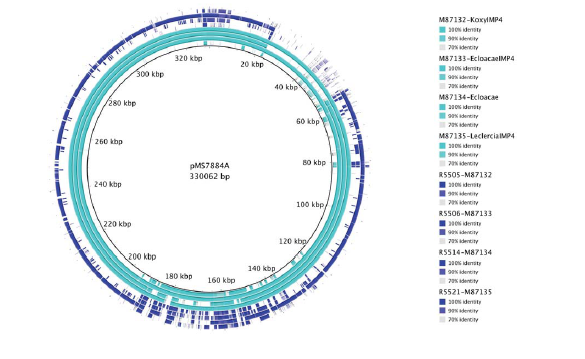
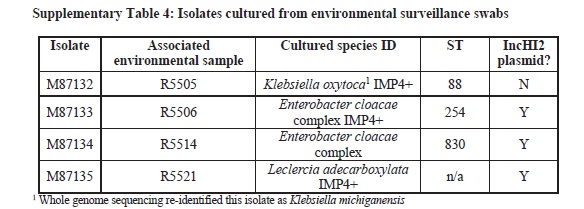
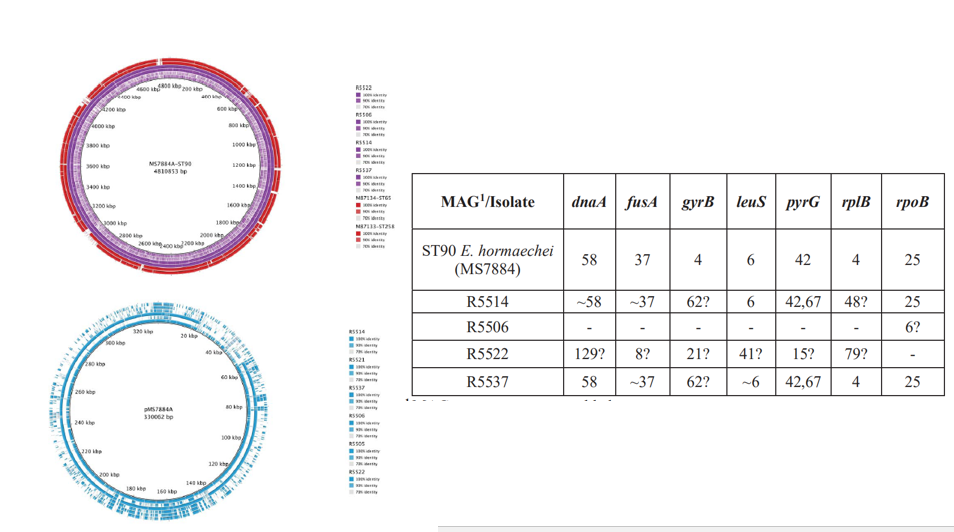
2020年1月《nature communications》期刊发表了一篇题为“Integrating multiple genomic technologies to investigate an outbreak of carbapenemase-producing Enterobacter hormaechei”的文章,该文章整合了多个平台的测序技术(Illumina、PacBio、Nanopore)调查产碳青霉烯肠杆菌的疫情爆发情况,为疫情提供了深入分析。 研究背景 碳青霉烯抗生素已成为治疗多药耐药(MDR)革兰氏阴性菌引起的严重感染的主要药物,该抗生素使用量的增加已导致碳青霉烯类药物产生耐药性,以及产碳青霉烯酶的肠杆菌科(CPE)和耐碳青霉烯酶的肠杆菌科(CRE)出现,其中包括常见的肠菌,如大肠杆菌、肺炎克雷伯菌和肠杆菌属。 2005年以前,99.9%的肠杆菌对碳青霉烯敏感。然而,随着CRE分离株的增加,现在世界卫生组织所有卫生区域都报道了该类微生物。据报道,CRE感染的死亡率高达48%,对用粘菌素代替碳青霉烯类的最后一线抗生素也出现了耐药性。 肠杆菌科细菌对碳青霉烯类耐药是通过一系列机制发生的,最令人关注的是碳青霉烯酶编码基因的获得,最常见的是通过移动遗传元件(MGEs)的转移,如质粒,偶尔携带多种与其他耐药决定因素共位的多个β-内酰胺酶,使这些菌株呈MDR或广泛耐药性(XDR)。 在过去的十年里,全基因组测序(WGS)变得更容易获得和负担得起,导致其在许多领域的应用增加,包括临床微生物学。使用WGS来表征临床相关细菌的主要特征之一是它能够在单个核苷酸的分辨率上提供菌株的亲缘关系。WGS已被用于了解医院环境下的传播情况,而不仅仅是使用传统的诊断方法来确定传播情况。然而这些研究中很多是回顾性的,并且主要是采用单一测序技术进行进行的,鲜少采取多种测序技术来研究疫情的传播情况。 材料与方法 1、实验材料与方法 (1)从3位烧伤患者的不同部位采取10份样本,周边医院取7份blaIMP-4阳性的菌株、进行基因组测序与传统培养; (2)选择ICU病房中收集到50个拭子和水的样本以及烧伤病房等疫情爆发无关的的环境样本进行宏基因组测序与传统培养。 2、测序及组装方法 (1)二代测序平台:Illumina Hiseq、NextSeq、MiSeq; (2)三代测序平台:Pacbio、Nanopore MinION (3)组装方法:Spades(v3.6.0和v3.11.1)、Canu (v1.3)、SMRT Analysis suite (version 2.3.0); 3、比较基因组分析 基因核心基因组SNP进化树构建;MLST分型;质粒分型;ANI分析;比较基因组圈图。 实验结果与分析 1、临床实验 2015年年中,RBWH ICU收治了3名烧伤患者。患者1和2在同一天入院,入院后,两名患者均出现耐碳青霉烯感染。患者1取两份样本(气管插管和尿液),患者2取四份样本(气管插管、尿液和血)。患者3于患者1、2出院37天后入院,患者3也因对碳青霉烯耐药的阴沟肠杆菌感染而感染,并从气管插管、血液和伤口部位提取了四份样本。经过抗生素和抗真菌治疗,患者在入ICU的第47天得到缓解。在确认三位患者感染了产blaIMP-4的Enterobacter hormaechei后,对所有10个分离株进行了测序和基因组学分析。 对得到的分离株进行药敏实验,发现除MS7889(在第36天从患者2的尿液中分离)外,其他分离株对头孢曲松、头孢他啶、替卡西林-克拉维酸、哌拉西林-他唑巴坦、美罗培南、庆大霉素和磺胺甲恶唑均耐药,MS7889则对碳青霉烯类完全敏感。 2、Illumina、Pacbio、Nanopore测序 对10个分离株进行WGS测序,结果表明所有的分离株都为ST90类型、都含有IncHI2质粒,大多数都有相同的抗性基因,包括100%相同的blaIMP-4基因(MS7889除外)。与数据库进行比对发现,分离株与2013年从被火烧伤的病人身上分离的ST90型菌株Ecl1近缘,并且表现出相同的耐药基因图谱。 以Ecl1为参考基因组进行SNP分析,发现RBWH分离株的差异小于5个核心SNP,这与祖先的进化关系一致。其中患者3缺失了一个原噬菌体,但是患者1的两个分离株和患者3的两个分离株在核心基因组水平上无法区分。Ecl1与这些分离株非常密切相关,不同的只有一个核心SNP。来自患者2的所有四个分离株均含有特定的SNP缺失,排除了患者2至患者3的传播。 为了确定周围医院产IMP肠杆菌的情况,对7个产blaIMP-4的分离株进行测序,MLST和SNP分析没有发现其与Ecl1之间有联系(与Ecl1亲缘关系最近的菌株也有50000个SNP)。虽然不是克隆株,但所有肠杆菌科分离株都具有非常相似的抗生素抗性基因谱,这可能通过MGEs(例如整合子和/或质粒)进行横向基因转移。WGS分析显示,本研究中的所有18个大肠杆菌分离株,都含有一个IncHI2质粒和一个相同的BLAIMP-4基因,这强烈表明医院肠杆菌科之间BLAIMP-4的质粒介导循环。 对具有代表性的MS7884分离物进行Pacbio测序,得到了4.8M左右的染色体和两个质粒。质粒pMS7884A长330,060bp,携带多个抗性操纵子以及一个~55kb的MDR区域。MDR区域被发现含有两个1类整合子(In809,In37)以及其他几个抗生素耐药基因。将这一多药耐药区域与公开的基因组进行比较,发现与2012年从澳大利亚悉尼的阴沟肠杆菌分离出的pEl1573非常相似。而pMS788B为不可分型质粒,大小为126,208bp,不含有抗生素抗性基因。 除MS7889外,Illumina测序的另外17个分离株中都存在相同的质粒;由于两个几乎相同的氨基糖苷类耐药基因之间的同源重组,MDR质粒(包括blaIMP-4)中丢失了约34 kb区域。值得注意的是,在15%的病例中,MS7884在美罗培南缺陷培养基中,会导致BlaIMp-4基因或整个质粒的丢失。 为了更好的了解MS14449与2015年的肠杆菌分离株之间的关系构建进化树,根据进化树来MS14449不属于ST90分型(后续Illumina测序发现该菌株属于新的ST175分型)。MS14449测序后与pMS7884A进行核苷酸比对,表明MS14449可能存在一个高度相似的质粒,但是pMS7884A与MS14449相比可能存在部分MDR基因的缺失。 3、宏基因组测序 使用传统的培养方法对医院环境进行检测,并未发现ST90型环境来源的肠杆菌。2018年5月,从ICU病房中收集到50个拭子和水的样本以及烧伤病房等疫情爆发无关的的环境样本进行测序和传统培养。传统培养检测到Klebsiella oxytoca, E. cloacae complex 和Leclercia adecarboxylata,Illumina测序确定这些分离株与疫情无关,但是这4株分离株中有3株上含有blaIMP-4基因,进一步比对发现其对应的IncHI2质粒与pMS7884A高度一致。 宏基因组组装结果与参考基因组Enterobacter hormaechei在染色体和质粒水平上有很高的相似性,ANI平均在93%-97%之间。MLST分析也能检测到一个几乎相同的等位基因ST90,抗性基因筛查也发现了blaIMP-4基因,和IncHI2型质粒的存在。 结论与讨论 1、通过Illumina测序,发现所有的产碳青霉烯的肠杆菌分离株序列都为ST90,且几乎相同;与公开可获得的数据进行比较,所有爆发的分离株都可以与该病在2013年一株分离株相联系起来,表明该医院存在环境来源; 2、使用Pacbio测序,发现了不同医院产IMP-4菌株中的IncHI2质粒上blaIMP-4基因的完整背景; 3、对医院管道中的样本进行宏基因组测序发现了ST90型肠杆菌和IncHI2质粒; 4、Nanopore测序结合Illumina测序迅速解决了后续分离物与最初爆发的真实关系。 虽然在WGS的成本和可用性方面有了显著改善,但将这些进展纳入常规临床微生物学仍是一个挑战。这几项研究表明,WGS能够在菌株之间提供最佳区分,从而有助于告知对暴发或医院内感染的反应。在此,本文证明,综合WGS方法可以帮助快速确定危重护理环境中的疫情,特别是传播途径。 文章索引:Roberts L W , Harris P N A , Forde B M , et al. Integrating multiple genomic technologies to investigate an outbreak of carbapenemase-producing Enterobacter hormaechei[J]. Nature Communications, 2020, 11(1). 点击阅读全文:https://www.nature.com/articles/s41467-019-14139-5#citeas