
2020-10-30
物种注释的本质是与参考序列数据库进行比对,利用identity、score、coverage、e-value等多项指标,对比对结果进行综合打分,根据相应的注释算法进行最后判定的过程。因此,注释结果的全面性和准确性不仅与参考数据库的选择有关,注释算法的应用也是很重要的一环。

此前,我们已经为大家详细介绍过QIIME 2分析流程的诸多优点(《【问答集锦】关于QIIME 2的一切,您想知道的都在这儿了》、《【派森诺微生物组】乘风破浪的QIIME2基因云,又放大招啦!!!》)(点击查看),而物种注释正是QIIME 2众多分析优点中的重要一环!今天就为大家带来QIIME 2中两种常用的注释方法的介绍:classify-sklearn算法(Bokulich et al., 2018,https://github.com/QIIME2/q2-feature-classifier)和brocc算法(Nilsson et al.,2006,https://github.com/kylebittinger/q2-brocc#the-brocc-algorithm)。
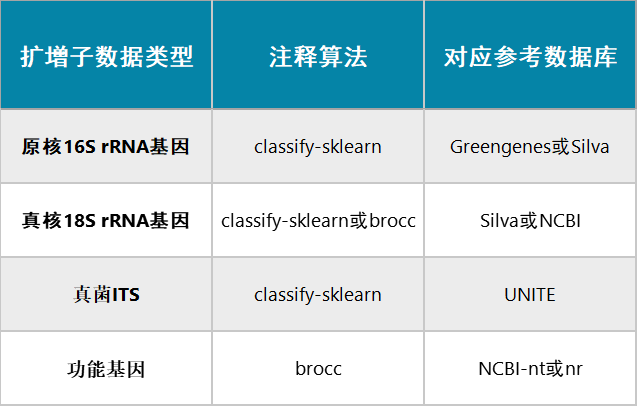
QIIME 2分析流程的常用注释算法和对应数据库
首先,大家可能会好奇:物种注释时,为什么需要考虑注释算法呢?简单而言,由于测序序列长度的限制,以及微生物种类的多样性,导致容易出现“一对多”(一条序列对应多个潜在物种注释结果)的现象,如果没有合理的注释方法,就会出现“选择困难症”。因此,在分析时,就需要对注释结果进行必要的取舍,既保证注释结果的可靠可信,又尽可能不损失物种注释的精度(保证尽可能还原物种的精细组成)。因此,注释算法也就应运而生啦~
classify-sklearn算法
QIIME 2分析流程的物种注释插件q2-feature-classifier中包含三种不同的分类方法。其中,classify-consensus-blast和classify-consensus-vsearch都是基于序列对齐的方法,可以在比对结果的top hits找到合适的注释信息,不需要预先训练。但是对于特定类型的群落样本和测序参数(包括用于扩增的引物和序列读长等信息),注释所使用的分类器在进行数据训练后,表现将优于上述基于序列对齐的方法。QIIME 2中,基于数据训练的机器学习分类器可通过classify-sklearn获得,理论上可以将scikit-learn中包含的任何分类方法都应用于各类微生物组扩增子测序数据的分类注释,具有较强的针对性,并体现出以下两大优点:
01、使用Naive Bayes分类器的classify-sklearn算法在16S rRNA基因和真菌ITS序列物种注释的精确度和严谨性方面优于其他的标准分类方法,可以最大程度上确保注释结果的可靠、准确。通过建立基于人工群落(mock community)、交叉验证(cross-validated)和新物种检出(novel taxa evaluations)的三维评价模型,可以发现classify-sklearn对于16S rRNA基因和真菌ITS序列在属水平和种水平上的注释结果更加精确和严谨。

Fig. 1 Classification accuracy comparison between mock community, cross-validated, and novel taxa evaluations. Scatterplots show mean F-measure scores for each method configuration, averaged across all samples, for classification of 16S rRNA genes at genus level (a) and species level (b), and fungal ITS sequences at genus level (c) and species level (d).
02、在分类器的运行性能方面,随着目标序列数的增加,注释分析的运算时间优于Vsearch、BLAST等方法;而当参考序列数据库增大时,注释分析的运算时间又优于RDP、SortMeRnA等方法。综合比较,Naive Bayes分类器在运行稳定性和速度上,更优于其它常见分类器。
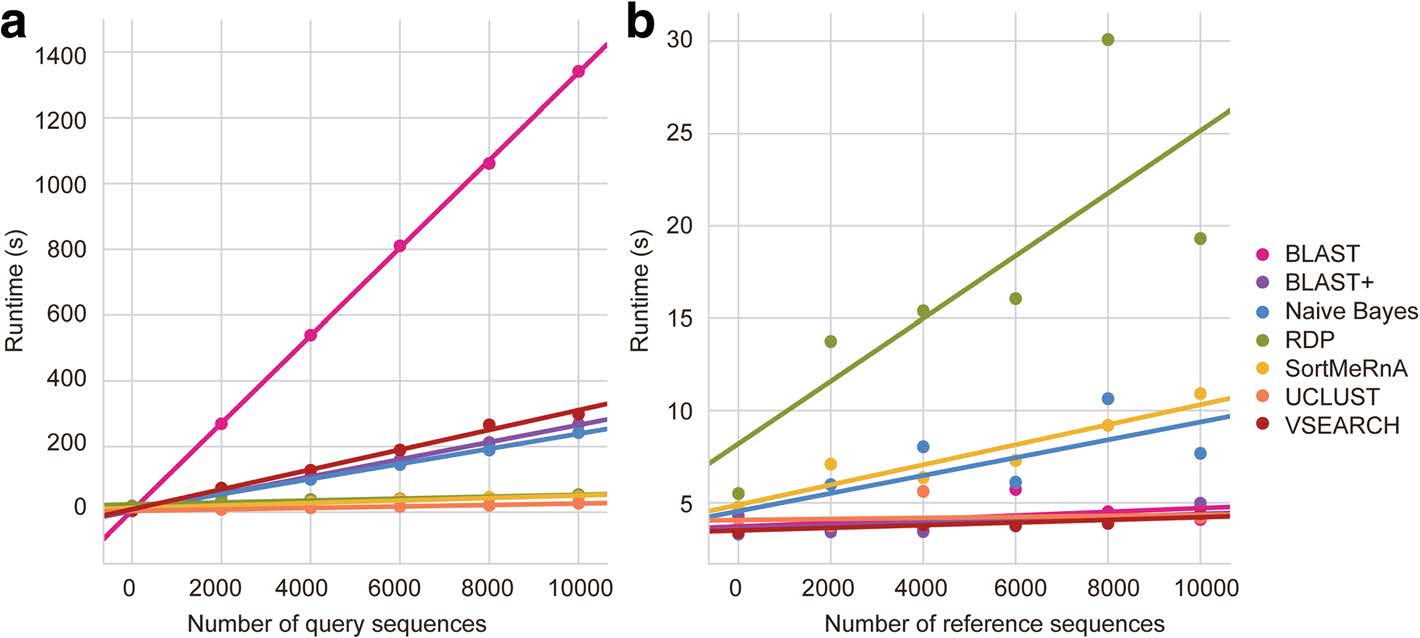
Fig. 2 Runtime performance comparison of taxonomy classifiers. Runtime (s) for each taxonomy classifier either varying the number of query sequences and keeping a constant 10,000 reference sequences (a) or varying the number of reference sequences and keeping a constant 1 query sequence (b).
brocc注释算法
相对于基于机器学习的classify-sklearn算法,QIIME 2分析流程中的brocc注释算法则更适用于选择nt或nr数据库进行注释的测序数据集,比如诸多功能基因测序的结果。首先,brocc将测序得到的序列与nt或nr数据库进行比对,利用identity、score、coverage、e-value等多项指标,对比对结果进行综合打分,选择综合得分前5的注释结果(top 5),进而运用brocc注释算法,追溯五条注释结果的共同祖先,作为此序列最终的注释结果,同时可以通过调整算法阈值(取值范围为0-100%),来调整注释结果:
Brocc注释算法结果展示表
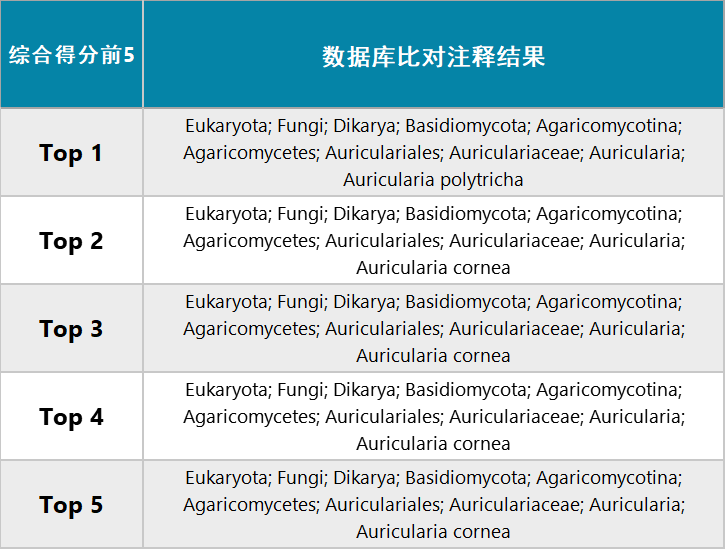
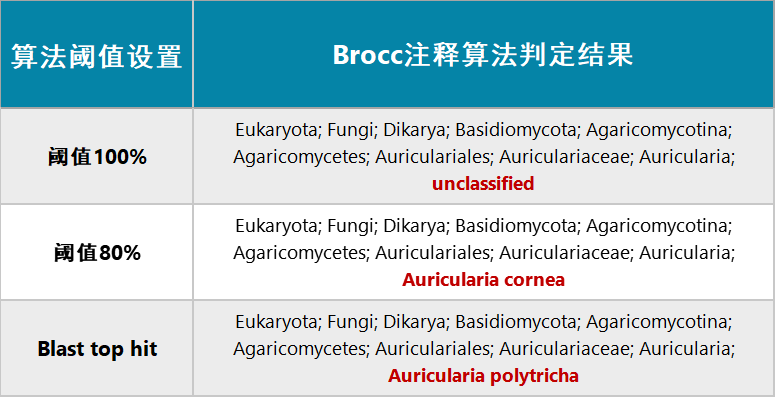
由此可见,在运用brocc注释算法时,通过对物种分类信息进行投票和控制,即对阈值进行调整,从而可以获取更严谨的物种注释结果:当设置算法阈值为100%时,5条注释结果追溯共同祖先,最终注释结果精确到属水平,而种水平上给出的判定结果是“unclassified”。这也解释了在注释得到的“各分类水平物种注释表(L1-L7)”中,通常会出现某等级注释结果为unclassified的情形:并不是未注释到物种信息,而是为了给出更加准确和严谨的注释结果,brocc通过投票和控制机制,对注释结果进行了修正。在这个示例中,当设置算法阈值为80%时,5条注释结果中有4条的注释结果是一致的,这时注释结果可以精确到种水平。
传统BLAST比对注释的问题
与brocc的投票控制方法相比,只依据BLAST比对结果进行物种注释,方法上就“简单粗暴”许多了:这种情况下,通常都是以top hit(比对后会产生多条比对结果,top hit是得分最高、e值最小的一条)为目标序列的比对结果,即使目标序列和其它参考序列也完全一致,也不再考虑其它候选参考物种,用这样的方法容易获得“好看”的结果,但更可能得到的是一个“不真实”的结果。尤其是对于功能基因测序而言,由于功能基因片段在不同物种之间、甚至在同一个菌属之内就会发生变异,序列未必100%相同,如果只依据BLAST对短片段序列的比对结果进行物种注释,那就有很大可能出现“假阳性”的注释结果,无法反映群落的真实特征。
结 语
今天的分享就到这里啦~物种注释也是一门技术活,简单粗暴的BLAST并不可取,QIIME 2的classify-sklearn+brocc才是好!派森诺已经自研构建了完整的QIIME 2注释分析流程,为您带来QIIME 2分析体验!大家感兴趣的话,记得登录派森诺基因云(https://www.genescloud.cn/)尝鲜哦!
参考文献:
[1] Bokulich, N. A. , Kaehler, B. D. , Ram, R. J. , Matthew, D. , Evan, B. , & Rob, K. , et al. (2018). Optimizing taxonomic classification of marker-gene amplicon sequences with qiime 2’s q2-feature-classifier plugin. Microbiome, 6(1), 90.
[2] Nilsson, R. H. , Ryberg, M. , Kristiansson, E. , Abarenkov, K. , Larsson, K. H. , & Urmas Kõljalg. (2006). Taxonomic reliability of DNA sequences in public sequence databases: a fungal perspective. PLOS ONE, 1.