2020-11-18
不少初次接触转录组测序的小伙伴,在拿到一份分析结果,并且兴致冲冲地打开差异表达分析的表格后,却被一连串陌生的名词浇灭了科研的热情:foldchange是什么?为什么还有个log2foldchange?Pvalue我认识,那padj又是什么?重要的是,这个basemean到底该怎么理解?报告里说:basemean是DESeq软件对一个组内所有生物学重复的基因表达量做“均一化”结果,可是我拿三个重复的fpkm算了平均数也对不上啊?
莫慌!我们这就用大家都耳熟能详的老朋友——Excel,来演示一下DESeq软件对basemean的计算过程,详细大家在看完这篇软文之后会对basemean有一个清晰的认知。
(温馨提示:文末有“太长不看总结版”)
假设我们获得了一张基因表达量表格,该表格中共有A1-A3,B1-B3共6个样品,1-11编号的11个基因,基因表达量以read count值表示:
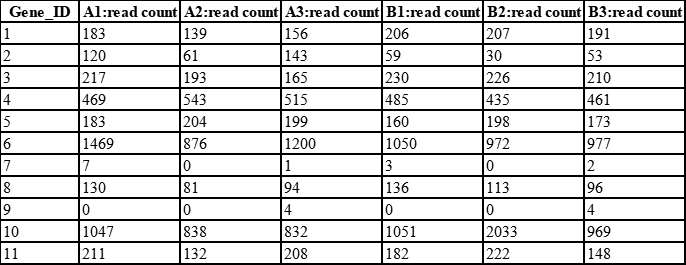
1、、对所有read count值取对数,0值仍记为0:
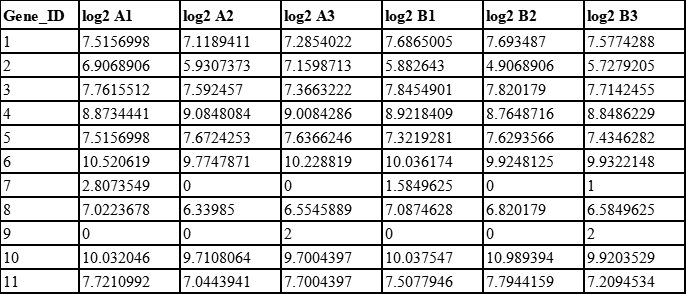
2、计算每个基因在6个样品中的表达量均值(横向计算)
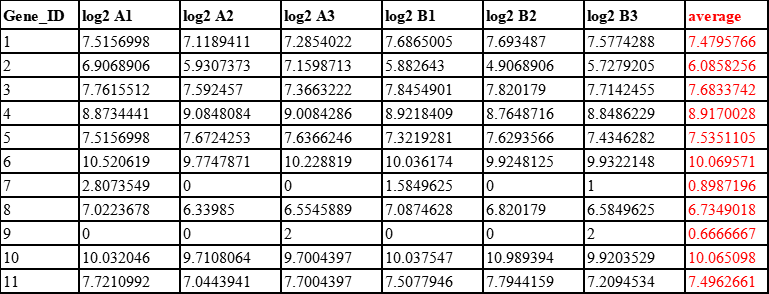
3、用每个基因的表达量减去对应的均值,即2-7列的数据依次减去第8列的数据
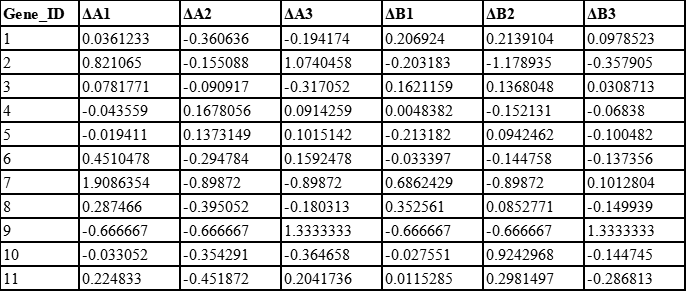
4、按样品取中位数
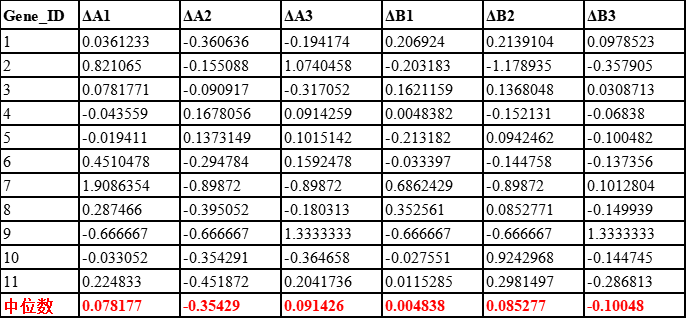
5、因为计算的第一步取了对数,所以现在将中位数利用指数函数转换回去,记为sizefactor
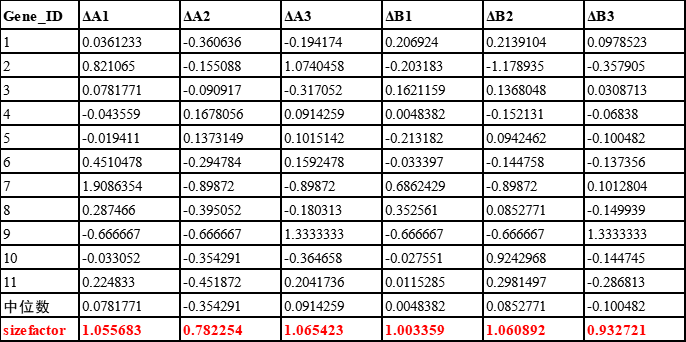
6、回到原始的read count表格,用每一个数值除以对应样品的sizefactor,得到均一化的表达量
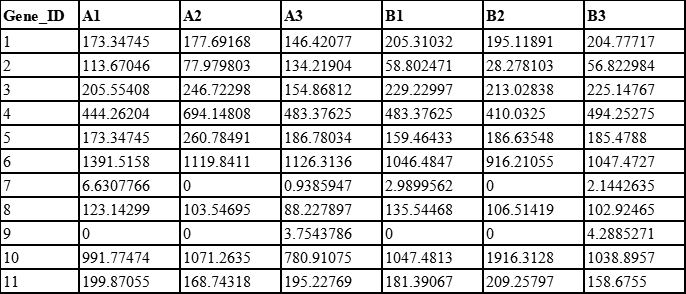
7、按照分组方式计算平均数,记为basemean(这里为了展示方便保留了两位小数)
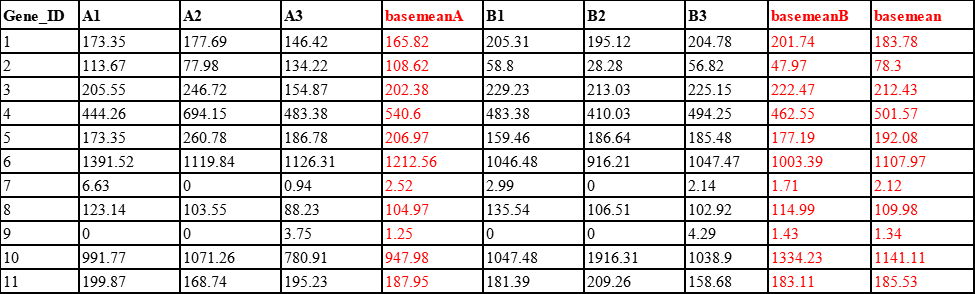
自此我们已经得出了A组与B组每个基因的basemean,接下来通过两个basemean相除,我们就可以得到差异倍数foldchange,再通过负二项分布检验,我们就得到了代表显著性的P值和padj值。这样一张差异表达分析表是不是就初见雏形了呢!
“太长不看总结版”
从DESeq的basemean的计算原理我们可以知道:
NO.1basemean不是通过fpkm计算的,也不是直接求平均数算的,是先用read count除以sizefactor进行均一化,再用均一化之后的read count按分组方式计算平均数。所以我们说basemean是基因表达量的“均一化”结果,而不能直接说是fpkm的平均数。
NO.2“牵一发而动全身”:计算sizefactor时先通过横向(样品)计算Δ值,再通过纵向(基因)找出Δ值的中位数,因此一旦改变任何一个样品或一个基因,所有的sizefactor都会跟着发生变化,也就会导致basemean变化。理解了这一点,我们就可以解决这样的问题了:“A VS C和A VS B两个DESeq表,A组没有变啊,为什么两个表里同一个基因的basemeanA是不同的?”
郑重申明:本文对于basemean的计算仅用于展示软件分析原理,实际结果请以派森诺提供的差异分析结果为准。


