2020-12-09
还在苦恼研究对象没有参考基因组吗?还被困在无参无法发高分的魔咒中吗?没有参考基因组?真的没关系!
接下来我们以2020年最新发表的两篇无参转录组文章为例,一起学习一下在高分杂志中无参转录组应该如何捕获编辑 “芳心”!
文章一
无参转录组分析揭示硬壳蛤(Mercenaria mercenaria)色素沉着的初步分子机制(IF:6.205)

研究背景
颜色在伪装,性选择,免疫和进化中起着至关重要的作用。软体动物具有鲜明的外壳颜色,色素沉着在幼年期就已经开始。硬壳蛤(Mercenaria mercenaria)是广受养殖的双壳类动物,经济价值很高。为了探究幼年蛤中色素沉着的分子机制,本研究对无色素、白色和红色硬壳蛤进行了RNA-Seq分析,选出差异基因并对其进行KEGG注释,初步揭示了硬壳蛤色素沉着的分子机制。
研究思路
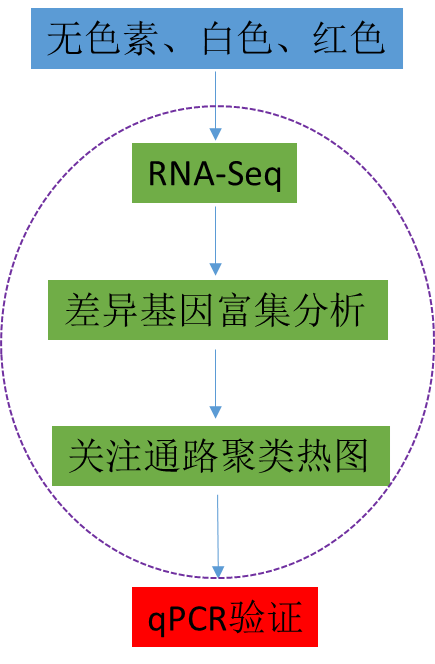
部分数据展示
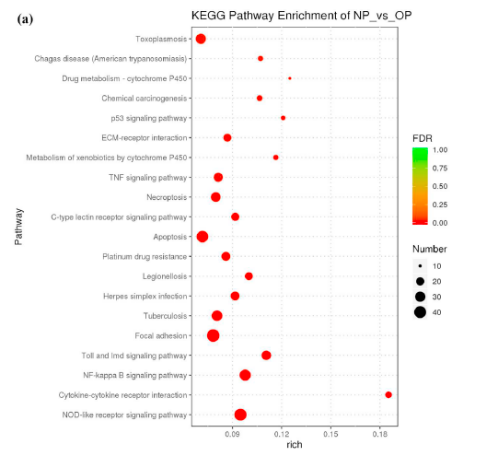
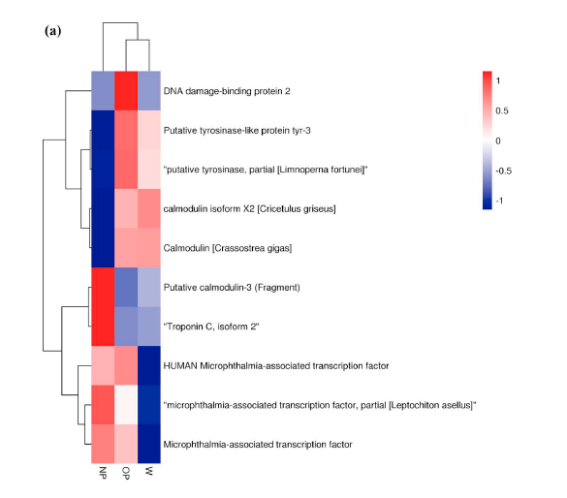

研究结论
与果蝇和斑马鱼的身体色素沉着相比,壳色素沉着是一个复杂的过程,尤其是在幼年时期,人们对其知之甚少。色素是贝壳颜色产生的主要原因。在本研究中,我们对非色素沉着、白色和红色硬壳蛤进行了RNA-Seq分析,以探讨其色素沉着的机制。DEGs参与黑色素瘤/黑色素形成、ABC转运体、类胡萝卜素代谢相关途径、卟啉和叶绿素代谢,可能参与色素的形成和调控。这些发现表明黑色素和类胡萝卜素可能是硬蛤壳的主要色素。特别是与脂质转运、ABC转运蛋白、卟啉和叶绿素代谢相关的基因,可能是这些蛤蜊壳呈红色的原因。
文章二
微小亚历山大藻及其产物膝沟藻毒素对卤虫(Artemia salina)毒害作用转录组

研究背景
有毒微藻类的扩散已成为对海洋环境,人类健康和水产养殖业的全球威胁。亚历山大藻属是最常见的会产生水华的藻类,并引起麻痹性贝类中毒(PSP),这是最关键和最普遍的有害微藻类中毒综合症之一。亚历山大(Alexandrum)属有许多种,其中微小亚历山大藻(Alexandrum minutum)在全世界沿海地区分布最广,且主要产生膝沟藻毒素。微小亚历山大藻(A. minutum)和膝沟藻毒素对营养水平较低的物种,特别是无脊椎动物,尤其是甲壳类动物的影响在很大程度上尚不清楚。在本研究中,以卤虫为研究有机体,以研究暴露于A. minutum或细胞外产物GTX2 / 3所产生的影响。
研究思路
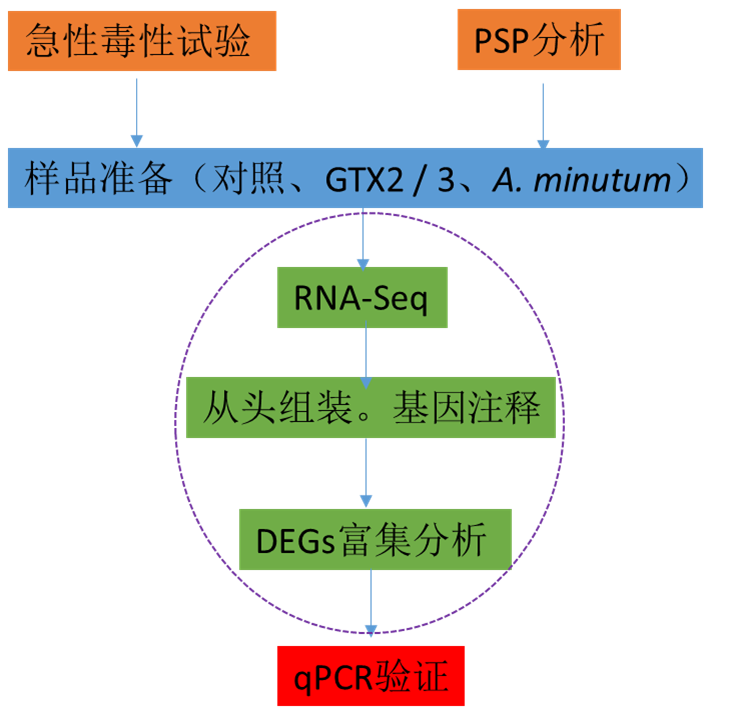
部分数据展示
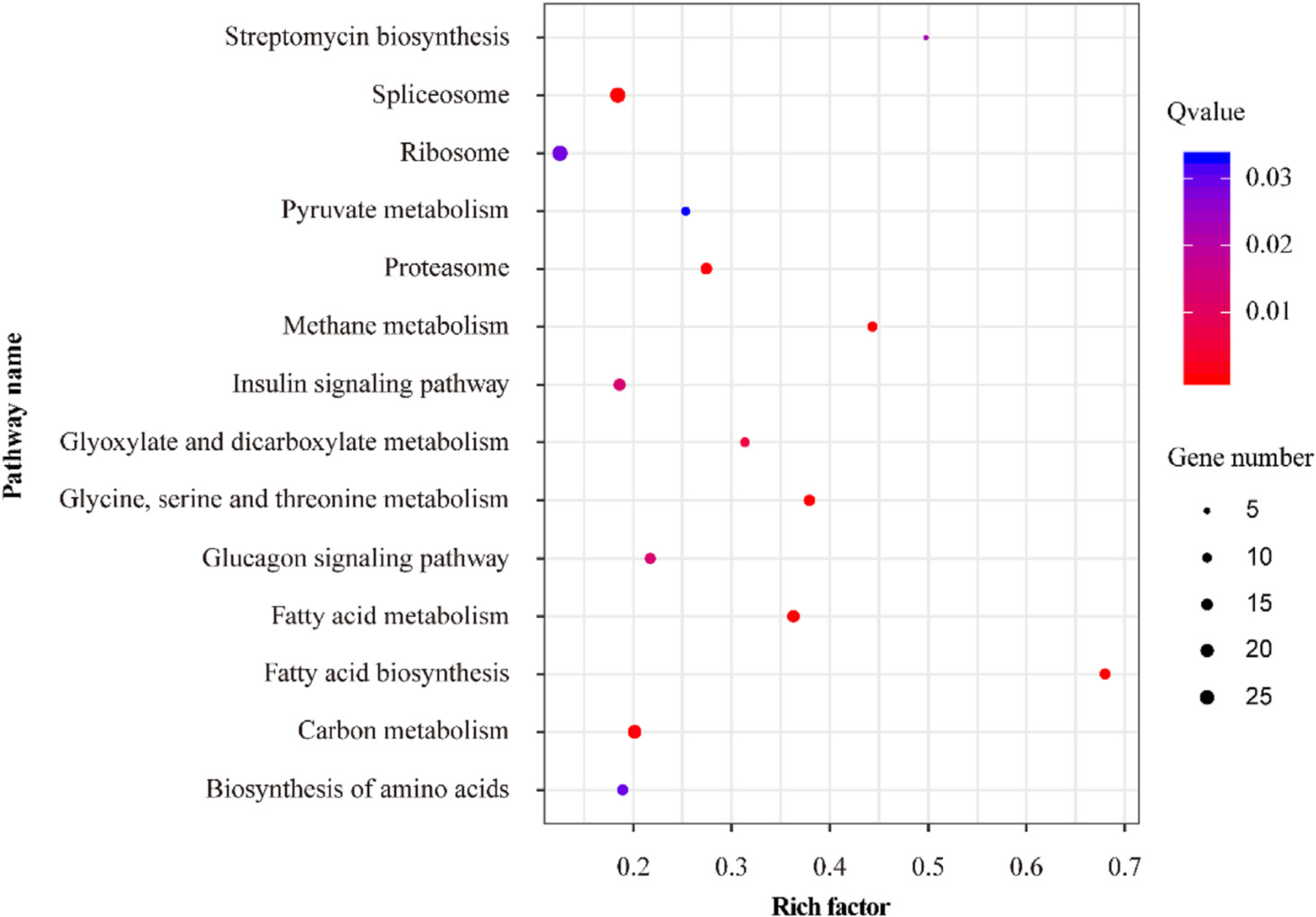
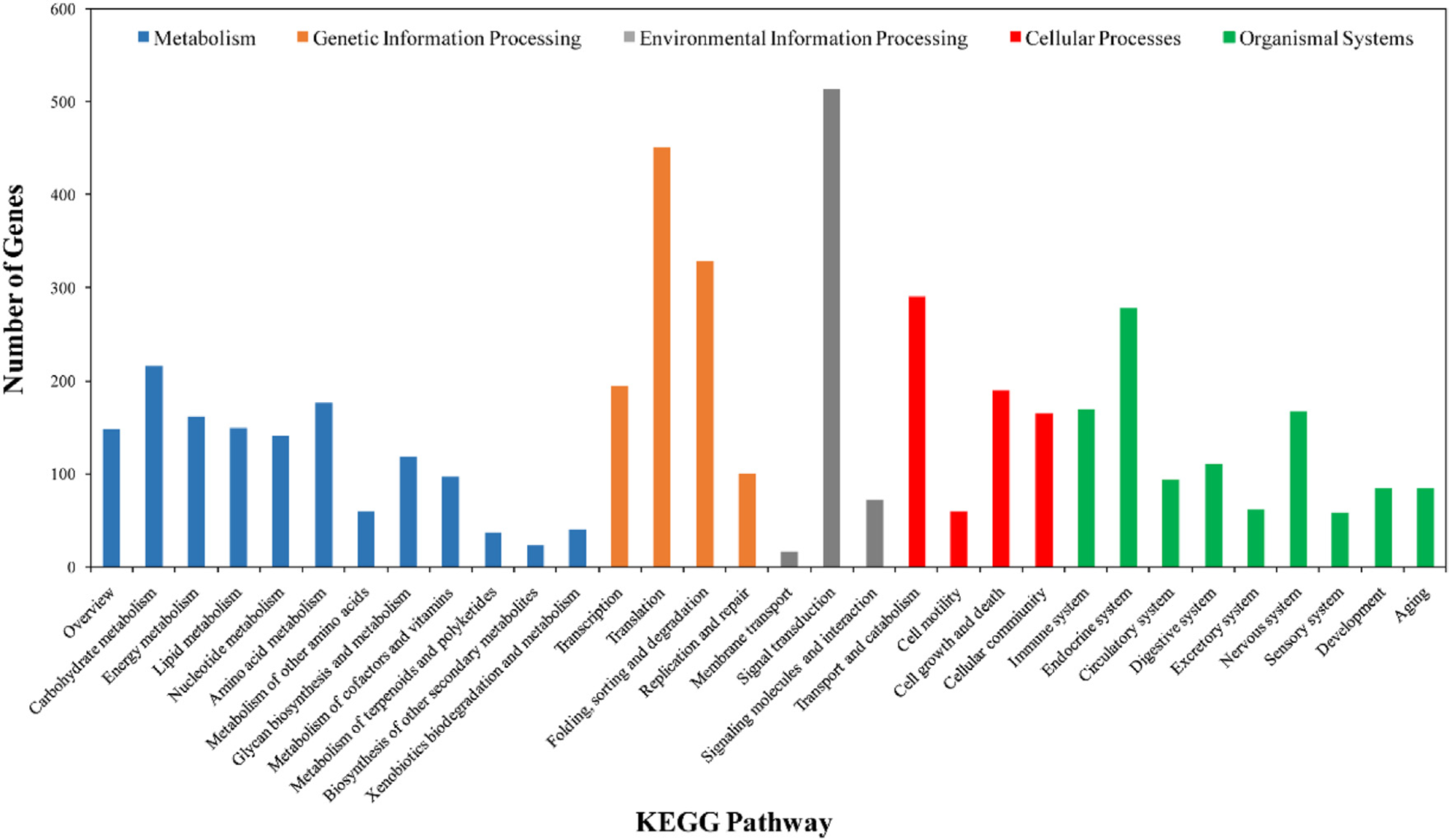
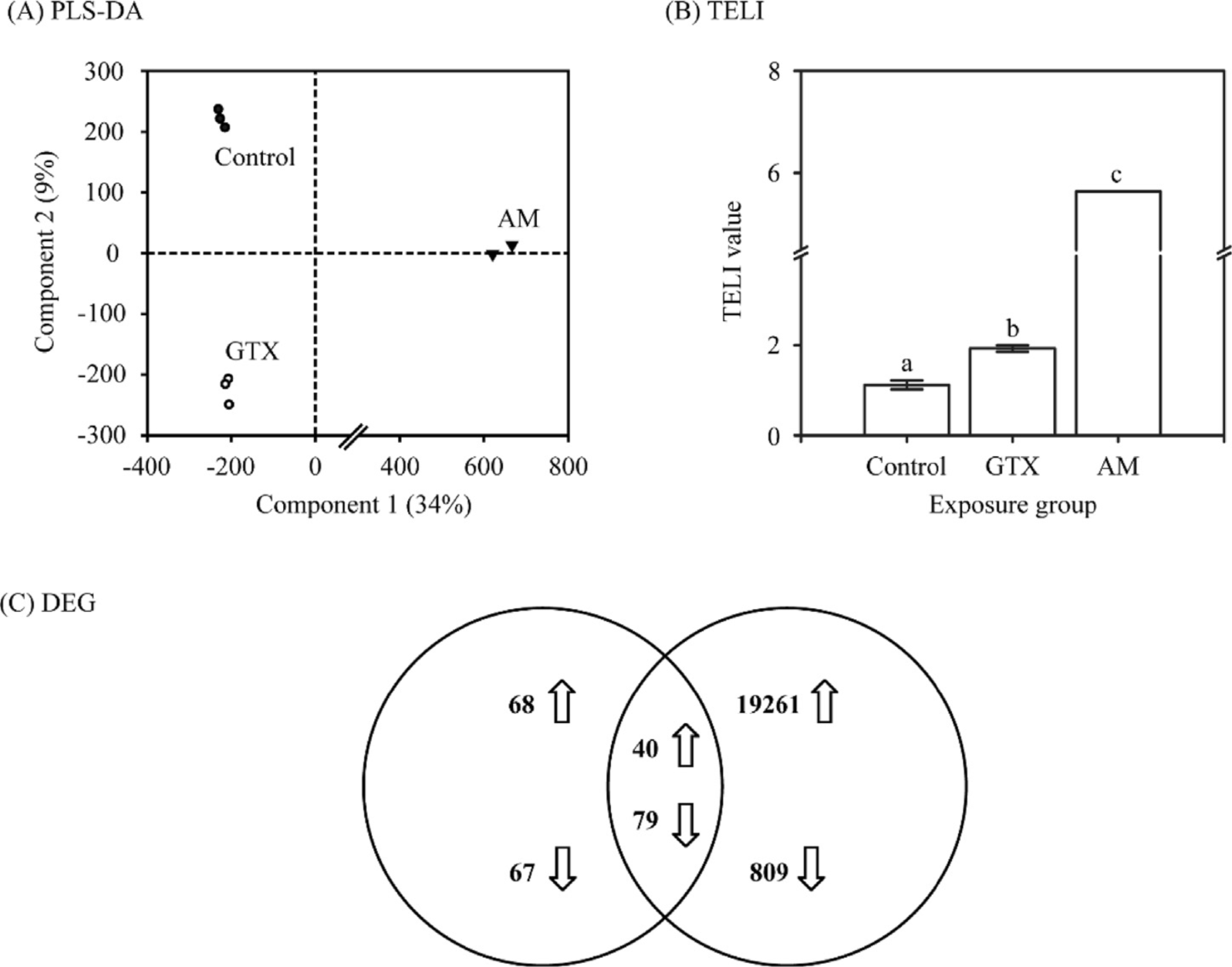
研究结论
在本研究中,采用转录组谱分析法证明了A. minutum或GTX2 / 3对卤虫的影响。该转录结果是首个应用RNA-Seq来研究在真实环境密度或浓度下,A. minutum或GTX2/3对卤虫毒害响应的转录组报告。暴露于A. minutum或GTX2 / 3的卤虫中转录组的反应并不完全相同,这可能为分析受到环境相关剂量的A. minutum或GTX2/3的影响以及卤虫如何适应暴露提供了新的机制方面的见解。
总 结
以上两个案例有两个最大的共同点:无参+高分!且研究思路都没有很复杂,都是我们普通转录组的研究方法,但是经过作者的精心雕琢,依然能被高影响因子杂志的编辑所认可,所以其写作和描述方式值得我们大家一块学习。
从这两个案例我们可以看出,即使没有前人乘凉的参考基因组,没有复杂多变的研究方法,用我们派森诺无参转录组标准分析数据加上客户的精准挖掘,依然能轻松占领高分营地!
下面链接为以上两篇文章详细信息,仅供学习使用。如有问题,欢迎与我们取得联系~
1.Zhi Hu, Hao Song, Cong Zhou, et al., De novo assembly transcriptome analysis reveals the preliminary molecular mechanism of pigmentation in juveniles of the hard clam Mercenaria mercenaria[J]. Genomics, 2020,Pages 3636-3647,ISSN 0888-7543.
https://doi.org/10.1016/j.ygeno.2020.04.020.
2. Xianliang Yi, Keke Zhang, Renyan Liu, et al., Transcriptomic responses of Artemia salina exposed to an environmentally relevant dose of Alexandrium minutum cells or Gonyautoxin2/3[J]. Chemosphere, Volume 238,2020,124661,ISSN 0045-6535.
https://doi.org/10.1016/j.chemosphere.2019.124661.


