2020-12-18

12月2日,派森诺基因云“云图汇”模块中上线了一批精品分析工具,不知各位小伙伴是否有尝鲜呢?【12月的第一批高效精美作图小工具热血上线,你get了吗?】(点击查看)今天我们先来了解下RDA/CCA/PCoA_envfit这几个分析内容。
对于微生态领域(多样性组成谱/宏基因组/宏转录组等)的测序数据,除了通过差异统计分析来筛选各组样本中的微生物标志物种或基因(Biomarker)的信息外,我们还需要将其它各种检测手段获得的数据,比如代谢组、蛋白组、或检测获得的理化指标、临床指标等各类数据,与微生物组的海量数据进行关联分析,以期找出与各类指标变化相关联的具体微生物物种及其基因。这种研究思路,统称为全微生物组关联分析(Microbiome-wide association study,MWAS)。目前已有多种算法,可以帮助我们进行上述多组学数据的联合分析。
RDA冗余分析(Redundancy analysis)是一种典型的约束排序方法,可以理解为“有约束条件”的PCA分析,通过多元线性回归(Multiple linear regression,MLR),将菌群结构数据与某一种或多种给定的影响因素互相拟合,并通过置换检验来判断这些因素对于菌群结构的影响是否显著。
CCA典型关联分析(Canonical Correlation Analysis)与RDA分析相似,亦是最常用的挖掘数据关联关系的算法之一。其中RDA分析是基于线性模型,CCA分析则是基于单峰模型(通常可以两种分析模型都做下尝试;有动手分析能力的小伙伴,还可以通过R-Vegan包中的decorana函数,进行DCA分析,如果DCA排序前4个轴中最大值超过4,选择单峰模型;如果是小于3,则选择线性模型;如果介于3与4之间,两者都可以哦~)。
但当我们利用约束排序方法(如RDA或CCA分析等),发现环境变量的解释程度较低、物种与环境变量间的排序结果较为混乱、组间差异不明显时,说明约束排序模型并不适用;这时我们可以考虑通过非约束排序模型(PCoA、NMDS、PCA分析等)拟合环境变量的方式来解释“环境—个体—菌群”间的关系,即这里提到的PCoA_envfit分析。该分析是基于样品间距离矩阵(bray_curtis或unifrac等),通过排序分析获得样本在排序轴上的分布,接着利用回归函数envfit进行环境因子与排序轴之间的拟合分析,最后通过置换检验(permutation test)进行显著性分析,一般默认选择重复999次,获取相关数据及绘图。
我们在派森诺基因云的“云图汇”模块中,已经发布了这三种分析作图的工具。同时,我们也在“云学院”的“云文档”中,提供了具体操作方法,小伙伴可以按照提示,来操作尝鲜哦!
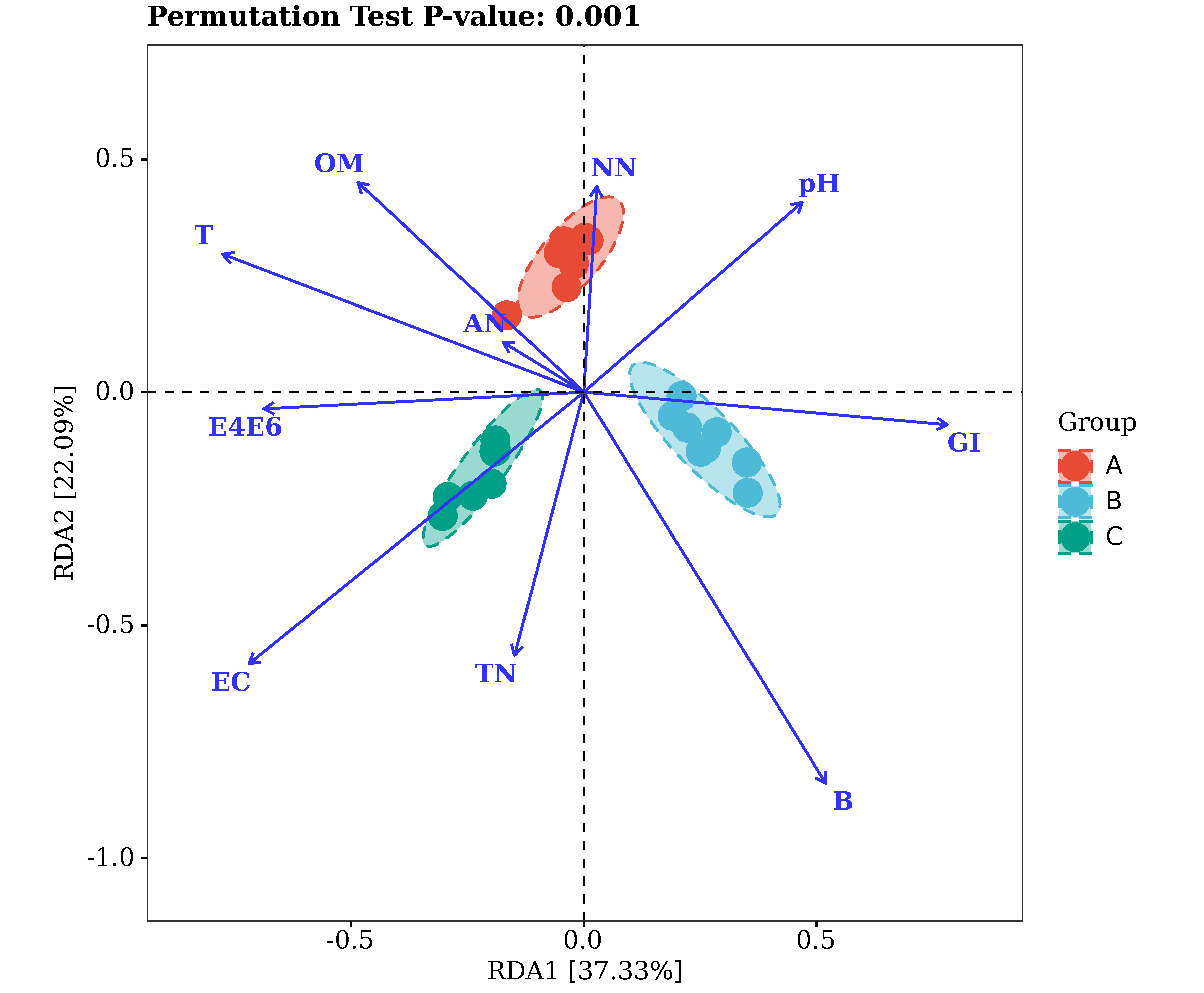
派森诺基因云RDA分析
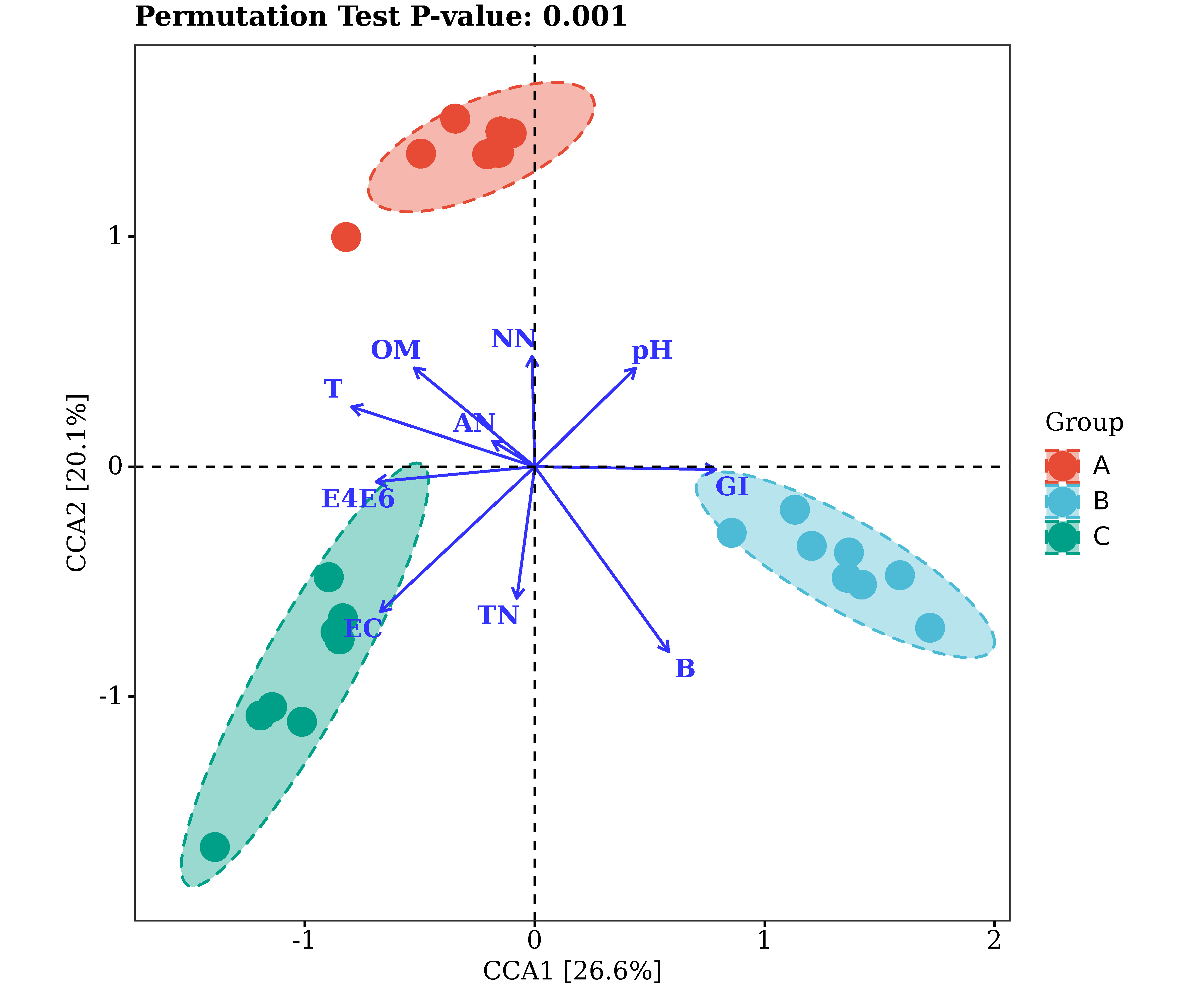
派森诺基因云CCA分析
注:图中,每个点代表一个样本,不同颜色的点属于不同分组,两点之间的距离越接近,说明两个样本的菌群组成/功能相似度越高。蓝色箭头分别代表不同的影响因素,影响因素之间的夹角代表它们之间相关性的大小,锐角表示两个因素正相关,直角为不相关,钝角时为负相关,射线越长,表明该因素对菌群组成/功能的影响作用越大;蓝色箭头射线和坐标轴的夹角代表某个环境因子与坐标轴的相关性大小,夹角越小,相关性越高。样本在蓝色箭头上投影点的位置,近似代表该因素在对应样本中的数值大小;排序图上方的P值代表采用蒙特卡洛置换检验获得的P值,P值越小,影响因素对于菌群组成/功能的作用越显著。坐标轴括号中的百分比代表了对应的坐标轴所能解释的原始数据中差异的比例。
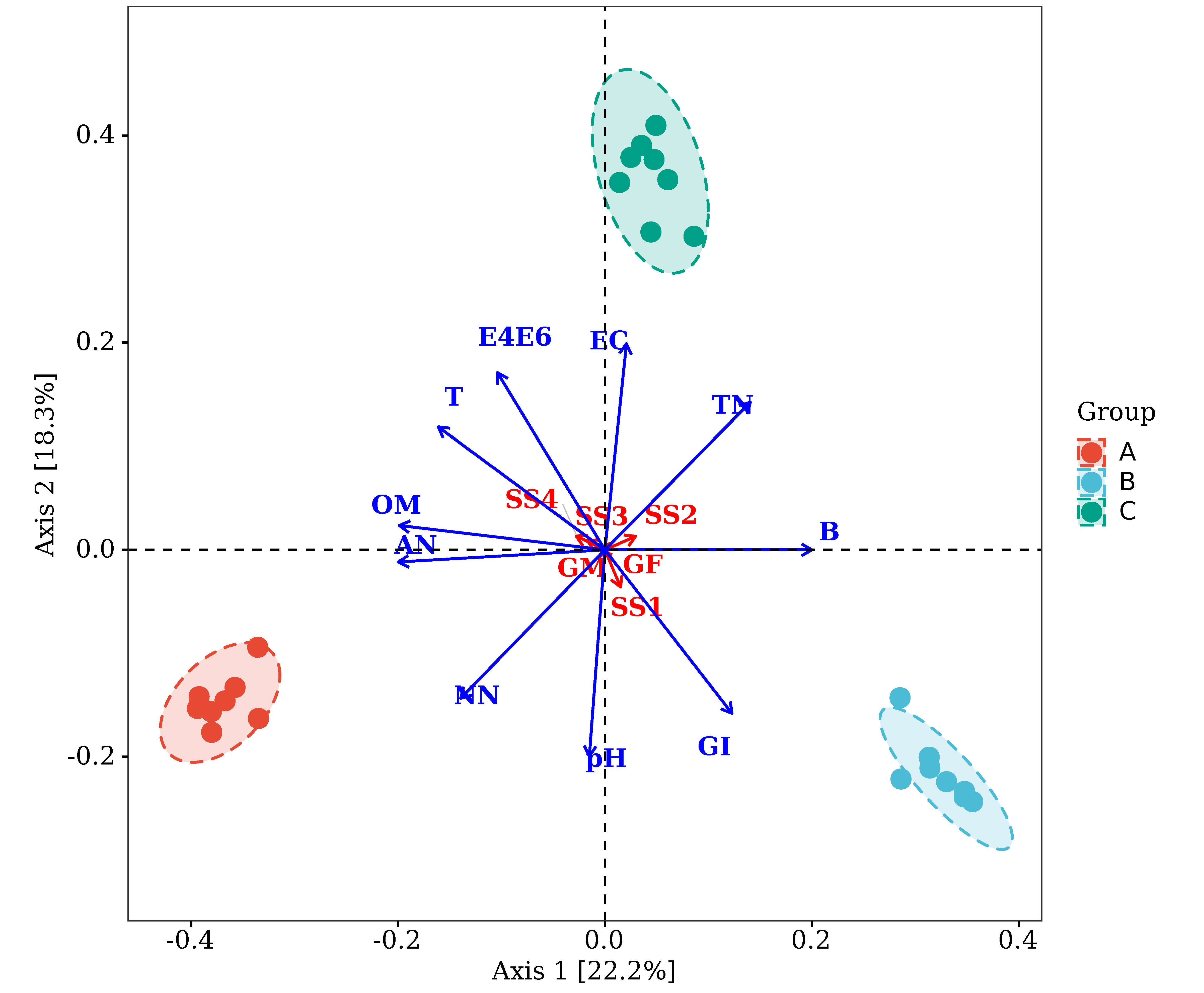
派森诺基因云PCoA_envfit分析
注:图中,每个点代表一个样本,不同颜色的点属于不同分组,两点之间的距离越接近,说明两个样本的菌群组成/功能相似度越高。蓝色箭头(数值变量:如pH、浓度等)与红色箭头(性状变量:如性别、部位、季节等)分别代表连续型和离散型的影响因素,影响因素之间的夹角代表它们之间相关性的大小,锐角表示两个因素正相关,直角为不相关,钝角时为负相关,射线越长,表明该因素对菌群组成/功能的影响作用越大;箭头射线和坐标轴的夹角代表某个环境因子与坐标轴的相关性大小,夹角越小,相关性越高。样本在箭头上投影点的位置,近似代表该因素在对应样本中的数值大小;坐标轴括号中的百分比代表了对应的坐标轴所能解释的原始数据中差异的比例。
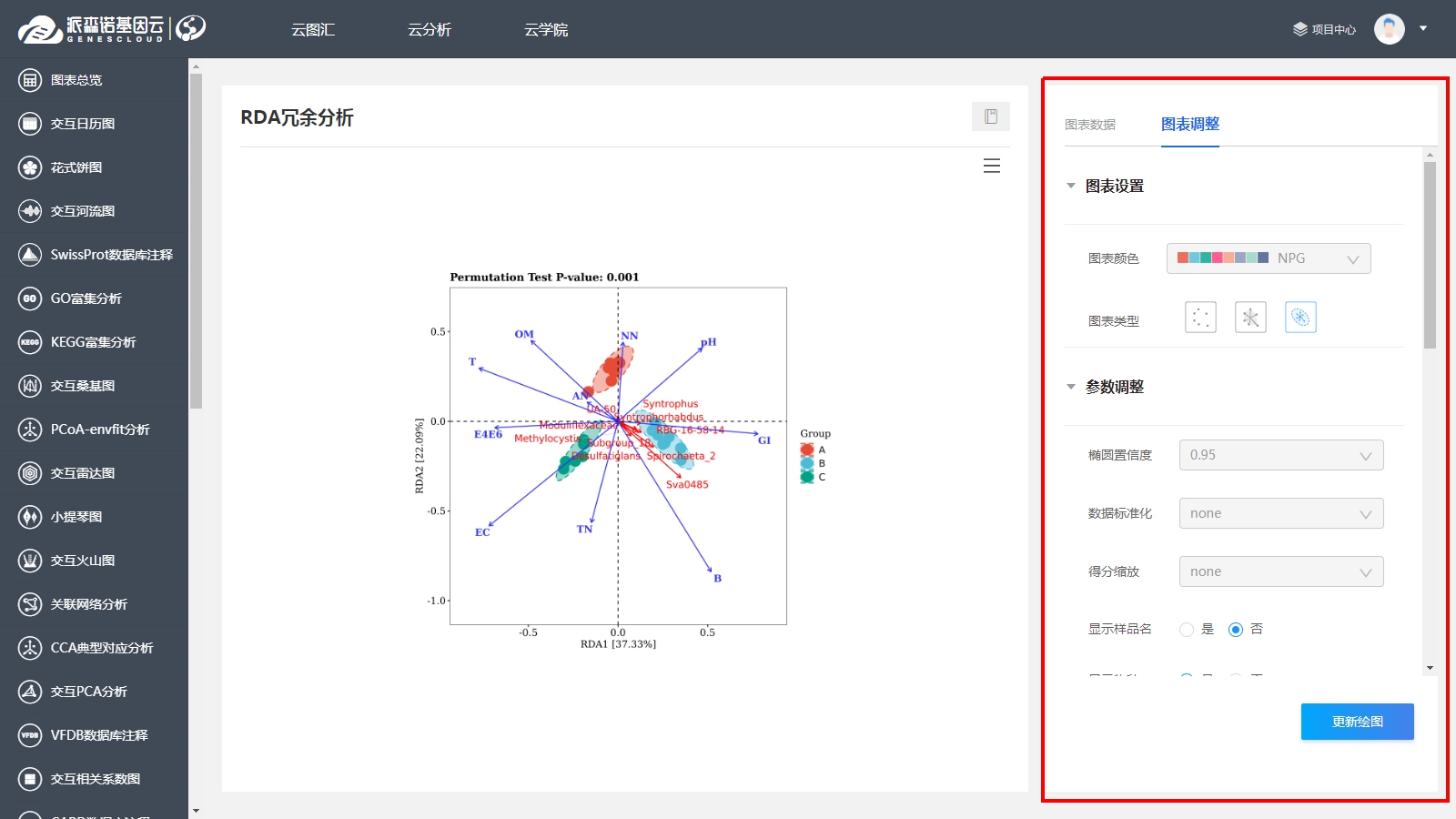
作图完成后,还可以通过右边侧栏的“图表调整”,进一步优化作图效果哦~
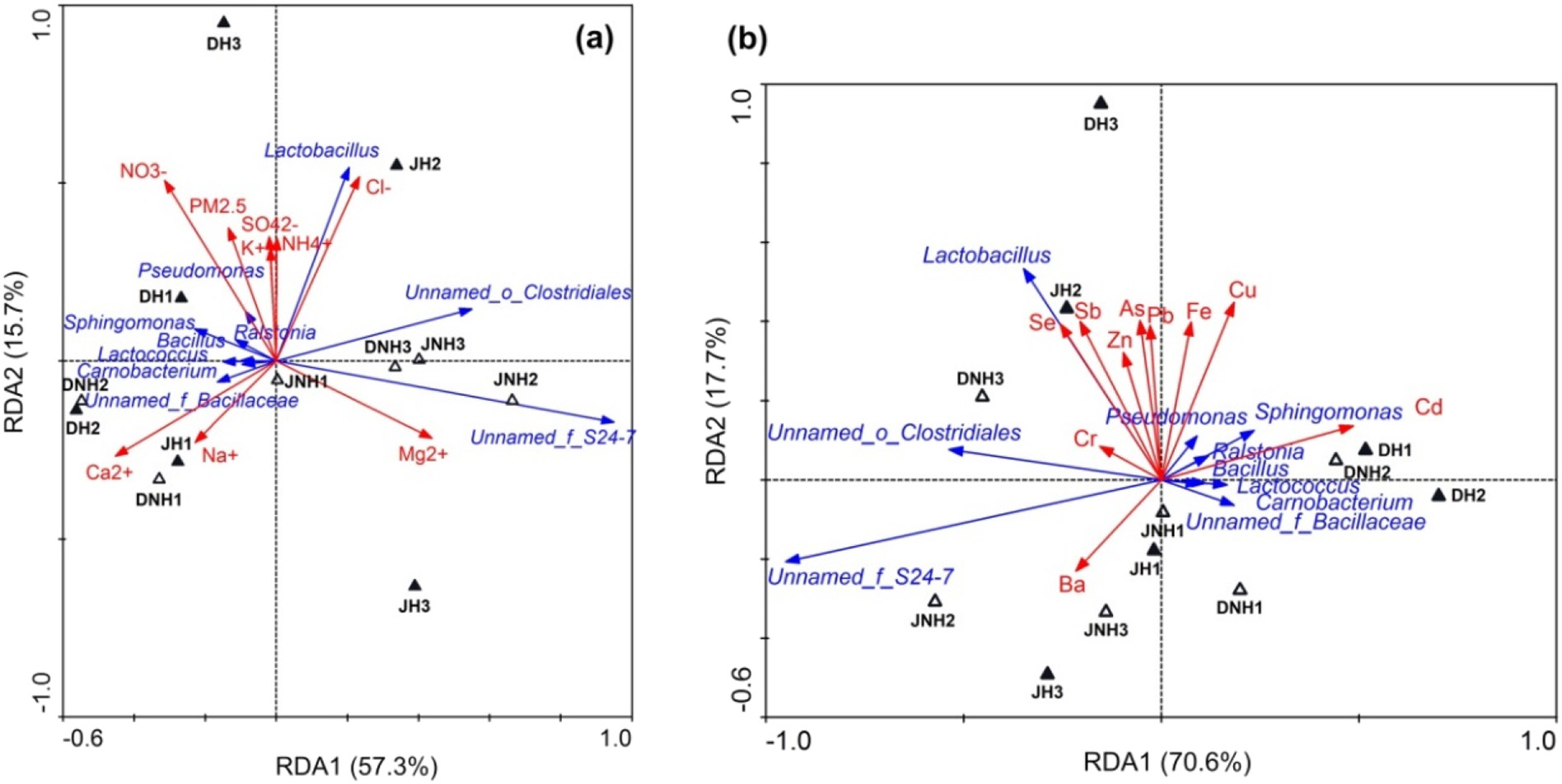
RDA/CCA/PCoA_envfit作为最常见的“环境—个体—菌群”的关联分析方法,已经在诸多研究中广泛应用,我们在这里也抛砖引玉,列举一二,供大家参考~



对于以上的分析内容,小伙伴们是否心动了呢?心动不如行动,快来派森诺基因云(https://www.genescloud.cn/)体验吧!


