2021-01-29

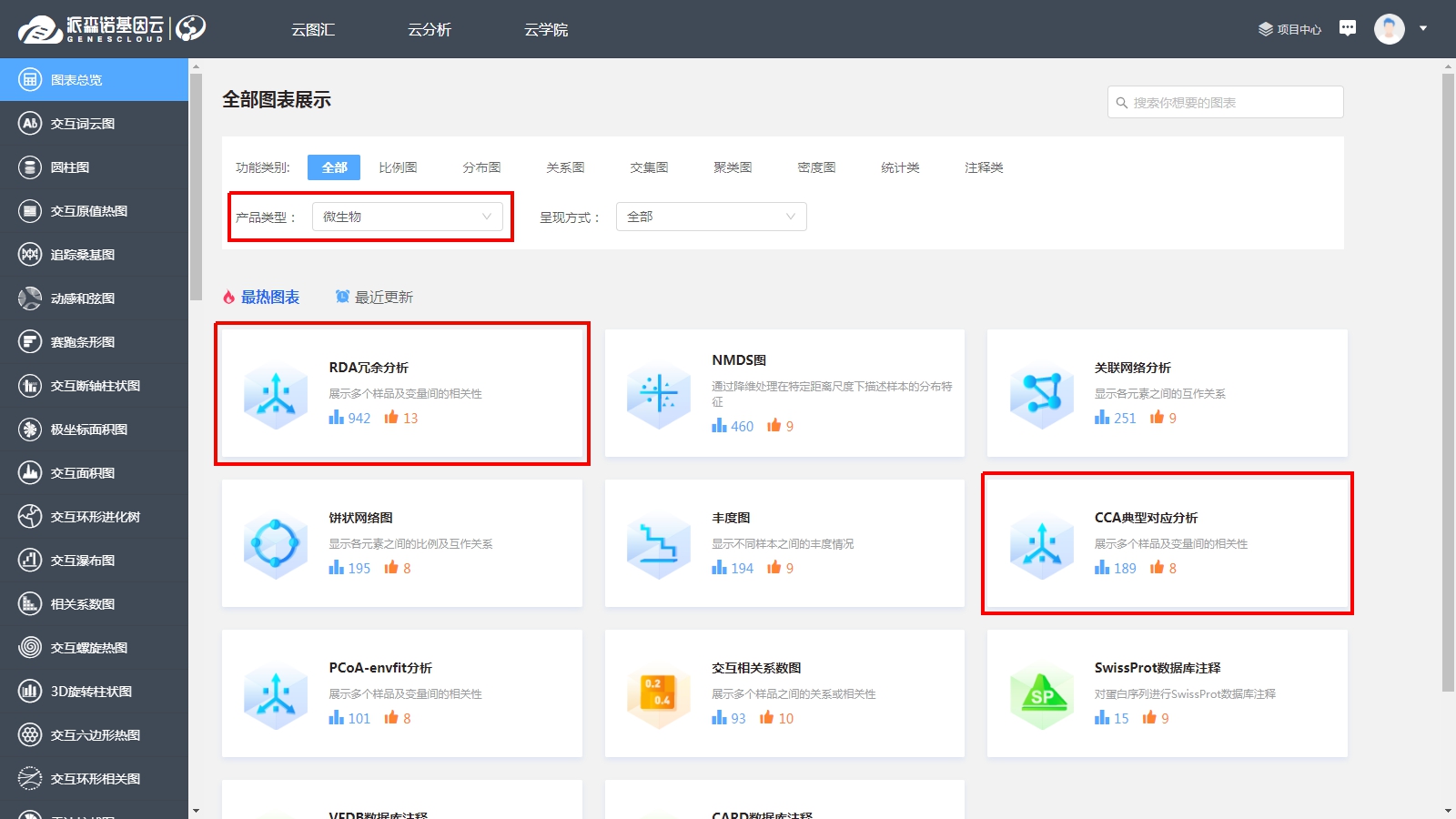
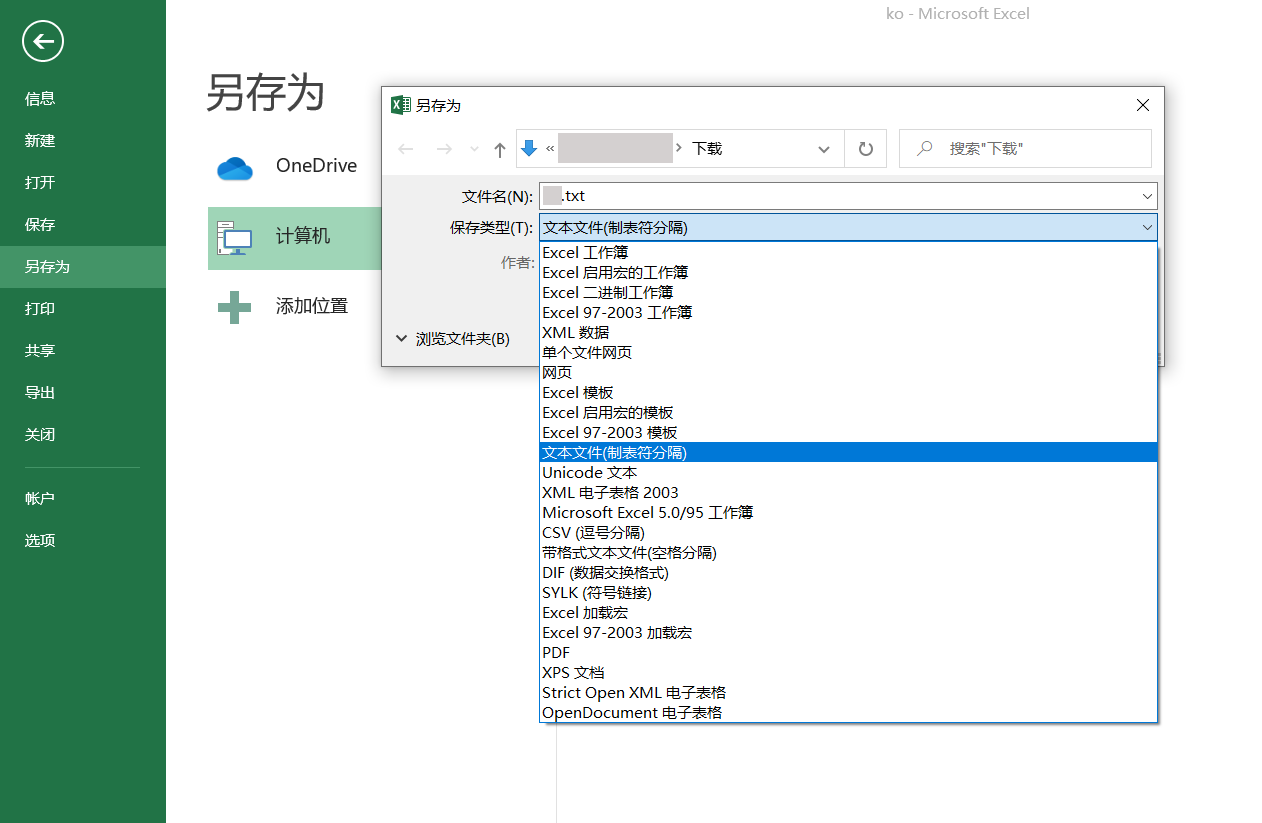
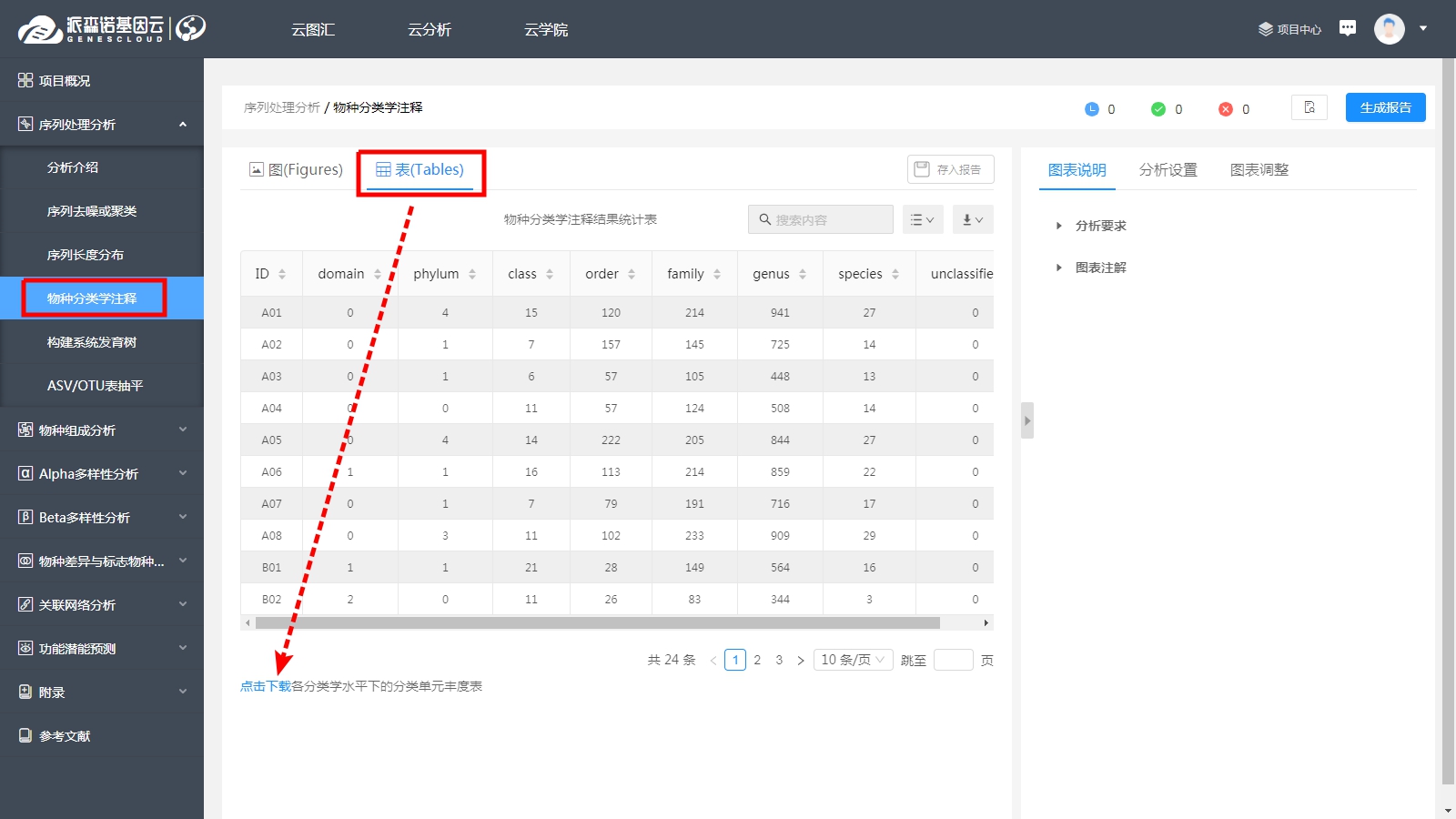
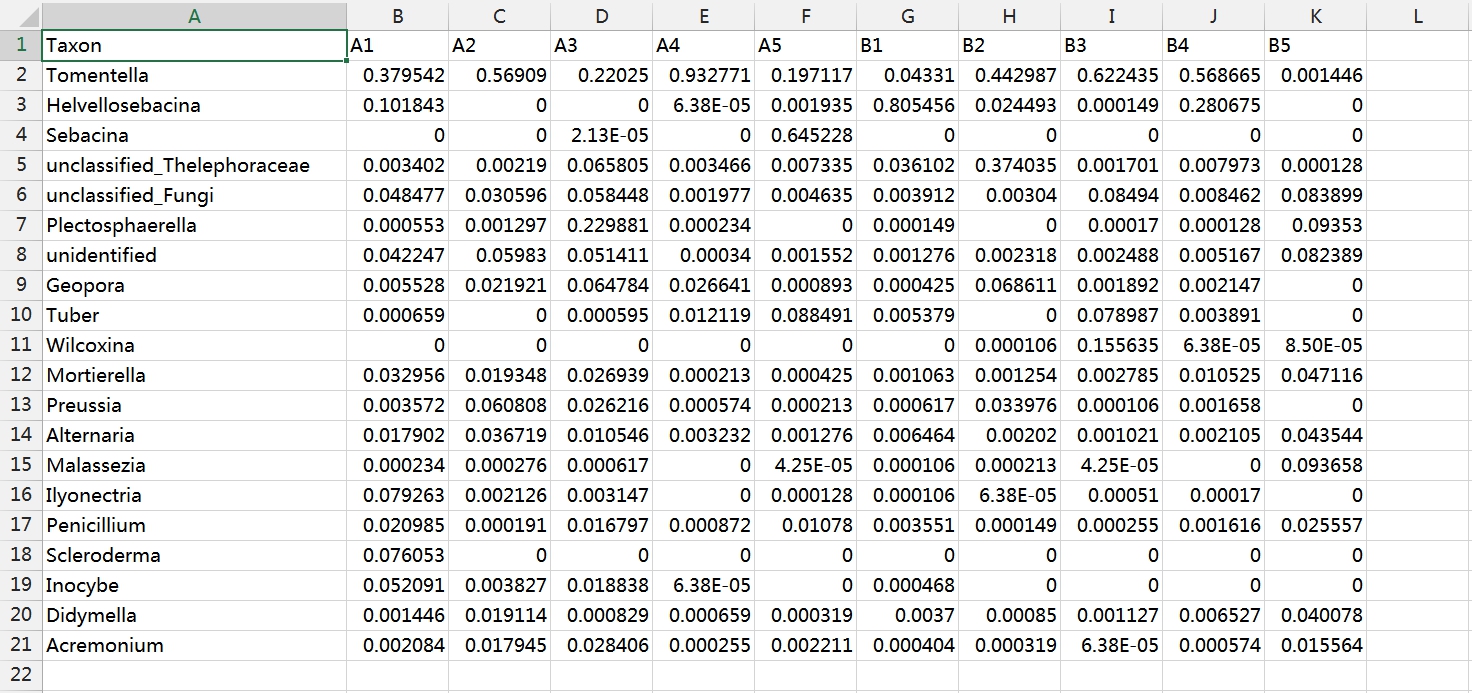
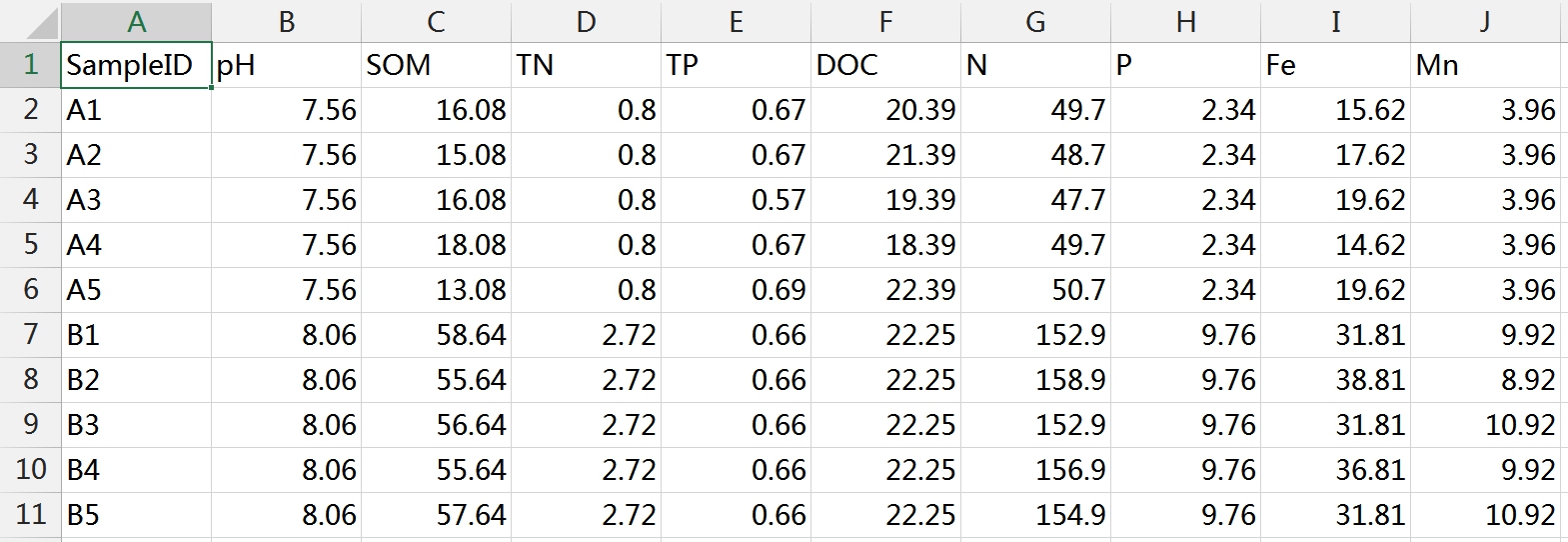
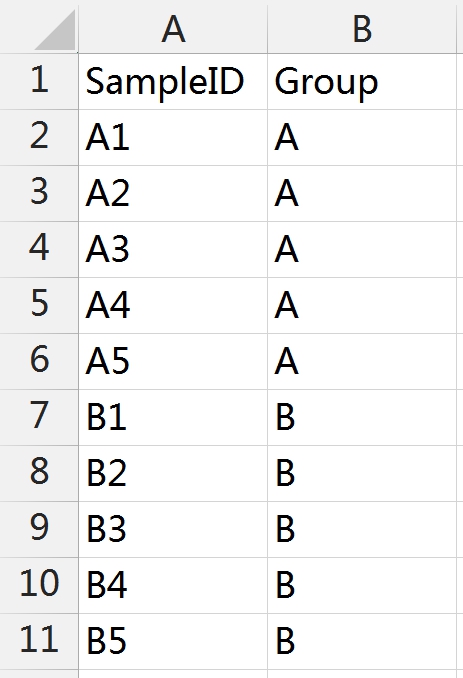
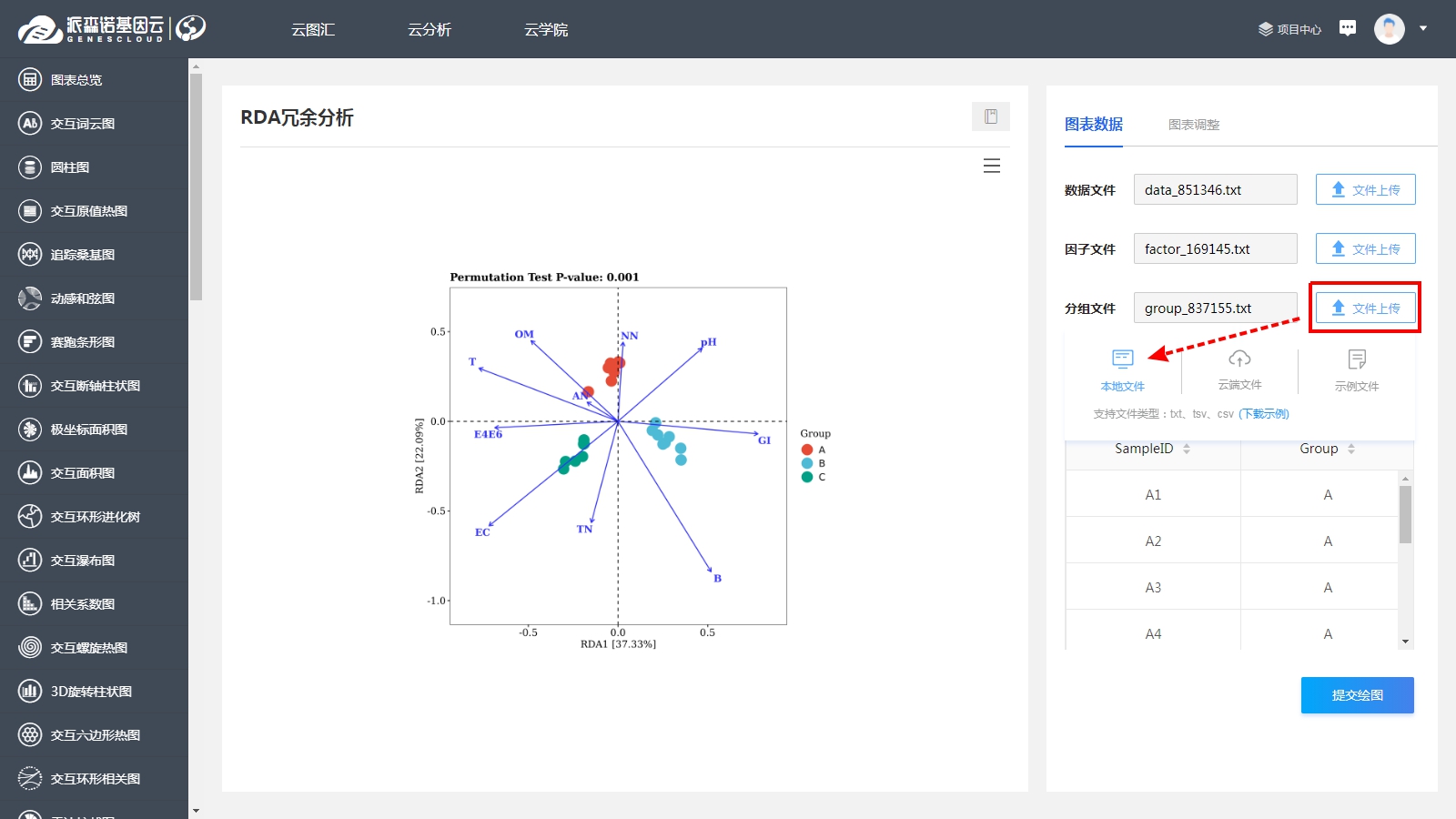
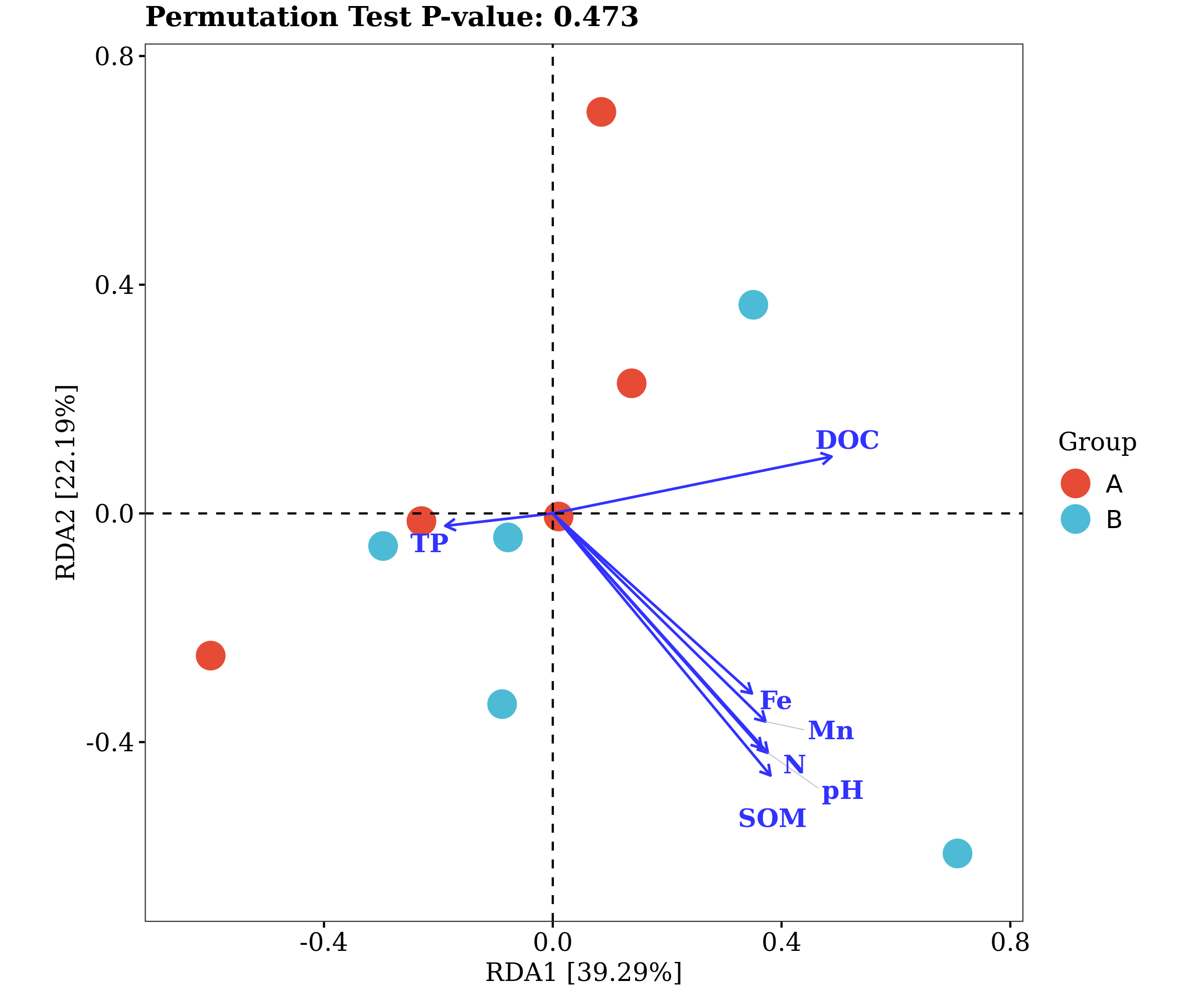
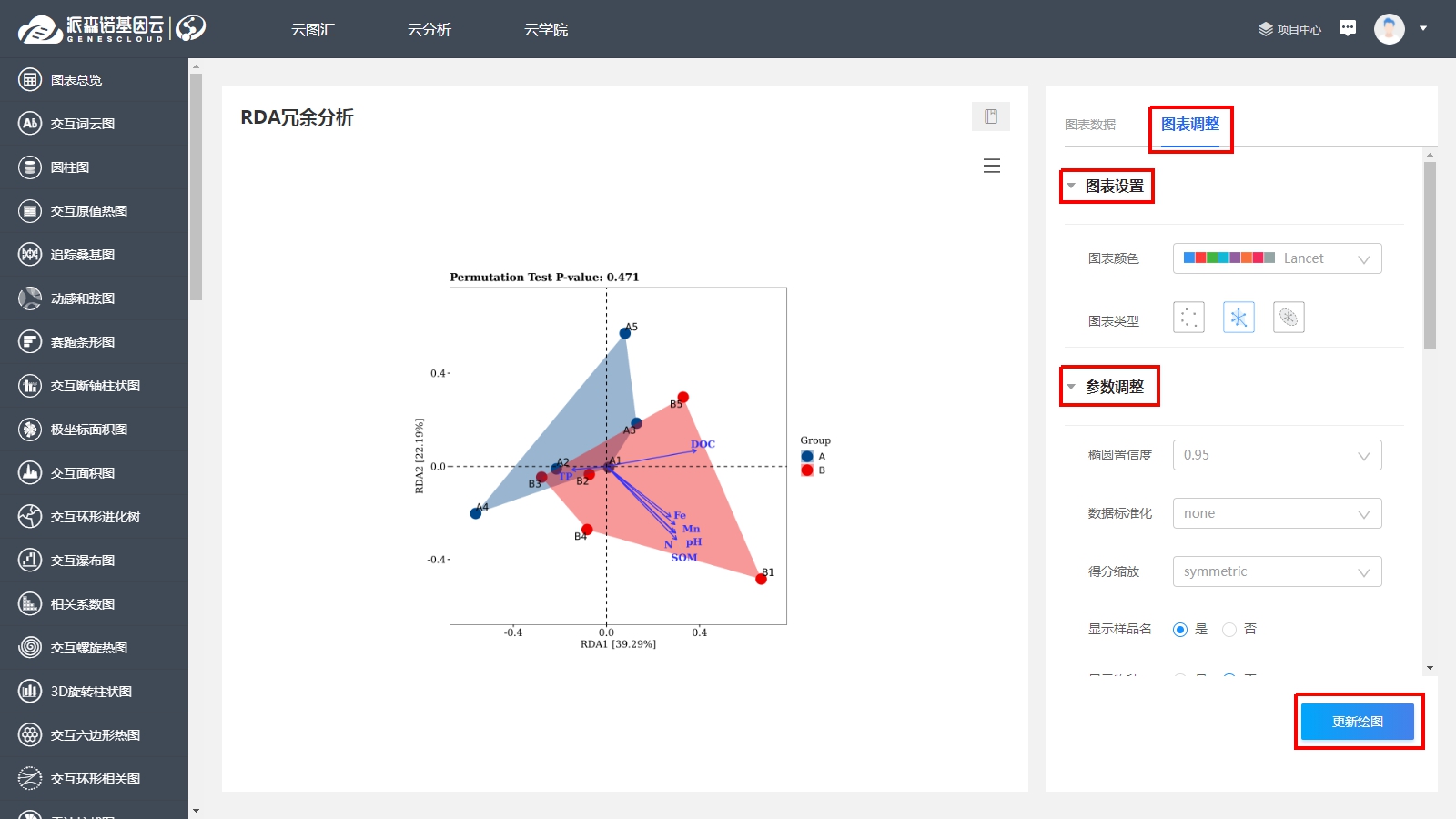
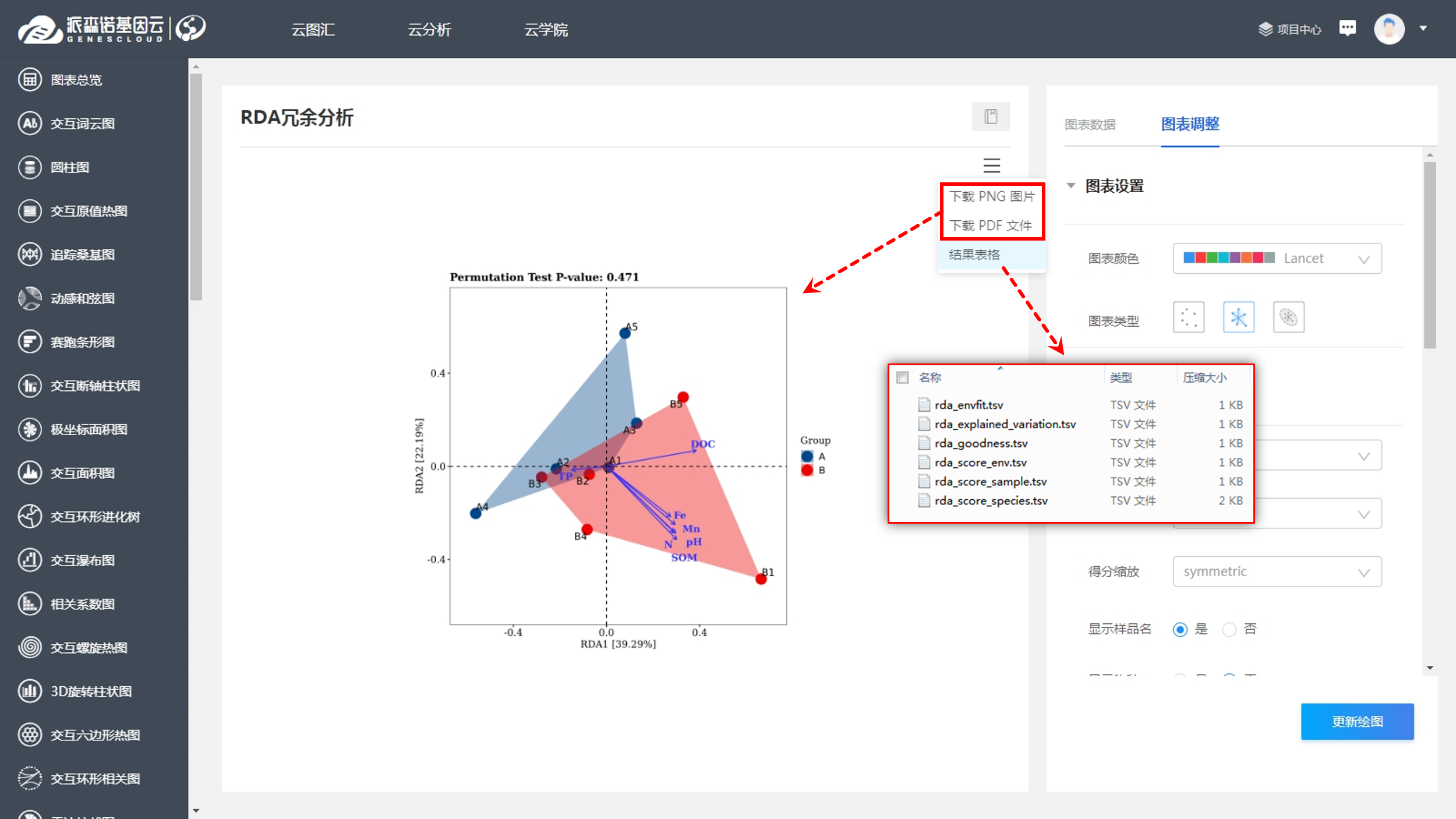
在对微生物群落进行研究时,除了分析不同群落样本之间在微生物群落组成、功能上的差异外,往往还想了解,具体有哪些因素会影响微生物群落,使其组成和功能发生变化?其中,哪种因素又占据了主导作用? 为了回答这一问题,需要将微生物群落样本的相关数据,与其对应的环境因子数据进行关联分析。在众多关联分析的方法中,出镜率最高的,就是RDA/CCA分析啦~ RDA/CCA分析一般需要使用CANOCO软件或运用R语言进行分析,这对生信小白来说,挑战还是很大滴。现在,RDA/CCA分析已经在派森诺基因云的“云图汇”模块中发布上线啦(网址:https://www.genescloud.cn/chart/)!我们也已做了初步介绍和解读(点击查看)。 简单而言,RDA(Redundancy Analysis)和CCA(Canonical Correspondence Analysis)均属于直接排序的分析方法,RDA基于线性模型,CCA则是基于单峰模型。一般情况下,两者都能有效分析微生物群落与环境因子之间的变化相关性。 那么问题来了,既然两者都可以进行,又该如何选择呢? 在进行排序分析之前,可以先对物种群落数据进行去趋势对应分析(Detrended correspondence analysis,DCA),根据结果中Lengths of gradient的值来进行判断,如果最大的值大于4,则应选择CCA,如果最大的值小于3,则选择RDA,而如果最大的值在3~4之间,则两种分析均可。然而,这种标准并不是100%合适,在实际的使用中,我们最好是同时进行CCA和RDA,根据结果进行选择~ 1、数据准备 需要准备物种组成数据(data.txt)、环境因子数据(factor.txt)、分组数据(group.txt),具体如下: 注意:在准备数据时,不能出现中文、空格、特殊字符如()、?、*、[ ]、$等。最后需要把整理好的Excel文件另存为“制表符分隔”的txt格式文件。 ① 物种组成丰度数据(data.txt) ● 通过网盘下载数据的项目:Treat*\2.2.2_taxa_summary\ungrouped\otu_table_L*.xls文件,对应门纲目科属种等不同分类水平下的物种组成丰度表(QIIME2版本); ● 通过基因云获取文件的项目:在下载链接中,下载“各分类学水平分类单元丰度表”。 以属水平上的数据为例,将表格整理成如下形式(第一列为属名,第一行为样本名)后,另存为txt文档: ② 环境因子数据(factor.txt) 根据各样本环境因子的测定结果,可将其整理成如下形式(第一列为样本名,第一行为因子名),整理好后,同样另存为txt文档。 注意:环境因子的个数应小于样本的个数。 ③ 样本分组文件(group.txt) 根据分组将样本整理成如下形式(第一列为样本名称,第二列为分组情况),整理好后,同样另存为txt文档。 2、将整理好的三个文件上传至“云图汇” ●以RDA为例(https://www.genescloud.cn/chart/RDA),在“图表数据”标签中,点击“文件上传→本地文件”,即可选择刚才我们已经整理好的文件进行分析。 ● 待3个文件均上传成功后,点击“提交绘图”,就可以轻松得到分析结果啦!是不是很简单呢? ● 随后,我们还可以在“图表调整”模块中,对样本颜色、分析参数等进行进一步的调整优化哦,调整完点击“更新绘图”,就可以看到结果啦~ ● 最后,我们可以点击下载结果图片及表格文件哦~ 以上就是今天给大家介绍的云图汇RDA/CCA操作指南了,当然,作图效果的好坏,很大程度上还是取决于分析结果是否理想、微生物群落是否和环境因子间真的存在关联(例如,上图中的P值是不显著的)。 欢迎大家进入基因云尝试使用哦~也欢迎大家在讨论区留言或者发邮件给我们哟(邮箱地址:metasupport@personalbio.cn)!