2021-02-05

“派森诺基因云”发布至今,基于QIIME 2内核的微生物群落交互分析可视化系统深受大家热爱,并以专业、灵活、强大、丰富的动态交互和可视化效果,为大家带来了全新的云端分析体验!
QIIME 2云分析主要包括序列处理分析、物种组成分析、Alpha多样性分析、Beta多样性分析、物种差异与标志物种分析、关联网络分析和功能潜能预测共7大分析模块,每个模块下又包含多种分析方法和步骤。对于生信小白而言,面对琳琅满目的分析方法、眼花缭乱的图表,难免会迷失“焦点”:我在研究报告中该呈现哪些分析结果?哪些分析结果最重要呢?
为此,我们悉心整理了各个分析模块中的热点分析内容,推出“5分钟小课堂系列”:为大家带来专业的解读!今天的第一讲,我们聚焦于物种组成分析和Alpha多样性分析两大模块,希望可以帮助大家更好地理解微生物组测序分析的结果哦~
01、物种组成分析
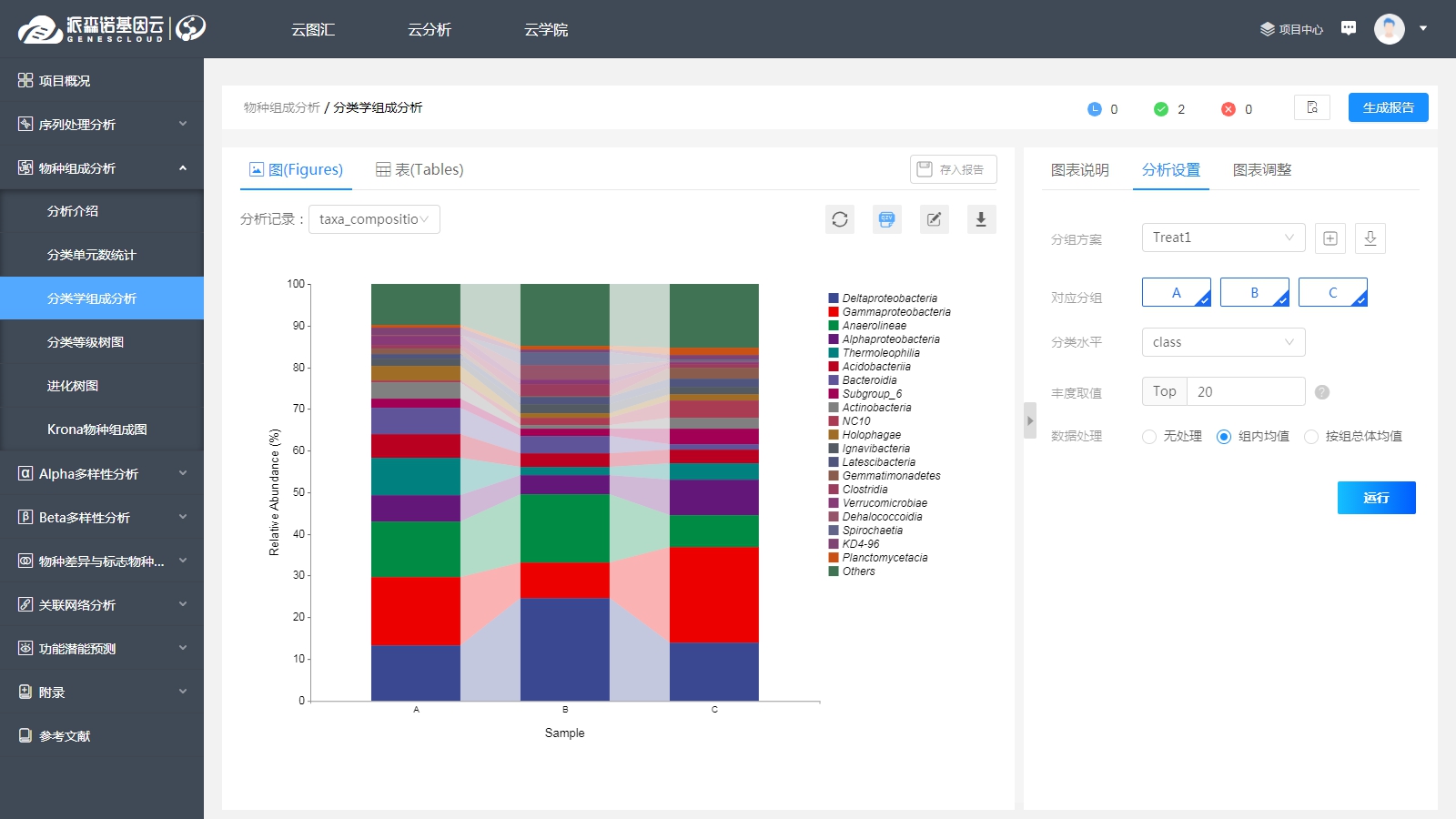
在这一模块中,常用的就是“分类学组成分析”了:可以通过堆积柱状图,展现各个/各组群落样本中,微生物的物种组成,主要用于描述群落样本中的优势物种的丰度及其变化规律。图中,每一个柱子代表一个群落样本或一个分组,每种颜色代表某一特定分类水平下(门/纲/目/科/属/种)的一种微生物,柱子越长,对应物种的丰度就越高。
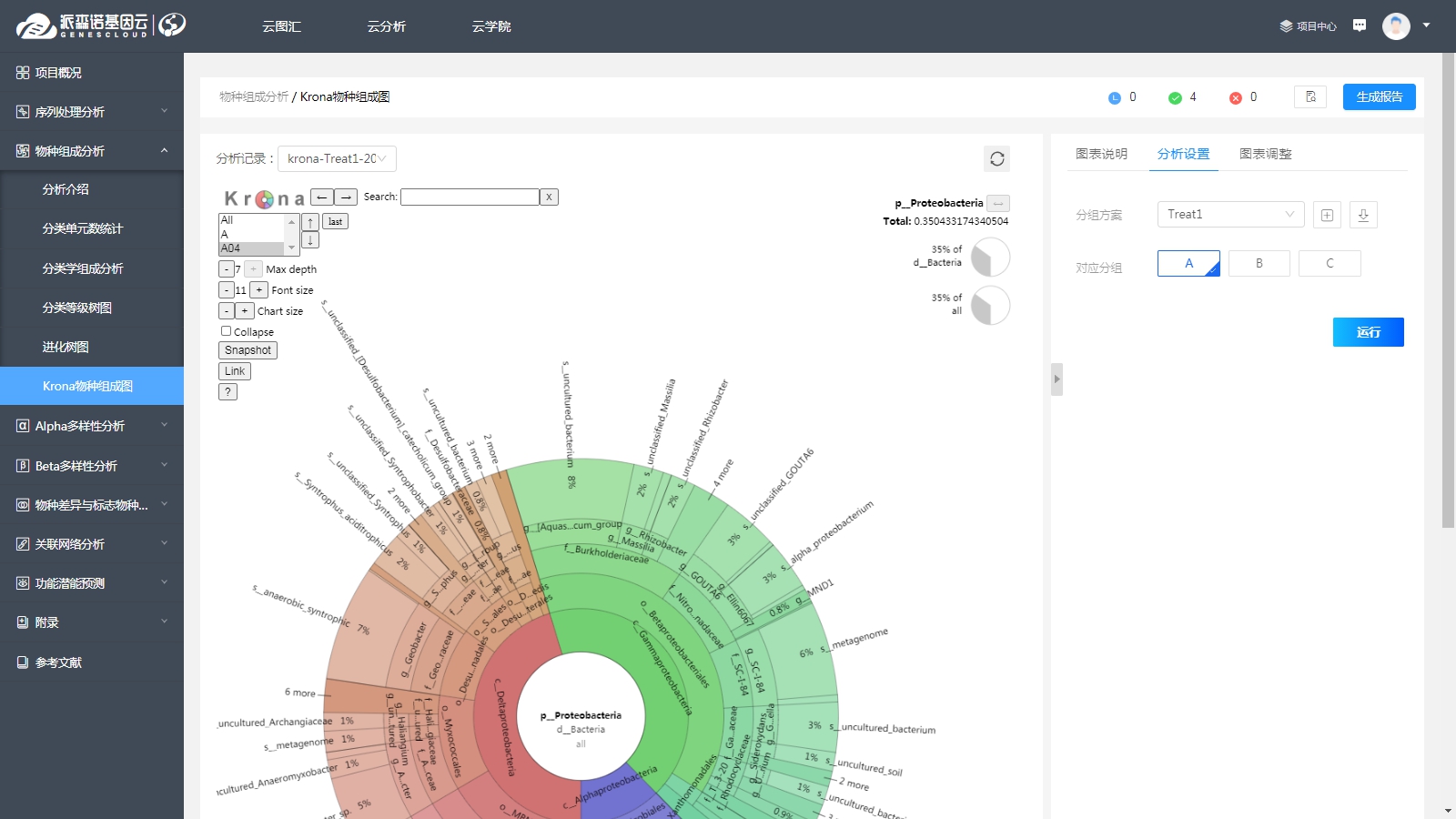
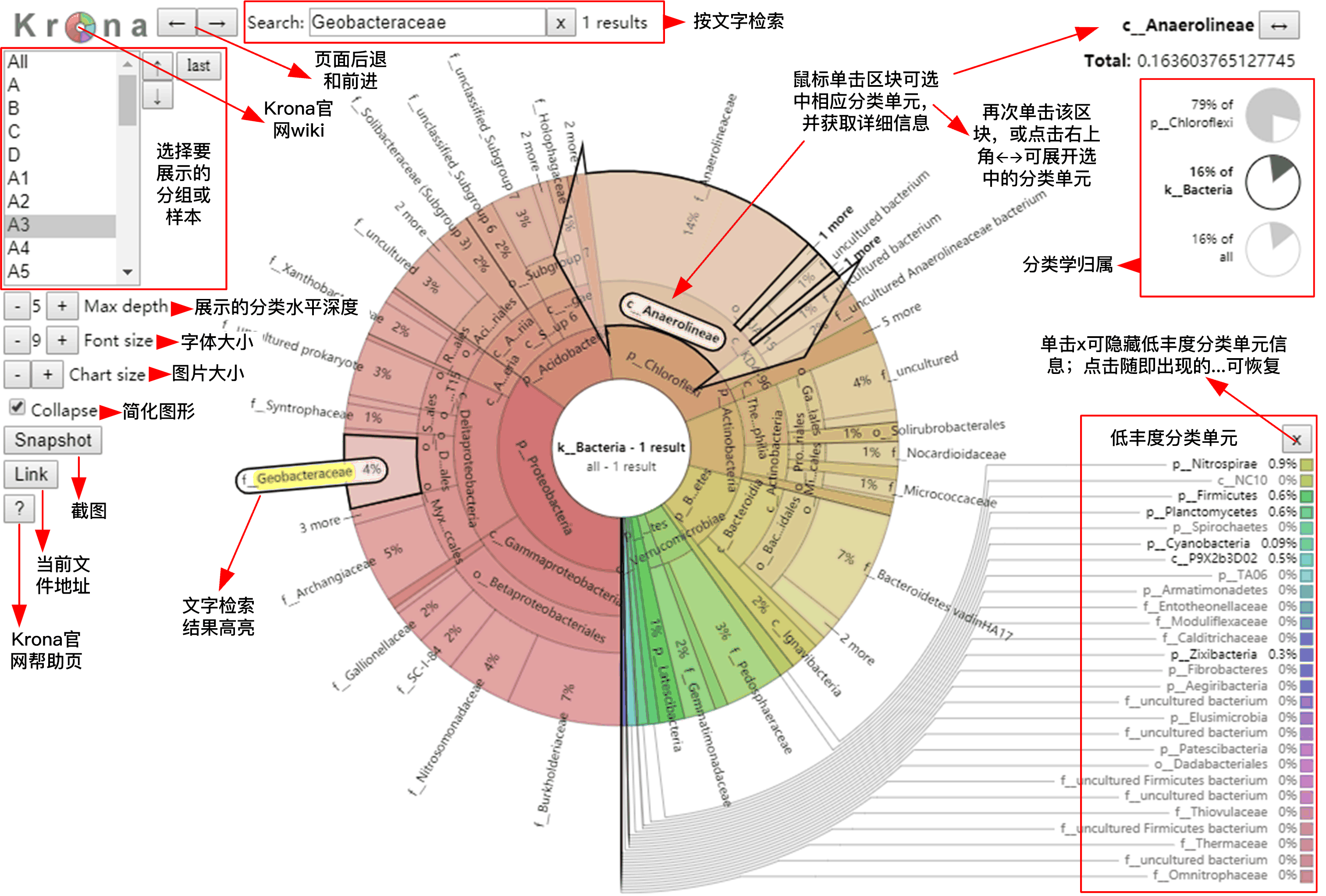
此外,我们也可以通过“Krona物种组成图”,对物种组成进行交互展示。与上述堆积柱状图不同,Krona分析是以圈图的形式展示,从内到外依次代表域、门、纲、目、科、属、种共7个分类水平,并可以点击进入对应分类水平的物种类群;扇形的大小反映了不同分类单元的相对丰度,各个分类单元以不同的颜色加以区分。
在Krona圈图展示页面的左上方,提供了多个图形可视化选项,可通过点击相应按钮调整图形的尺寸和显示的分类水平深度等;同时,点击图形中某一分类单元,将在圈图右上方显示其在各分类层次中所占的比例。
一般而言,Krona分析常用于比较各个分组的总体群落组成的差异,且能够通过圈图同时展现各个分类水平的物种组成特征。因此,可以作为堆积柱状图的替代方案,在研究报告中加以应用。
02、Alpha多样性分析
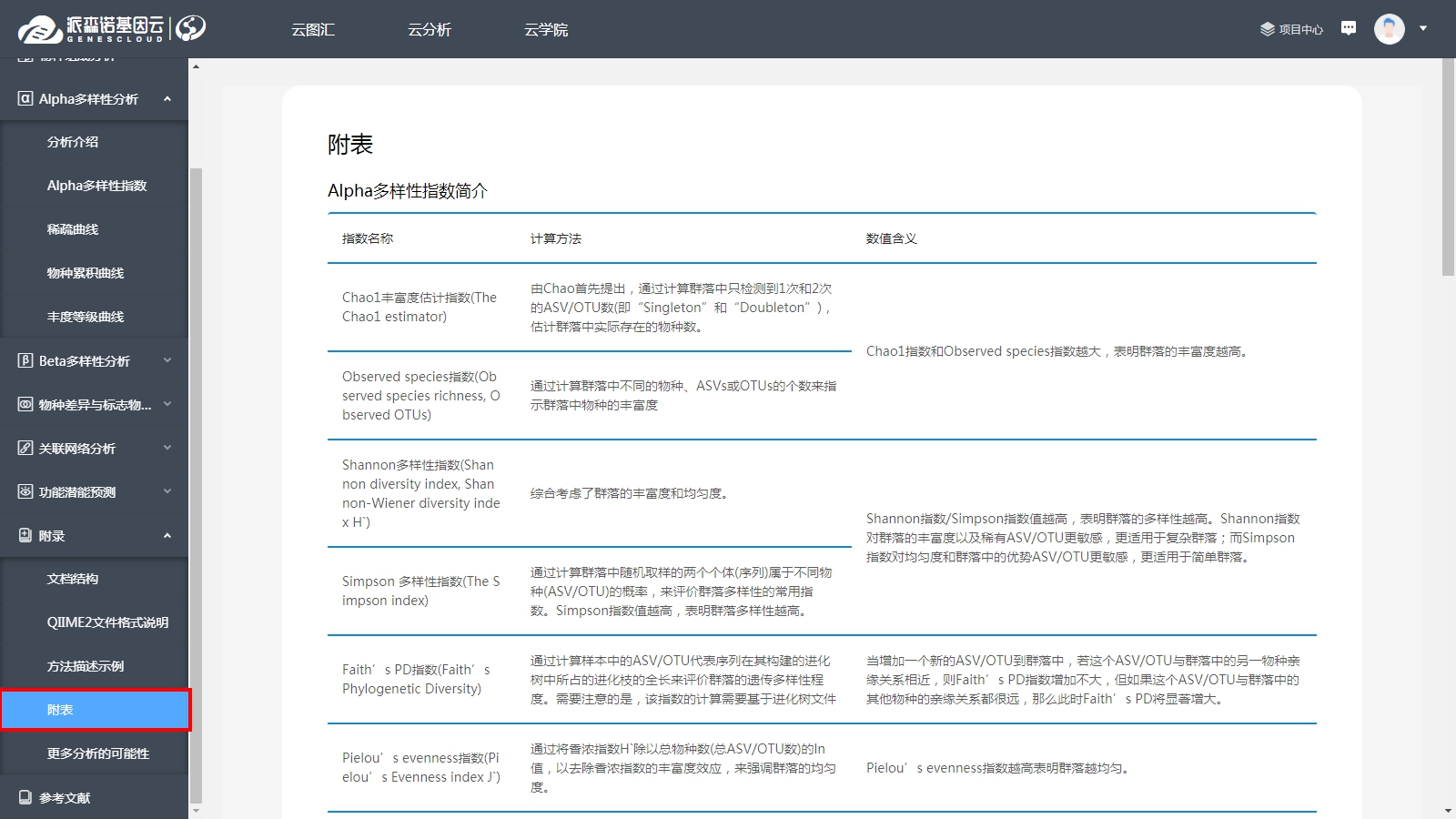
Alpha多样性是微生态学研究中的重要评价标准之一,主要通过Alpha多样性指数的大小,来衡量各个/各组群落样本中微生物物种的多样性、丰富度、均匀度、覆盖度等等。目前云分析支持7种常见Alpha多样性指数的计算,每种指数的具体意义与详细用法,可以通过“附录”进行查询。总体而言, Chao1和Observed species指数表征丰富度,Shannon和Simpson指数表征多样性,Faith’s PD指数表征基于进化的多样性,Pielou’s evenness指数表征均匀度,Good’s coverage指数表征覆盖度:
更详细的算法说明请参见:
http://scikit-bio.org/docs/latest/generated/skbio.diversity.alpha.html
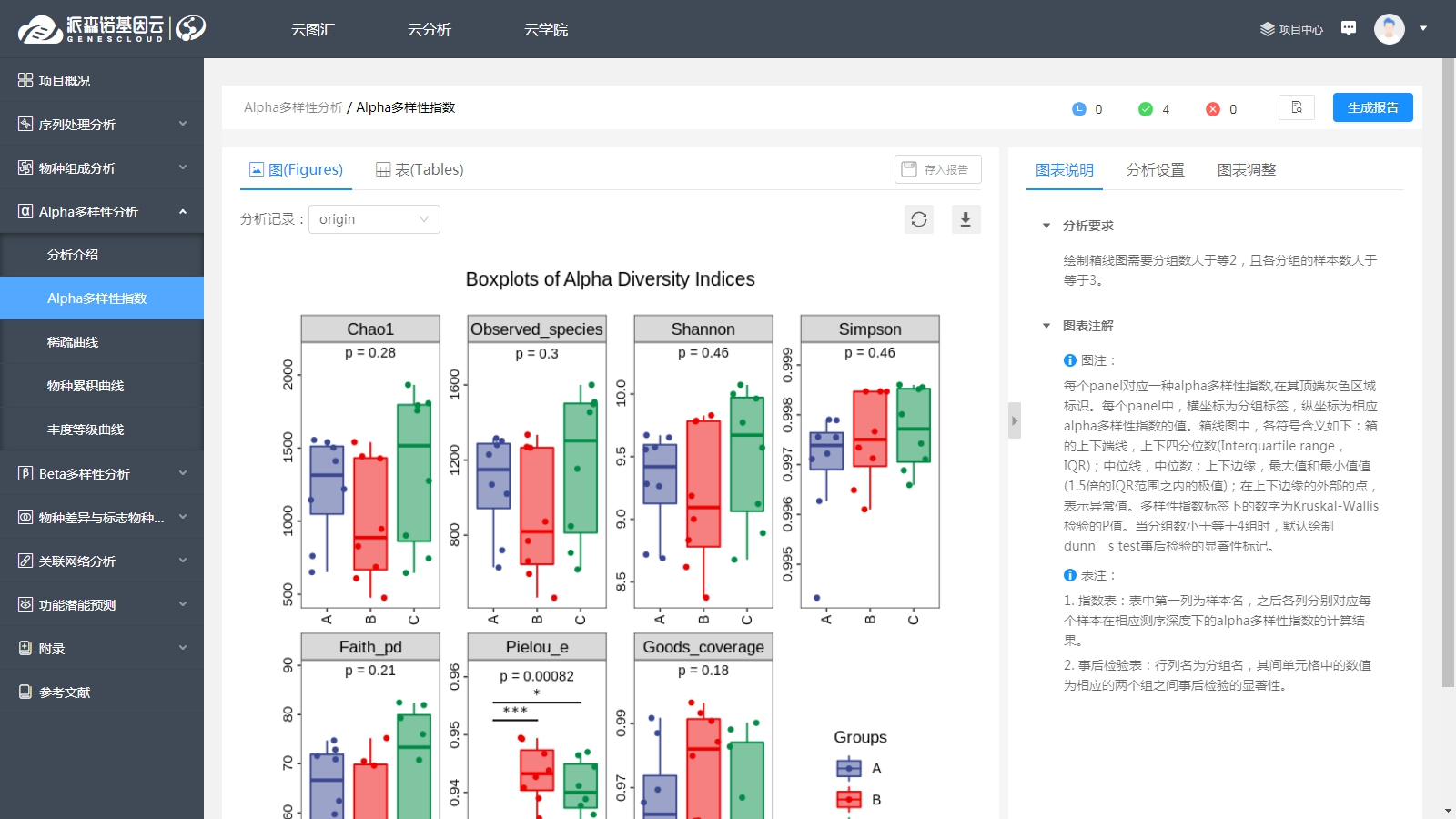
对于分析得到的每个群落样本的Alpha多样性指数,可以通过箱线图来展示比较组间Alpha多样性指数的差异大小。图中,每一个箱子代表一个分组,组与组之间的差异可以通过箱子内的中位线的高低来表征,中位线相差越大,则两组间的多样性差异越大。箱子上方的P值指示该多样性指数在所有分组之间是否具有显著差异;而箱子上方的直线,则指示直线两端所属的分组,是否具有显著差异,直线上的星号越多,显著性越强。
以上就是今天带给大家的云分析结果解读,后续我们会对Beta多样性分析、物种差异与标志物种分析等云分析结果,作系列解读哦~
欢迎大家进入基因云尝试使用,也欢迎大家在讨论区留言或者发邮件给我们哟(邮箱地址:metasupport@personalbio.cn)!


