2021-03-22

对于QIIME 2云分析,我们悉心整理了各个分析模块中的热点分析内容,推出了“5分钟小课堂系列”。此前已带来了第1讲(点击查看),对物种组成分析和Alpha多样性分析两大模块进行了解读。今天我们将对Beta多样性分析进行解读,希望可以帮助大家更好地理解微生物组测序分析的结果哦~
Beta多样性分析主要用于比较各样本中微生物群落整体结构的差异。通过QIIME 2云分析,我们共计算了4种常见的Beta多样性距离,每种距离可用于Beta多样性分析,但都有针对性的应用场景。对于这4种距离的介绍,可以参见“附录 / 附表 / Beta多样指数简介”:

具体的分析内容,主要包括PCoA分析、NMDS分析、层次聚类分析和组间差异分析:
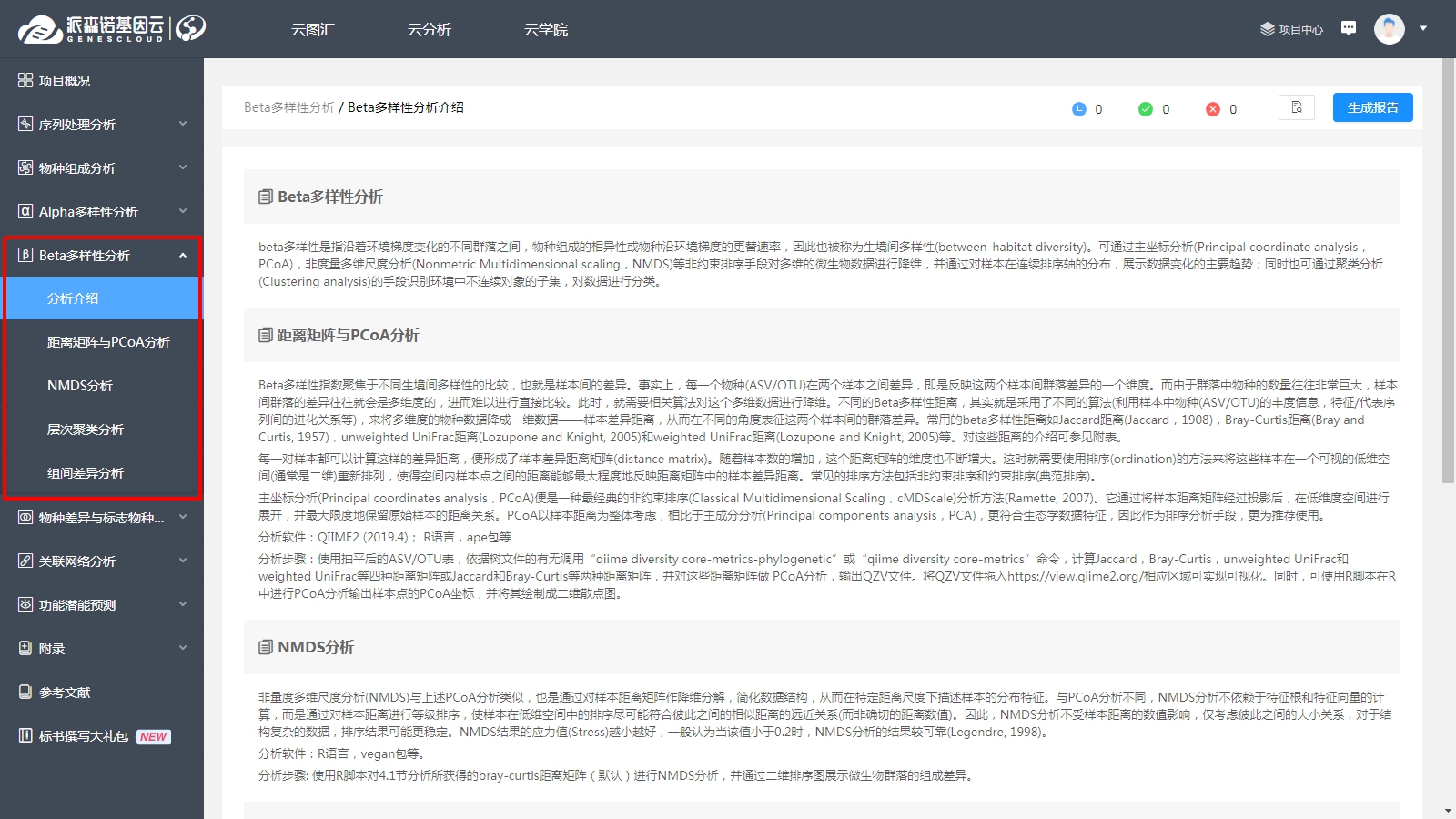
1、距离矩阵与PCoA分析
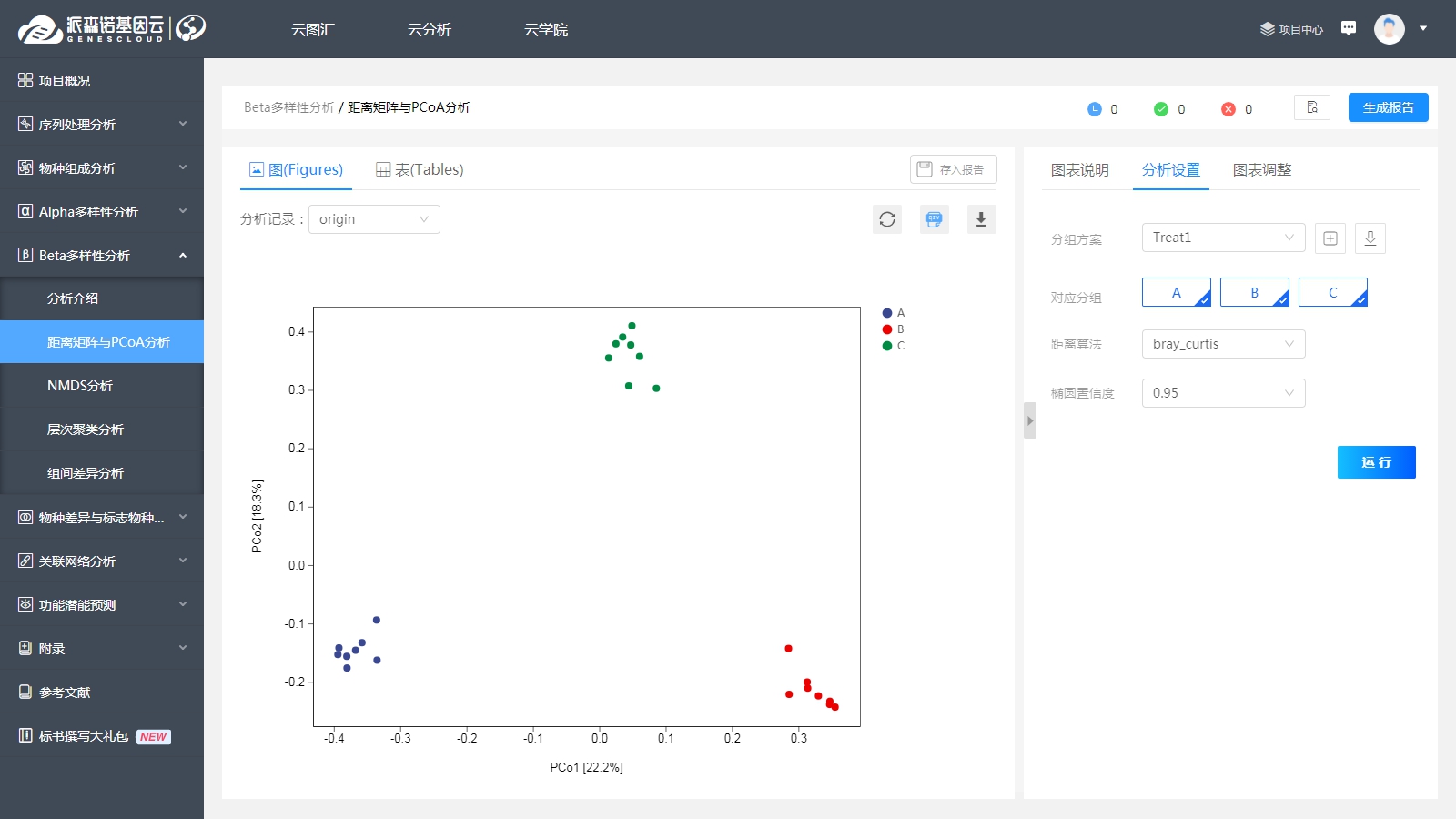
PCoA分析中即我们常见的主坐标分析,一般通过二维或三维散点图来进行展示。图中,每个点代表一个微生物群落样本,不同样本或分组用不同颜色/图形表示,点与点在坐标轴上的投影距离越远,则对应两个群落样本中的整体组成结构差异越大。同时,坐标轴括号中的百分比,代表了对应的坐标轴所能解释的样本差异数据(距离矩阵)的比例。
比如下图中A组与B组的群落样本,在PCoA1轴上的投影距离较远,但在PCoA2轴上的投影距离很近,表明两组群落样本在第一轴的维度上具有较大差异,但在第二轴的维度上却很相似;第一轴解释了约22.2%的样本间差异,而第二轴则解释了约18.3%的样本间差异。
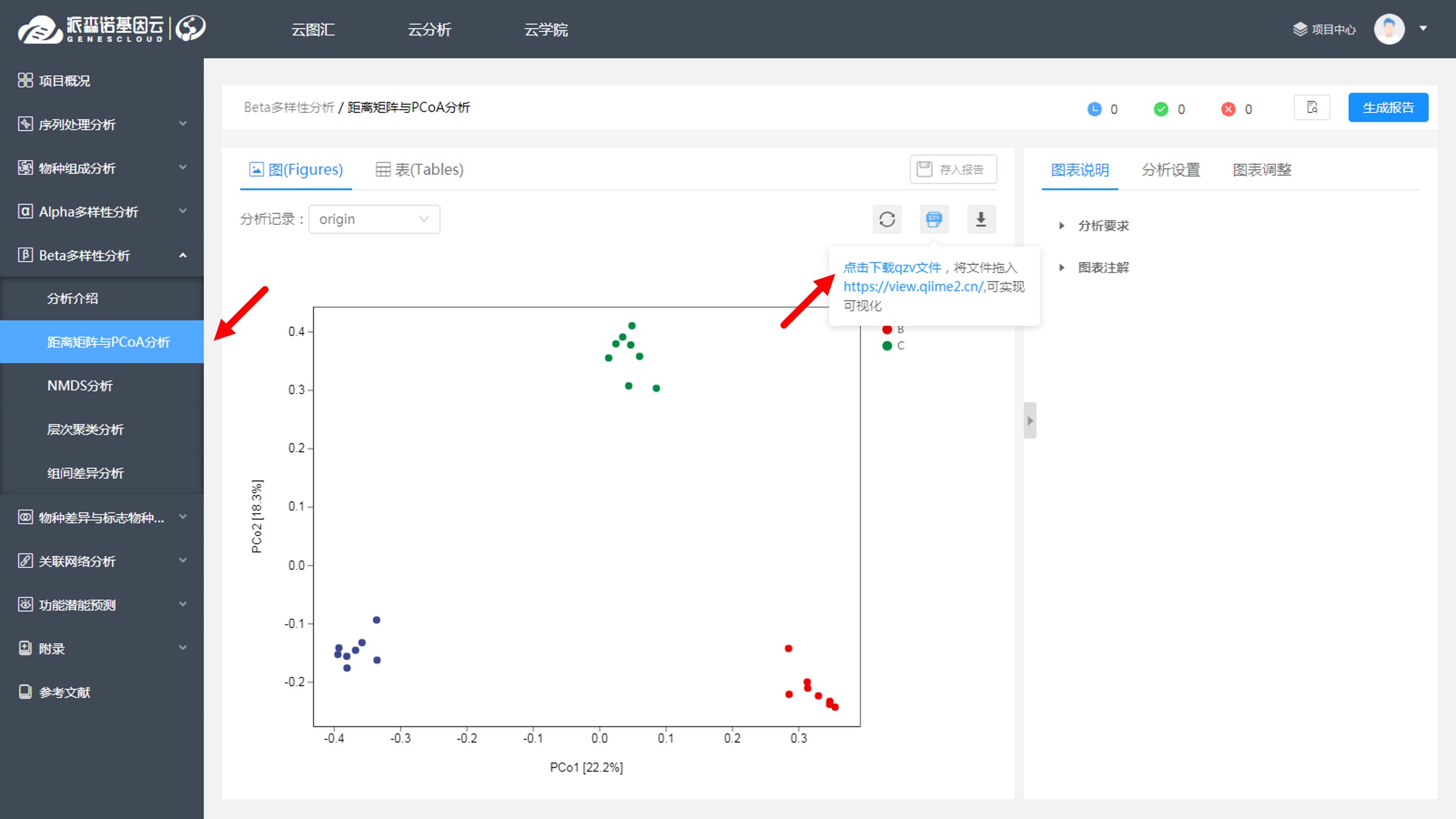
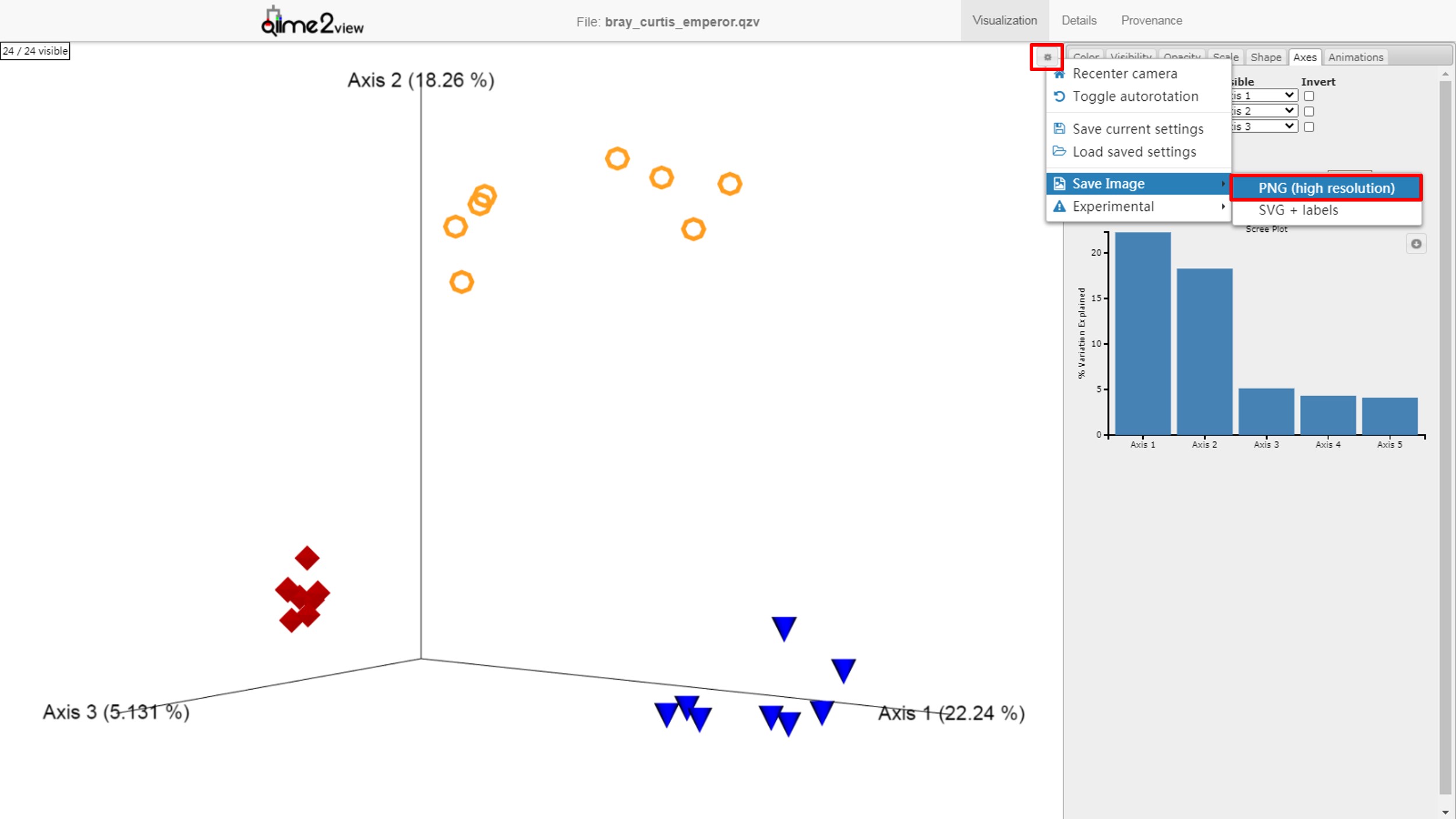
当然,我们也可以通过QIIME 2专有的QZV文件,在网站中(https://view.qiime2.cn/)实现三维可视化展示(具体操作可参见:【派森诺基因云】一文读懂QZV,一键玩转3D-PCoA!点击查看)。
2、NMDS分析
NMDS分析即非量度多维尺度分析,与PCoA相似,每个点代表一个群落样本,不同样本或分组用不同颜色/图形表示。与PCoA不同的是,NMDS分析不用参考投影距离,而是直接通过样本之间直线距离的远近,衡量群落组成结构的差异大小。因此,可近似认为两点之间的距离越近(远),表明两个样本中微生物群落的整体组成结构差异越小(大)。
另外,图片上方的Stress值表明分析结果的应力值,可用于衡量分析结果的可靠性。一般而言,当Stress值小于0.2时,NMDS分析的结果较可靠。

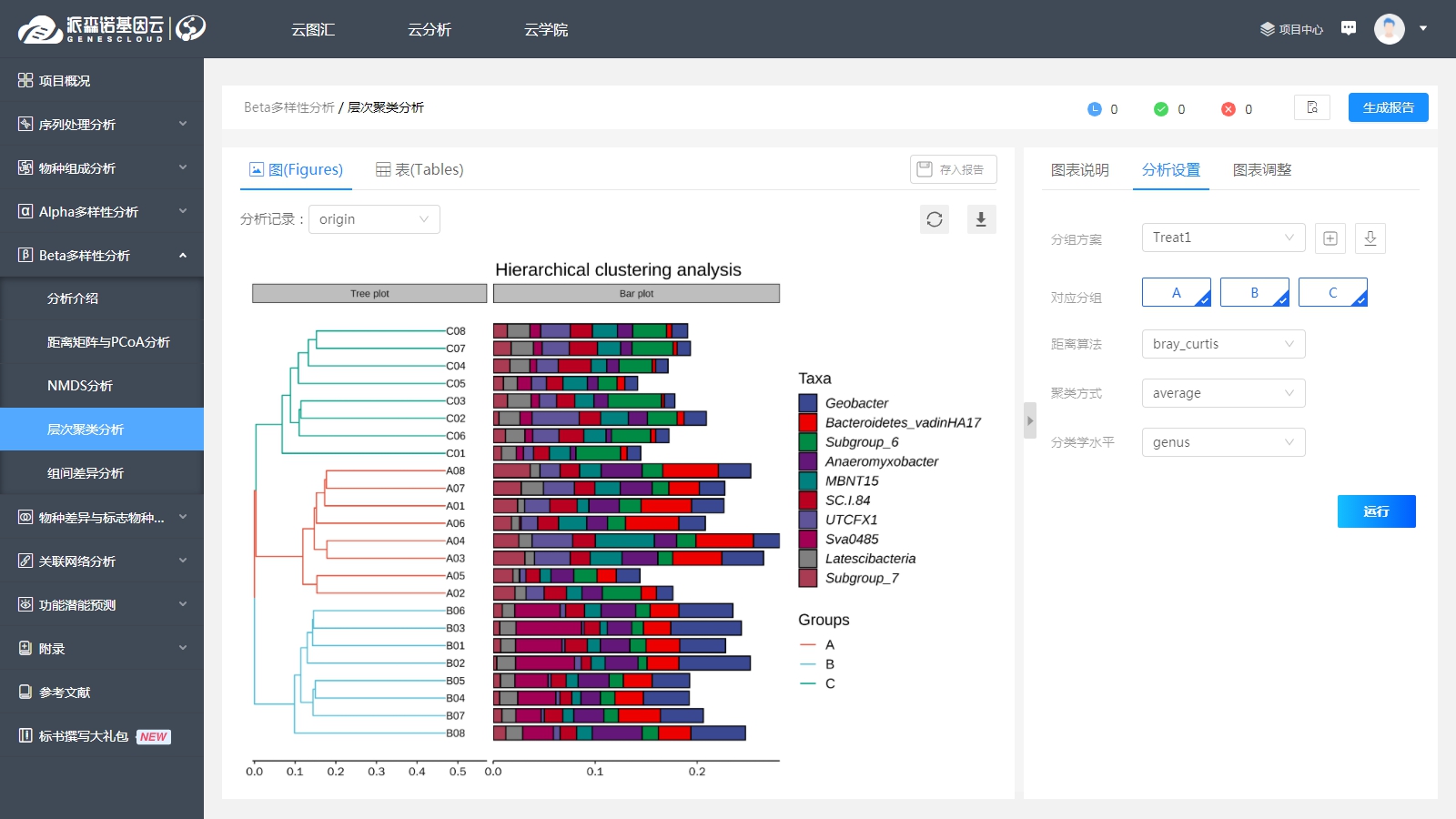
3、层次聚类分析
层次聚类分析由左右两部分组成:左图代表群落样本的层次聚类树图,右图代表物种组成的堆叠柱状图。
左图中,样本根据彼此之间的相似度聚类,样本间的分支长度越短、聚类效果越好,则两样本的群落整体组成结构越相似;右图默认显示丰度排名前10(或分析时自定义的个数)的属水平(或分析时自定义的分类水平)微生物组成,丰度越高,柱子长度越长。下图示例中,A、B、C三组样本分别聚类形成三个分枝,表明三组群落样本的整体组成结构有明显差异;同时,A组样本中,A02和A05样本,与组内其它样本的整体组成结构有较大不同,形成了独立的聚类分枝。而右图的物种组成,则从优势物种丰度分布的角度,进一步展现了上述的群落组成结构差异。
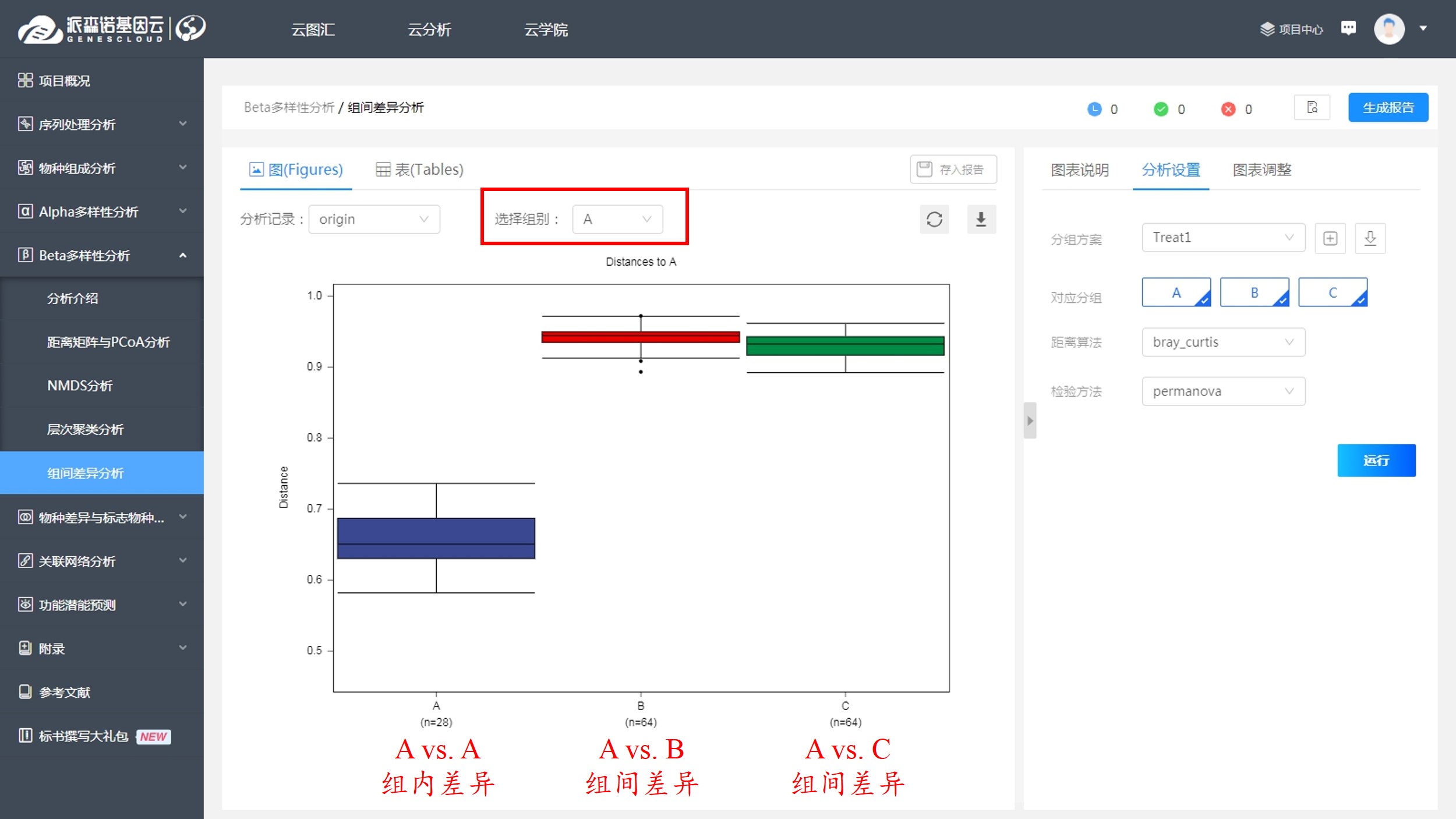
4、组间差异分析
组间差异分析用于比较组内差异与组间差异。具体而言,组内差异指的是指定分组内的群落样本之间的距离大小,组间差异是指该分组内的群落样本,与其余各分组的样本间的距离大小。距离值通过箱线图进行展示,以体现各组内/组间比较的差异大小。箱的上下端线代表上下四分位数(Interquartile range,IQR);箱中的横线代表中位数;箱外延伸的上下触须的边缘,对应1.5倍的IQR范围之内的极值(最大值和最小值);在触须边缘外部的点,表示离群的异常值。
图形上方可以指定组内/组间比较所需参考的分组。图中,第一个箱线图代表组内差异,中位线越低、触须越短,则表明组内差异越小、组内群落样本的重复性越好。其余箱线图则代表指定分组与其余各分组间的组间差异,中位线越高,表明组间差异越大(一般而言,一个理想的研究设计,应当是组内差异显著小于组间差异的)。
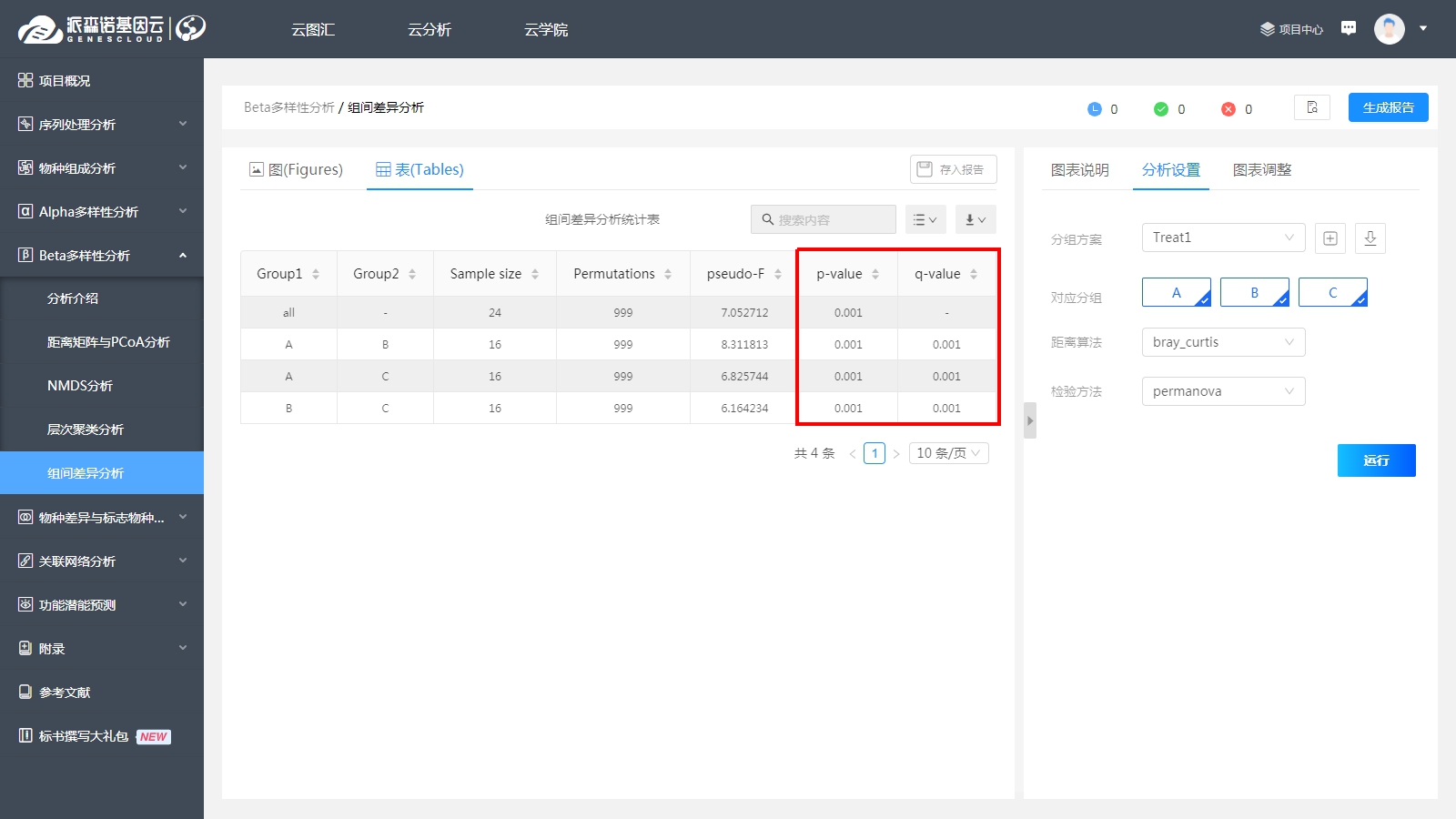
同时,我们也提供组间两两差异比较的P值与Q值,P值或Q值小于0.05时,一般认为有统计学意义的显著性。
以上就是今天给大家介绍的云分析结果解读哦~一般而言,不同的分析手段获得的结果,可能有所差异,因此还是建议大家可以多种方法都尝试比较,选择较为理想的分析结果进行展示。当然,分析结果和作图效果的好坏,很大程度上还是取决于微生物群落本身的特性哦~
欢迎大家进入基因云尝试使用,也可关注我们将上线的网络直播系列课程,在线互动交流哦!


