2021-06-25
对于QIIME 2云分析,我们悉心整理了各个分析模块中的热点分析内容,推出了“5分钟小课堂系列”(点击查看)。今天开始,我们将带来微生物组研究中的热门分析方法——关联网络分析的讲解,带您从入门走向精通!

提到关联网络分析,大家首先想到的,可能只是一张张网络图,比如:
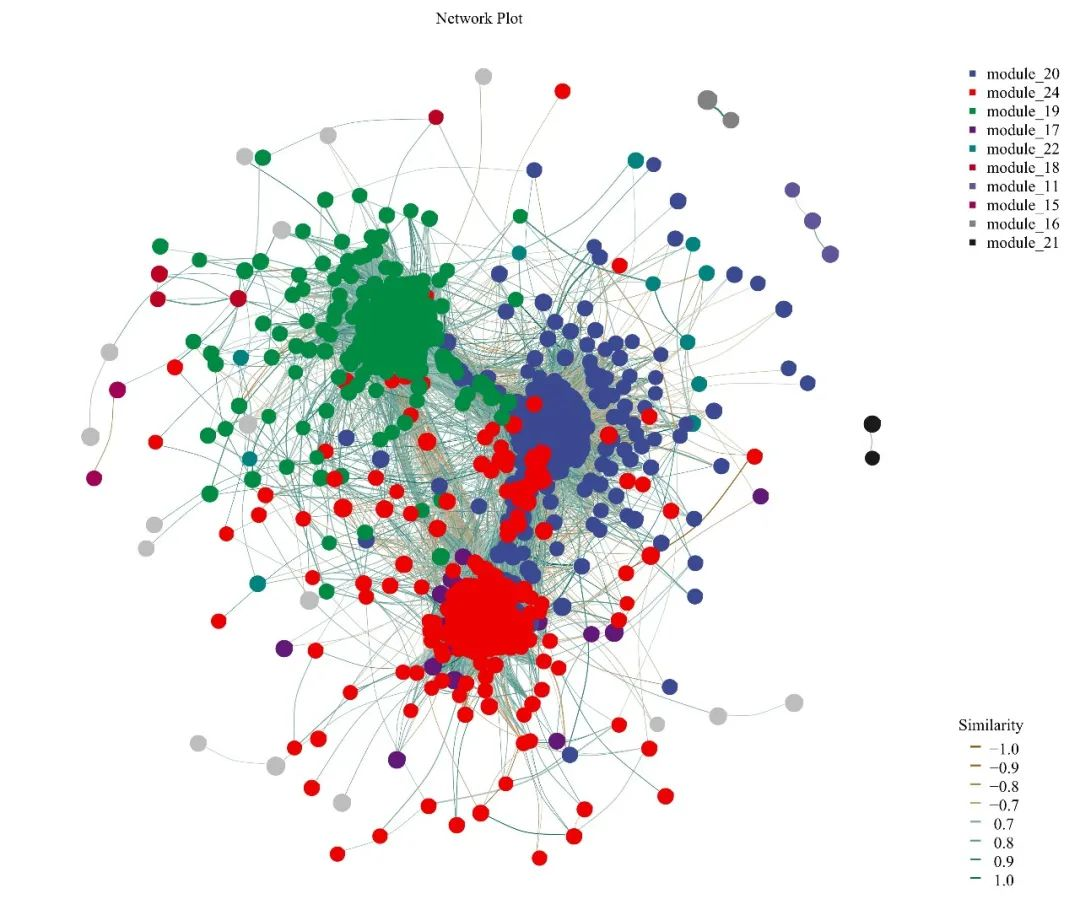
实际上,网络图确实是关联网络分析结果的一种直观展示形式,也是在论文中“出镜率”较高的。但是,网络图展示了关联网络分析得到的一部分内容,相当于“门面”,看上去比较酷炫,实则背后还有很多信息量有待深入挖掘哦!
知识点1
什么是关联网络分析?
关联网络分析主要用于寻找特定微生物群落在时空变化,环境过程驱动下所呈现的共现(Co-occurrence)或互斥(Co-exclusion)的固有模式,从而分析环境差异或实验处理是否导致了群落物种装配的差异,探究微生物群落中是否存在特定的模块单元(Module)以完成特定的生态功能,并据此寻找足以撬动整个群落的组成变化的关键物种(Hubs或keystone species)。
知识点2
什么是模块单元(Module)?
模块单元是指在网络中,相较于其余节点,关系更紧密复杂的节点相互之间组成的一系列结构,通常认为,同一模块的微生物在生存或功能上关系可能更紧密。
关联网络分析主要涉及5个环节:
1. 构建关联网络
2. 绘制关联网络
3. 计算拓扑指数
4. 评估度分布模式
5. ZiPi分析——关键物种查找
今天,我们先来讲解关联网络的构建和绘制,即识别微生物之间的相互关系,筛选可靠的相关性结果。
1. 构建关联网络
在关联网络的构建上,我们选用目前公认的成熟的SparCC算法(SparCC优于Spearman、Pearson等简单计算相关性的算法,更适合生态学数据),根据微生物群落的组成数据,计算群落成员之间的相关性。但初步的计算结果中,往往会有一些随机噪声干扰,阻碍我们挖掘有生物学意义的相关值,因此,初步的相关性结果需要经过筛选,将有生物学意义的相关性筛选出来。为达成这一目的,我们选用随机矩阵理论(RMT)来确定相关性阈值,结果如下图:
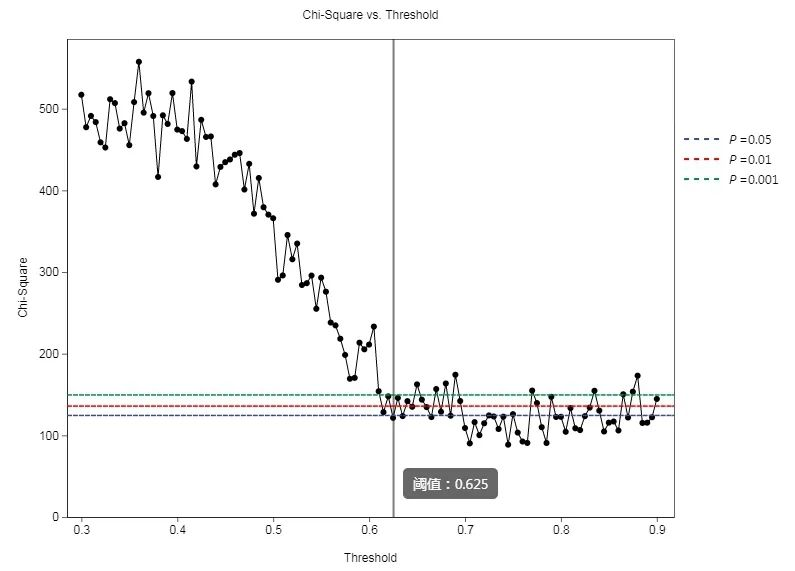
图中,X轴代表相关性阈值(r),y轴代表卡方检验值(P),竖线与X轴的交叉处,即筛选得到的相关性阈值。比如上图,后续构建相关性网络,便筛选相关性系数(r)绝对值在0.625以上的相关性进行绘图(若图中没有竖线,则以第一个低于虚线的点为阈值,虚线优先级:蓝线>红线>绿线;若既没有竖线,也没有低于虚线的值,则以0.6为阈值)。
2. 绘制关联网络
根据第一步筛选后的相关性数据,我们就能绘制关联网络图啦~绘制成功的示例图说明如下:
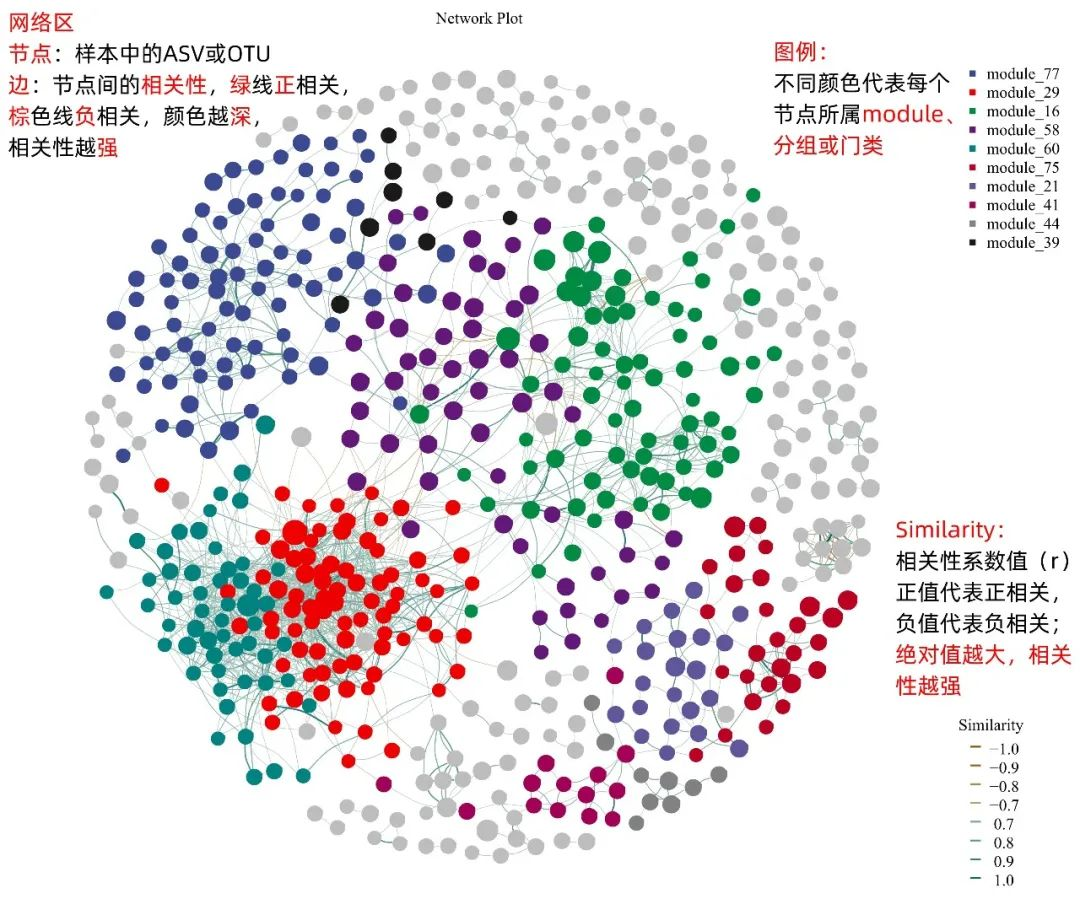
默认情况下,基因云中的关联网络图是基于丰度前100的ASV或OTU绘制的,如果想掌握更完整的关联分析结果,可以将分析设置 | 丰度数目的数值调大。比如,上述例图展示的,便是丰度数目调大至1000的结果。
另外,网络图的颜色、注释方式等都可以在图表调整部分进行修改哦~我们还可以将上述按照模块单元来展示的网络图,改变为按照物种组成或分组来着色、注释和展示~
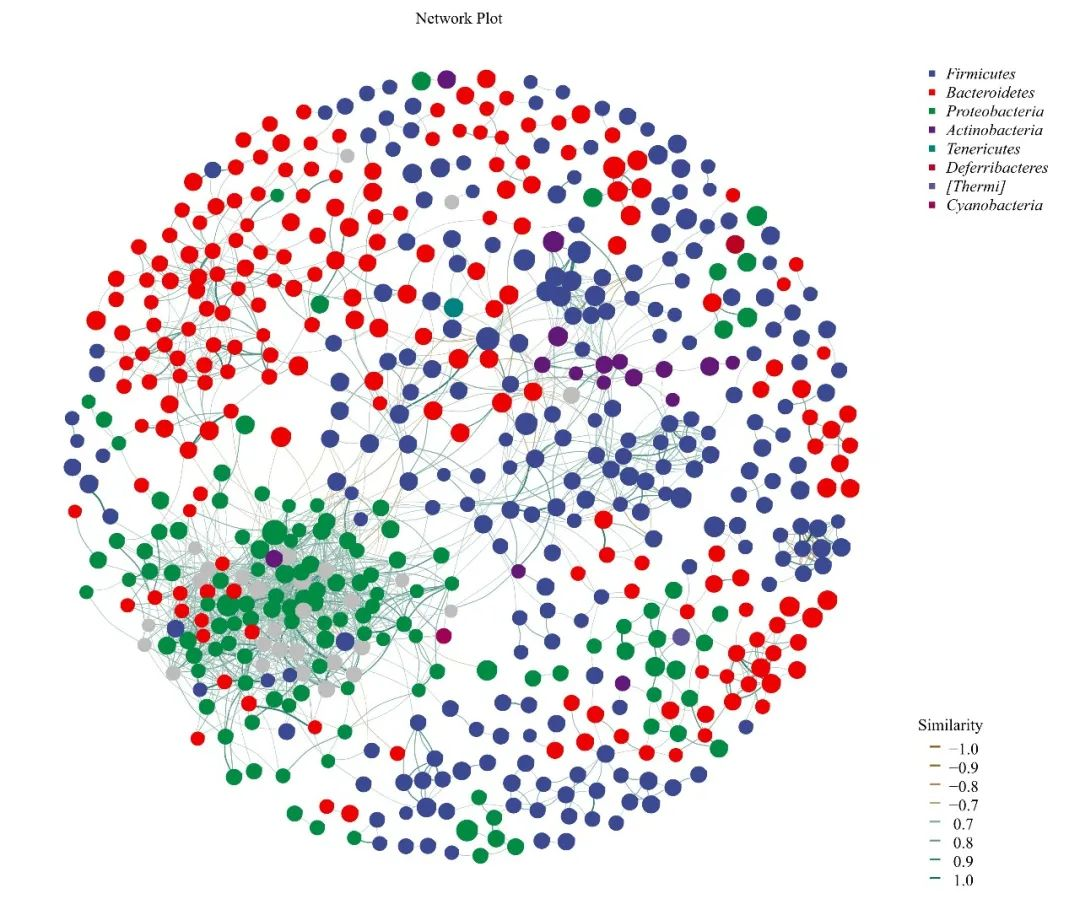
按门水平物种着色和注释的关联网络图,注意节点和边的关系是不会改变的
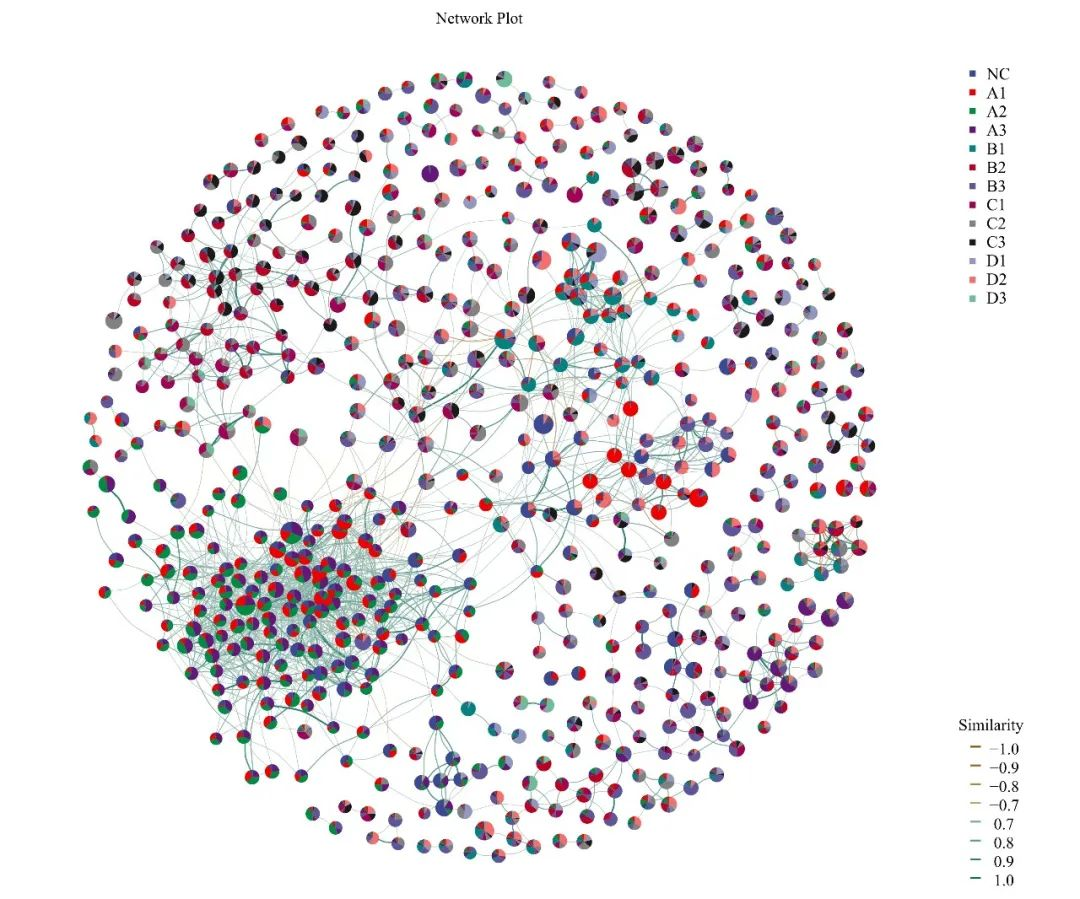
按分组着色和注释的关联网络图,注意节点和边的关系是不会改变的
如果想要自行绘制更加个性化的关联网络图,也可以通过基因云下载gml文件,导入gephi或Cytoscape软件中,按照需要自行绘制。
关联网络图绘制成功后,我们根据图中展示的内容,即可分析整个微生物群落中的成员互作关系。比如,根据模块化着色的网络图结果,可探究微生物类群间是否存在特定的模块单元能完成特定的生态功能;根据物种着色的网络图,可用于分析具有相关性的物种主要属于哪些分类单元等等。
以上就是本次的QIIME 2云分析内容讲解哦,下次我们将继续带来网络拓扑指数的讲解,敬请关注哦!我们也衷心欢迎大家进入派森诺基因云尝试使用哦!


