
2021-07-02
上一篇10x单细胞数据分析我们介绍了如何对单细胞进行表达量分析,这一篇我们介绍一下单细胞分析流行的R包Seurat的分析步骤。
在开始分析之前,首先需要安装最新版的R(https://cran.r-project.org/bin/windows/base/)和Rstudio(https://www.rstudio.com/products/rstudio/download/#download)。
打开Rstudio,需要安装Seurat包,在控制台中输入:
install.packages("Seurat")安装好以后,先将需要的包进行加载:
library(Seurat) library(dplyr) library(magrittr)
测试数据选择10X Genomics 免费提供的外周血单核细胞 (PBMC) 数据集,约有2700个细胞(https://cf.10xgenomics.com/samples/cell/pbmc3k/pbmc3k_filtered_gene_bc_matrices.tar.gz)。下载好以后需要进行解压。从压缩包的名字中我们可以看到,其实就是上一篇cellranger分析的结果中的filtered_gene_bc_matrices文件夹。测试数据的细胞数较少,普通的台式机就可以分析。小编在测试的时候,分析完大约花了10分钟,只用1G左右的内存,所以大家也可以在自己电脑上试着分析一下哦!
1、数据读入
Seurat需要的输入信息为表达量矩阵,矩阵行为基因,列为细胞。使用Seurat提供的Read10X函数可以很方便的将10x结果读入到R矩阵中。使用CreateSeuratObject生成Seurat对象,后续分析都是在该对象上进行操作。
pbmc.data <- Read10X("C:/Users/my/Desktop/pbmc3k_filtered_gene_bc_matrices/filtered_gene_bc_matrices/hg19/")
pbmc <- CreateSeuratObject(counts = pbmc.data, project = "pbmc3k", min.cells = 3, min.features = 200)这里的min.cells=3表示从表达量矩阵中删除在少于3个细胞中表达的基因(行),min.features=200表示删除表达的基因数小于200的细胞(列)。
2、质控和过滤
质控的目的是去除掉低质量的数据,包括破损或死亡的细胞、没捕获到细胞的empty droplet和捕获到2个以上细胞的doublets。一般低质量的细胞或者empty droplet通常含有很少的基因,而doublets容易测到更多的基因。另一方面,低质量或者死亡细胞会测到更多的线粒体基因表达的RNA。
使用PercentageFeatureSet函数评估每个细胞中的线粒体表达比例:
pbmc[["percent.mt"]] <- PercentageFeatureSet(pbmc, pattern = "^MT-")
pattern = "^MT-"表示线粒体基因的匹配模式,人的线粒体基因以MT-为前缀,小鼠的以Mt-为前缀。
Seurat对象在生成时,已经将每个细胞表达的基因数和reads数记录在meta.data的nFeature_RNA和nCount_RNA中。我们使用VlnPlot函数将nFeature_RNA、nCount_RNA和percent.mt三个指标进行可视化:
VlnPlot(pbmc, features = c("nFeature_RNA", "nCount_RNA", "percent.mt"), ncol = 3)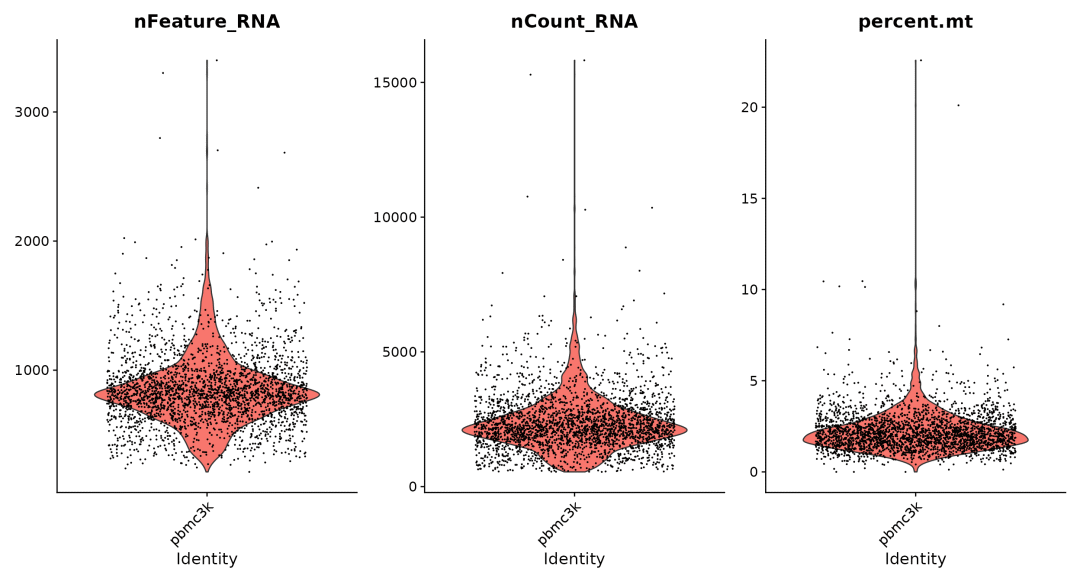
根据小提琴图的数值分布情况确定筛选的阈值。这里,我们以 200 < nFeature_RNA < 2500 且percent.mt < 5为条件对数据进行筛选:
pbmc <- subset(pbmc, subset = nFeature_RNA > 200 & nFeature_RNA < 2500 & percent.mt < 5)
3、标准化
基因的表达矩阵需要经过标准化后才能进行后续的分析。使用NormalizeData函数进行标准化,方法选择LogNormalize:
pbmc <- NormalizeData(pbmc, normalization.method = "LogNormalize", scale.factor = 10000)
4、挑选高变基因
接下来需要挑选出在细胞间高变异的基因,这些基因有助于突出单细胞数据中的生物学信息。使用FindVariableFeatures函数来挑选高变基因,默认的情况下,挑选2000个。
pbmc <- FindVariableFeatures(pbmc, selection.method = "vst", nfeatures = 2000)
5、数据降维(PCA)
在PCA之前,需要讲数据进行归一化:
all.genes <- rownames(pbmc) pbmc <- ScaleData(pbmc, features = all.genes)
归一化后的数据进行PCA分析。默认的,只用前面挑选的高变基因进行PCA分析,也可以使用features参数设置自己选择的基因进行PCA分析。
pbmc <- RunPCA(pbmc, features = VariableFeatures(object = pbmc))
6、聚类分析
在PCA分析后,就要根据PCA结果来进行聚类分析了。但是在进行聚类分析之前,需要选择使用对少个主成分进行计算。每个主成分实际上代表的是相关基因集的信息,因此确定多少个主成分是一个重要的步骤,我们可以根据ElbowPlot函数来判断。
pbmc <- JackStraw(pbmc, num.replicate = 100) pbmc <- ScoreJackStraw(pbmc, dims = 1:20) ElbowPlot(pbmc)
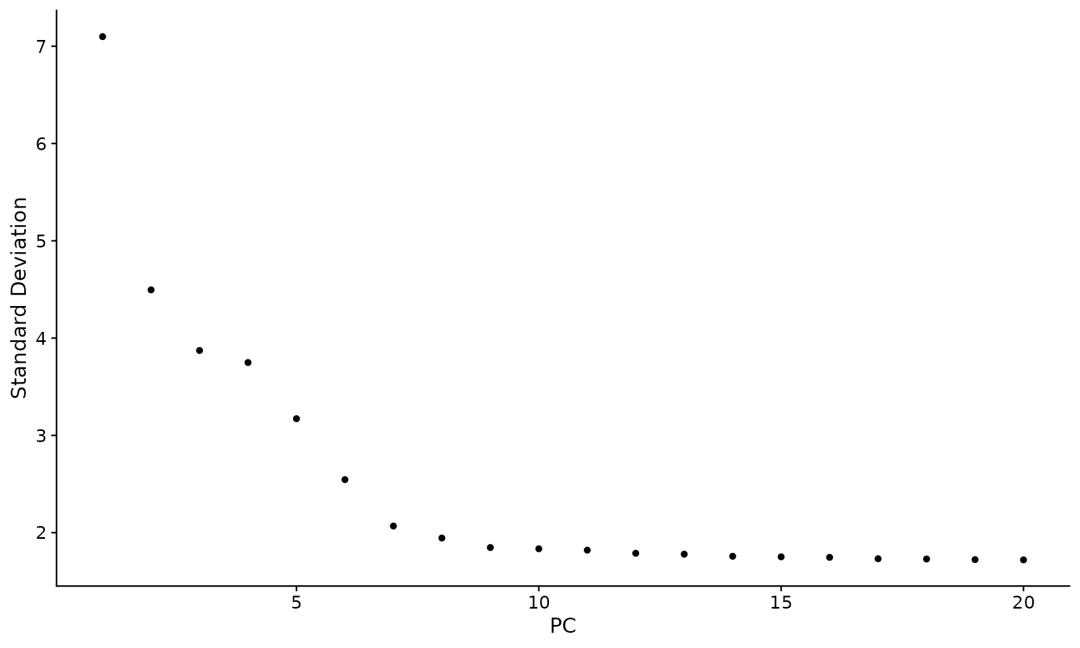
通常我们会将图中拐点所对应的值作为筛选的阈值,本例中我们选择前10个PC来进行聚类分析(通常使用更高的PC对结果的影响并不大,但是使用较小的PC会对结果产生不利的影响)。
Seurat基于 PCA 空间中的欧几里德距离构建一个 KNN 图,并根据其局部邻域中的共享重叠优化任意两个单元之间的边权重,基于Louvain算法对细胞进行聚类分群。
pbmc <- FindNeighbors(pbmc, dims = 1:10) pbmc <- FindClusters(pbmc, resolution = 0.5)
resolution表示分群的分辨率,更高的值会分出更多的细胞群。
7、非线性降维
Seurat提供几个非线性降维方法,如UMAP和tSNE,来可视化和探索单细胞数据。这些算法是通过学习数据的底层流形,来将细胞放到二维空间中进行展示:
pbmc <- RunUMAP(pbmc, dims = 1:10) pbmc <- RunTSNE(pbmc, dims = 1:10)
这一步并不会影响细胞的分群结果,所以也可以在分群前运行。可以通过DimPlot函数对非线性降维的结果进行展示:
umap.plot<-DimPlot(pbmc, reduction = "umap", label=T) tsne.plot<-DimPlot(pbmc, reduction = "tsne", label=T) umap.plot+tsne.plot
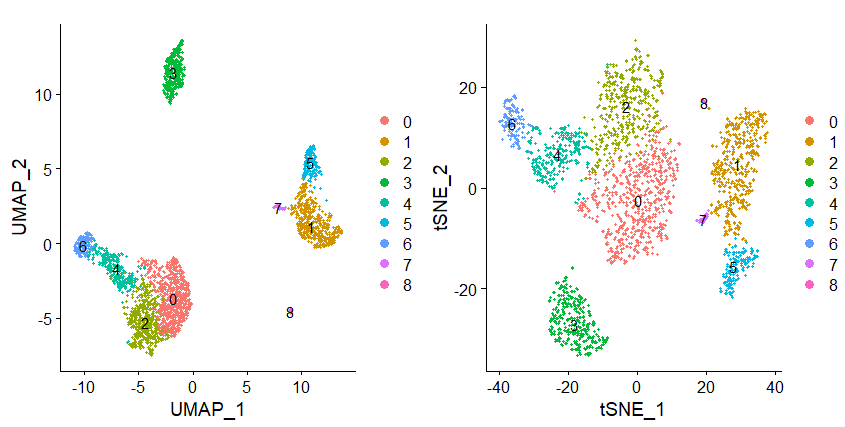
8、鉴定细胞群间Marker基因
为了鉴定各个细胞群对应的细胞类型,我们需要找出每个细胞群相对高表达的基因。使用Seurat的FindAllMarkers函数分别计算每个亚群与剩下的所有细胞的差异性,通常只保留高表达的基因作为marker:
pbmc.markers <- FindAllMarkers(pbmc, only.pos = TRUE, min.pct = 0.25, logfc.threshold = 0.25)
当然,对于PBMC这种已知细胞类型marker的数据,也可以直接使用已知的marker。以Seurat官方提供的PBMC marker为例:
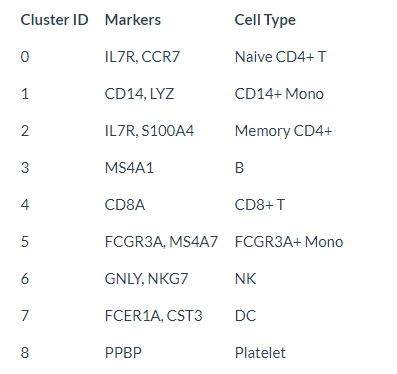
对这些marker进行可视化:
VlnPlot(pbmc, features = c("MS4A1", "CD79A"))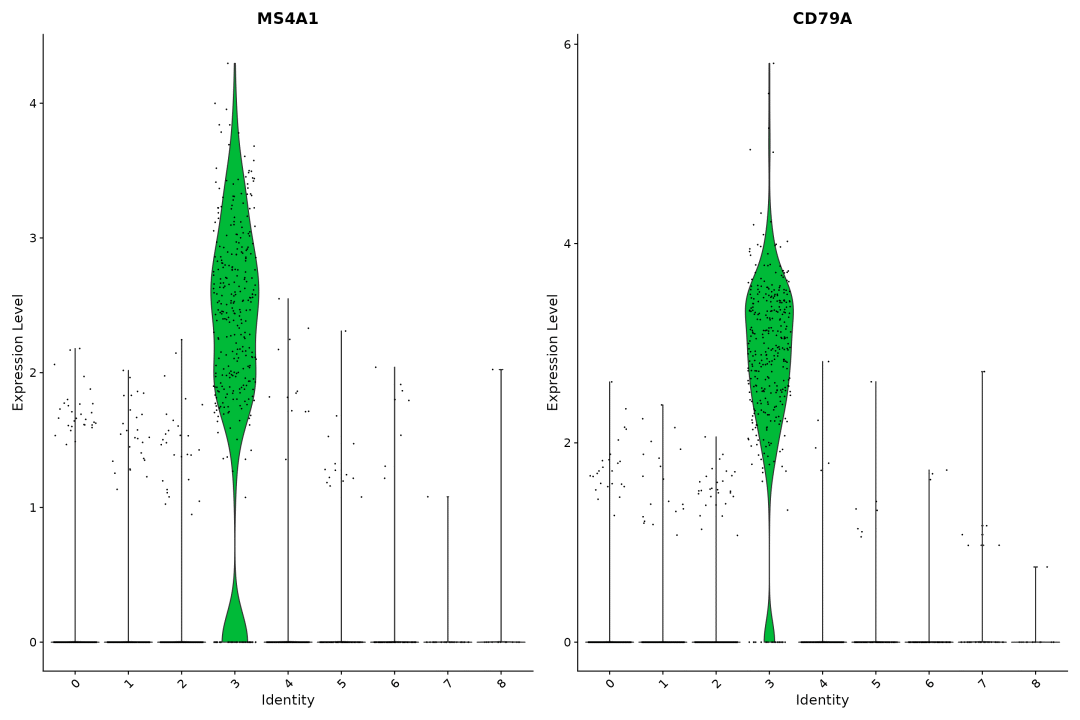
FeaturePlot(pbmc, features = c("MS4A1", "GNLY", "CD3E", "CD14", "FCER1A", "FCGR3A", "LYZ", "PPBP", "CD8A"))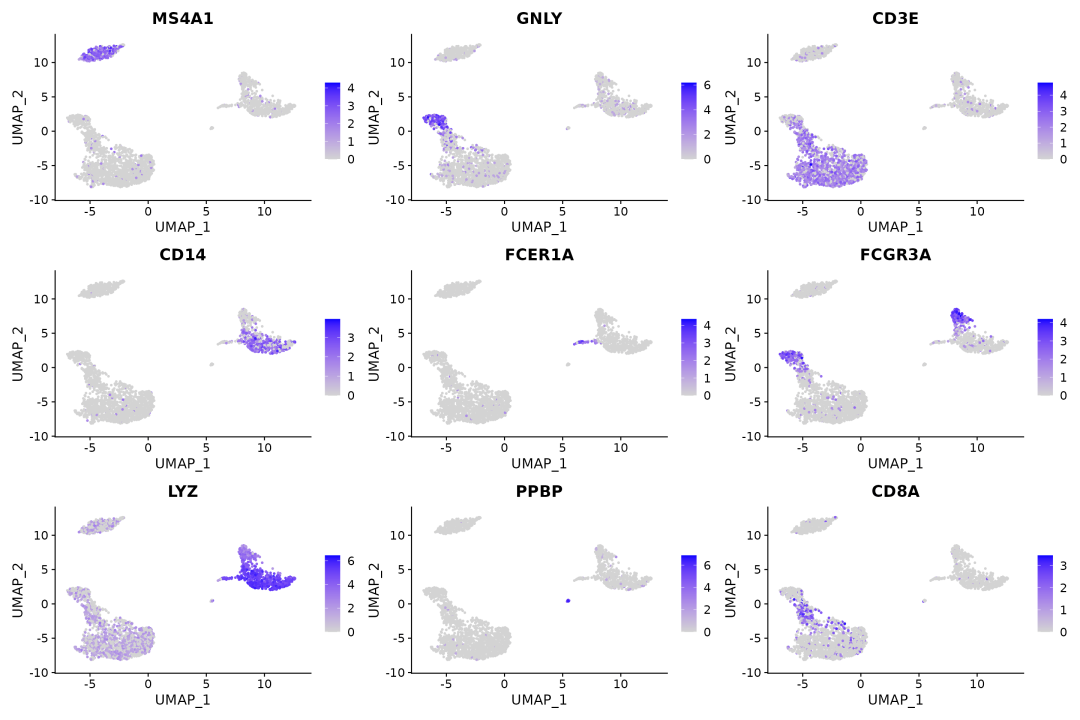
根据marker基因的展示结果可以对细胞群赋予正式的细胞类型
new.cluster.ids <- c("Naive CD4 T", "CD14+ Mono", "Memory CD4 T", "B", "CD8 T", "FCGR3A+ Mono",
"NK", "DC", "Platelet")
names(new.cluster.ids) <- levels(pbmc)
pbmc <- RenameIdents(pbmc, new.cluster.ids)
DimPlot(pbmc, reduction = "umap", label = TRUE, pt.size = 0.5) + NoLegend()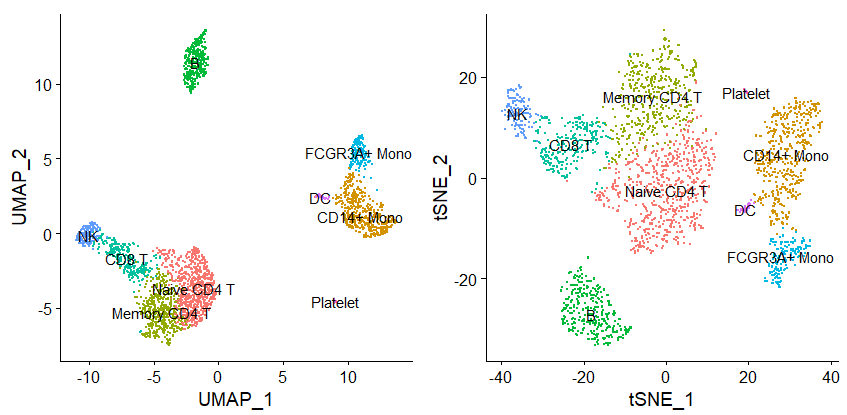
当然,细胞类型鉴定也有一些软件可以进行分析,如SingleR、Cell-ID等,后续的单细胞分析请关注派森诺生物官网