2021-10-26
连锁不平衡(linkage disequilibrium, LD)分析是群体遗传学研究中常见的分析内容,也是关联分析的基础,在很多的GWAS文章中都会出现LD衰减图及单倍型block图,接下来一起连锁不平衡(linkage disequilibrium, LD)初探。
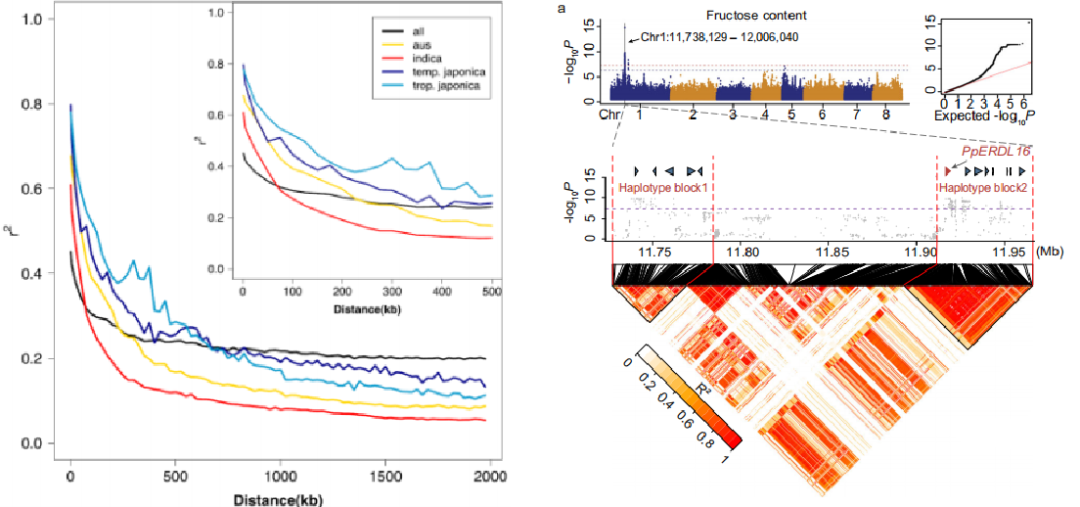
图1 水稻自然群体连锁不平衡衰减与桃果糖含量位点相关单倍型区块
1、LD的概念
当位于某一座位的特定等位基因与另一座位的某一等位基因同时出现的概率大于群体中因随机分布的两个等位基因同时出现的概率时,称这两个座位处于连锁不平衡状态。
2、LD的计算方法与度量指标
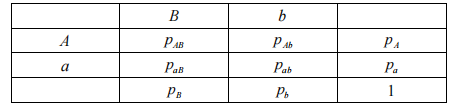
2.1 D值的计算
LD的基本单位是D值,度量观察到的单倍型频率与平衡状态下期望频率的偏差。D值根据单倍型频率必≥0,计算取值范围为[-0.25,0.25]。
D=Pr(A,B)-Pr(A)×Pr(B)
=PAB-PAPB
=PAB-(1-Pa)(1-Pb)
=PAB-(1-Pa-Pb+PaPb)
=PAB-(PA-Pb+PaPb)
=PAB-[PAB+PAb-(PAb+Pab)+PaPb)]
=PAB-(PAB-Pab+PaPb)
=Pab-PaPb
2.2 标准化指标:D’和r2
由于D值严格依赖于等位基因频率(allele frequency),故不适合应用于表述实际的LD强度,最常用度量LD的是D’和r2,两者都基于D。D’反应群体的重组历史,适用于研究群体连锁不平衡程度,r2反应等位基因相关程度,适用于GWAS。LD衰减作图中通常采用r2来表示群体的LD水平;Haplotype Block中通常采用D'来定义Block;迁移、突变、选择、有限的群体大小以及其他引起等位基因频率改变的因素,这些都会引起LD变化,下面尝试计算一下LD吧~
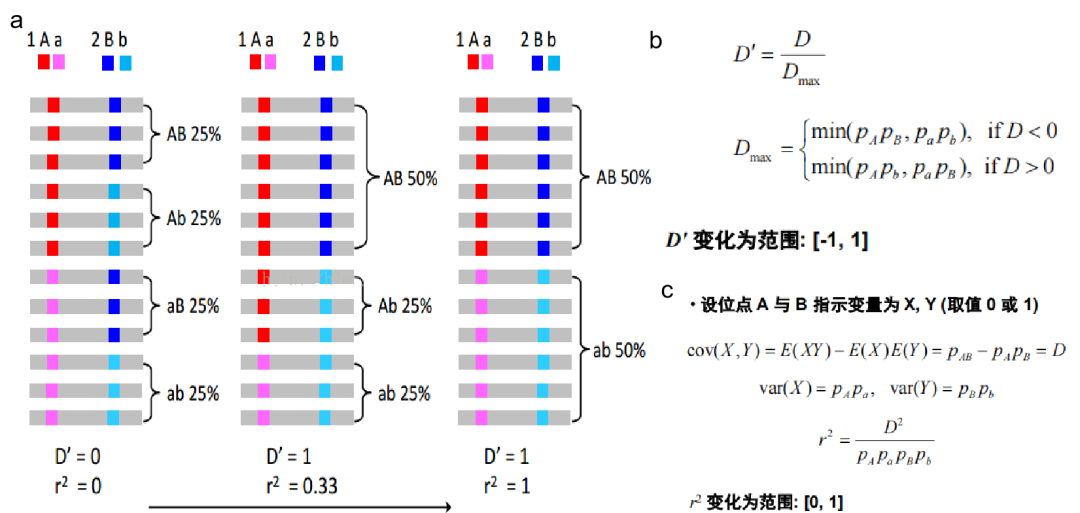
图2 D’和r2的计算
小Tips:
当D'=0,r2=0时,处于完全连锁平衡状态
当D'=1,r2=1时,处于完全连锁不平衡状态。
其中,从0—1之间的度量越高,LD越高,如果两个位点连锁,连锁程度也越高。
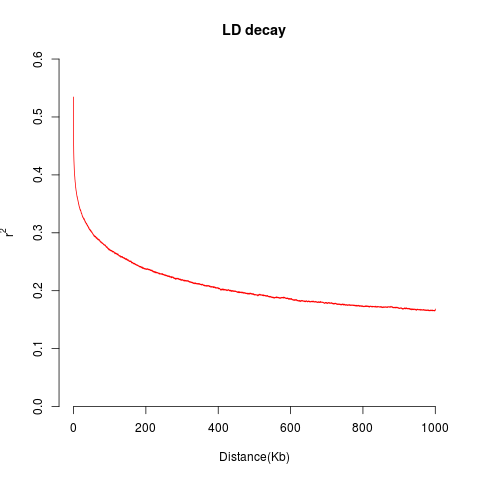
3、LD衰减
LD衰减指位点间由连锁不平衡到连锁平衡的演变过程;LD衰减的速度在不同物种间或同物种的不同亚群间,差异非常大。所以,通常会使用“LD衰减距离”来描述LD衰减速度的快慢,不同文章中“LD衰减距离”标准不同,常见的标准有:LD系数降低到最大值的一半、LD系数降低到0.5以下、LD系数降低到不同物种的基线水平等,我们在阅读文献时有必要留意文章使用的标准~
LD衰减距离在群体遗传学中的应用也非常广泛,一方面可以判断GWAS所需标记量,决定GWAS的检测效力以及精度;另外也可以辅助分析进化与选择,在同一个连锁群上,LD衰减慢说明该群体受到选择,一般来说,野生群体比驯化改良群体LD衰减快,异花授粉植物比自花授粉植物LD衰减快。
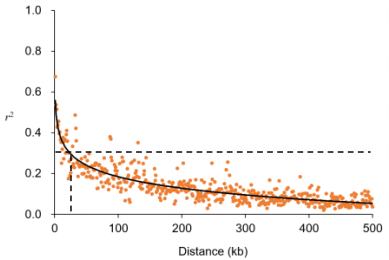
图3 LD下降到最大值一半对应的物理距离
4、LD分析的软件与画图命令
目前比较常用的计算 LD 的软件为 PLINK ,但不支持直接读取 VCF 格式的文件,使用 PLINK 计算 LD 之前需要先将 VCF 格式的文件转换为 PED 格式或 bed + bim + fam 的格式。这样的格式转换会造成额外的存储负担。而另一款软件PopLDdecay 一个主要的优点在于可以读取 VCF 格式的文件,直接生成 LD 统计数据并画出 LD 的衰减图。以软件PopLDdecay计算绘制LD图的命令如下:
#PopLDdecay -InVCF Final_snps.vcf -OutStat Out.LDdecay -SubPop pop.list
PopLDdecay软件常用参数如下:
Usage: PopLDdecay -InVCF <in.vcf.gz> -OutStat <out.stat>-InVCF <str> Input SNP VCF Format-InGenotype <str> Input SNP Genotype Format-OutStat <str> OutPut Stat Dist ~ r^2 File-SubPop <str> SubGroup SampleList of VCFFile [ALLsample]-MaxDist <int> Max Distance (kb) between two SNP [300]-MAF <float> Min minor allele frequency filter [0.005]-Het <float> Max ratio of het allele filter [0.88]-Miss <float> Max ratio of miss allele filter [0.25]-EHH <str> To Run EHH Region decay set StartSite [NA]-OutFilterSNP OutPut the final SNP to calculate-OutType <int> 1: R^2 result 2: R^2 & D' result 3:PairWise LD Out[1]See the Help for more OutType [1-8] details-help Show more help [hewm2008 v3.41]
更多技术咨询,请关注派森诺官网www.personalbio.cn


