
2021-12-10
单细胞 CUT&Tag 分析
随着10X、BD等平台技术的推进,单细胞层面解析表达和开放染色质水平的研究方法已经得到很好的建立,并且在动植物种中都得到了很好的尝试。
对于研究特定组蛋白修饰或转录因子结合的染色质区域的单细胞分析在技术上具有挑战性。今天小编和大家分享一篇今年四月份发表于Nature Biotechnology 的article 《Single-cell CUT&Tag profiles histone modifications and transcription factors in complex tissues》。
这篇文章使用调整了基于10x Genomics单细胞 ATAC-seq平台,开发并应用了单细胞Cut&Tag (scCUT&Tag)手段,以研究小鼠大脑中单细胞水平的组蛋白修饰谱。在之前的研究中作者发现少突胶质细胞谱系 (OLG) 具有异质性,并且能够在发育和疾病期间转变为替代细胞状态。基于此,作者构建了组蛋白修饰、染色质结构因子和cohesin复合物RAD21的亚基以及OLG特异性转录因子 (TF) OLIG2等非组蛋白的单细胞层面的结合谱。
技术路线
为了研究OLGs,作者使用了一个表达Sox10:Cre/Rosa26:(CAG-LSL-EGFP)小鼠(RCE)的小鼠模型,该模型主要标记小鼠中枢神经系统(CNS)中的OLGs。作者从P15和P25的小鼠大脑中分离细胞,同时,作者为了更加明确OLGs的分化,在P25时期做了两个重复,分别在少突胶质细胞分化的高峰和髓鞘形成的开始时,并分为GFP+和GFP−群体;分离细胞核并与抗染色质修饰或TFs的特异性抗体孵育,使用蛋白A-Tn5融合标记,用10x Genomics 的scATAC-seq协议处理。
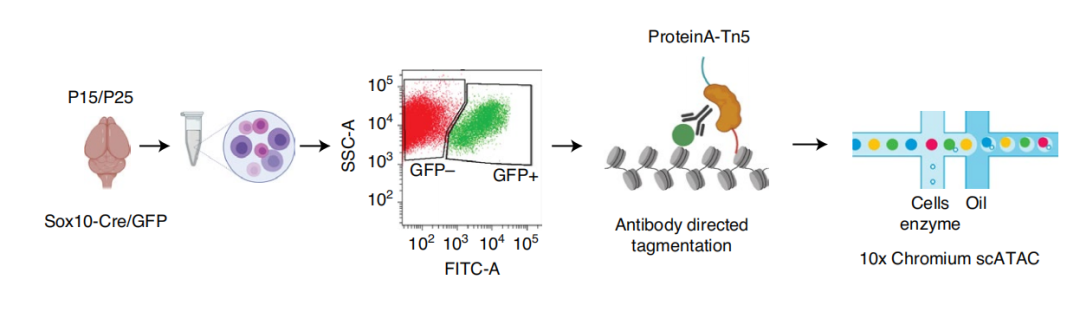
实验技术路线
分析路线
将scCUT&Tag信号整合成一个细胞×bin的矩阵,其中,bin分成不同的大小(5kb或50kb);使用LSI和UMAP进行降维,并使用SNN进行聚类。使用细胞簇来识别标记区域,计算每个细胞的基因活性评分,并与其他数据集进行整合。
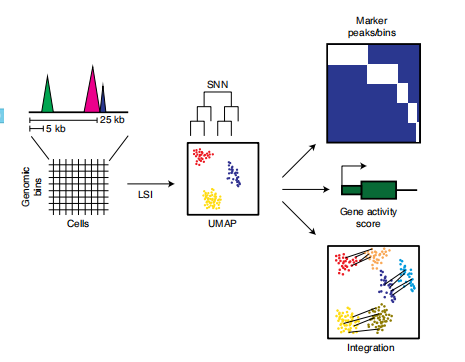
分析路线
在开始正式分析之前为了验证 scCUT&Tag 数据是否可用于分辨异质细胞群,作者制备了三种细胞系的混合物:小鼠胚胎干细胞(mESC,C57Bl/6J 来源)、小鼠胚胎成纤维细胞 (NIH-3T3) (ATCC) 和小鼠少突胶质祖细胞模型细胞系(Oli-neu)。然后使用10x Genomics Chromium 平台对标记的细胞核进行条形码标记,使用 v.1 (rep1) 和 v.1.1 (rep2) 10x Genomics scATAC–seq 试剂盒在两个技术重复中针对 H3K27me3 组蛋白修饰进行 scCUT&Tag优化,具体可以参考文章中的方法。最终获得了4,872和3,873个单细胞的H3K27me3 谱,每个细胞分别具有597和568个独特的片段。使用5-kb窗口聚合数据并为所有数据集生成单元特征矩阵。为了减少数据集的维度,作者使用了LSI及UMAP进行降维,并使用共享最近邻 (SNN) 和使用 Signac/Seurat v.3 包实现的Leiden算法对单元进行了聚类。降维和聚类产生了一个被识别为3T3细胞的簇和Oli-neu(Oli-neu_A 和 Oli-neu_B)和 mESC(mES_A 和 mESC_B)的两个亚簇(下图a)。作者的数据与其他参考数据集显示出了告诉的相似性(下图b)。同时还对前150个最可变的标记峰进行了主成分分析 (PCA),并观察到了各自的批量数据和 scCUT&Tag 数据的共聚类和相关性(下图C)。我们还观察到相应簇之间的两个 scCUT&Tag技术复制之间的峰值信号的高度相关性(下图d)和识别的细胞身份比例的一致性(下图e)。最后,我们在从ChIP-seq 或 CUT&Run 数据中绘制了缩小的的H3K27me3信号的元基因图谱,对比发现合并大约 200-500 个3T3细胞和 20-50个mESCs 产生了与之相当的高质量图谱(下图f-g)。
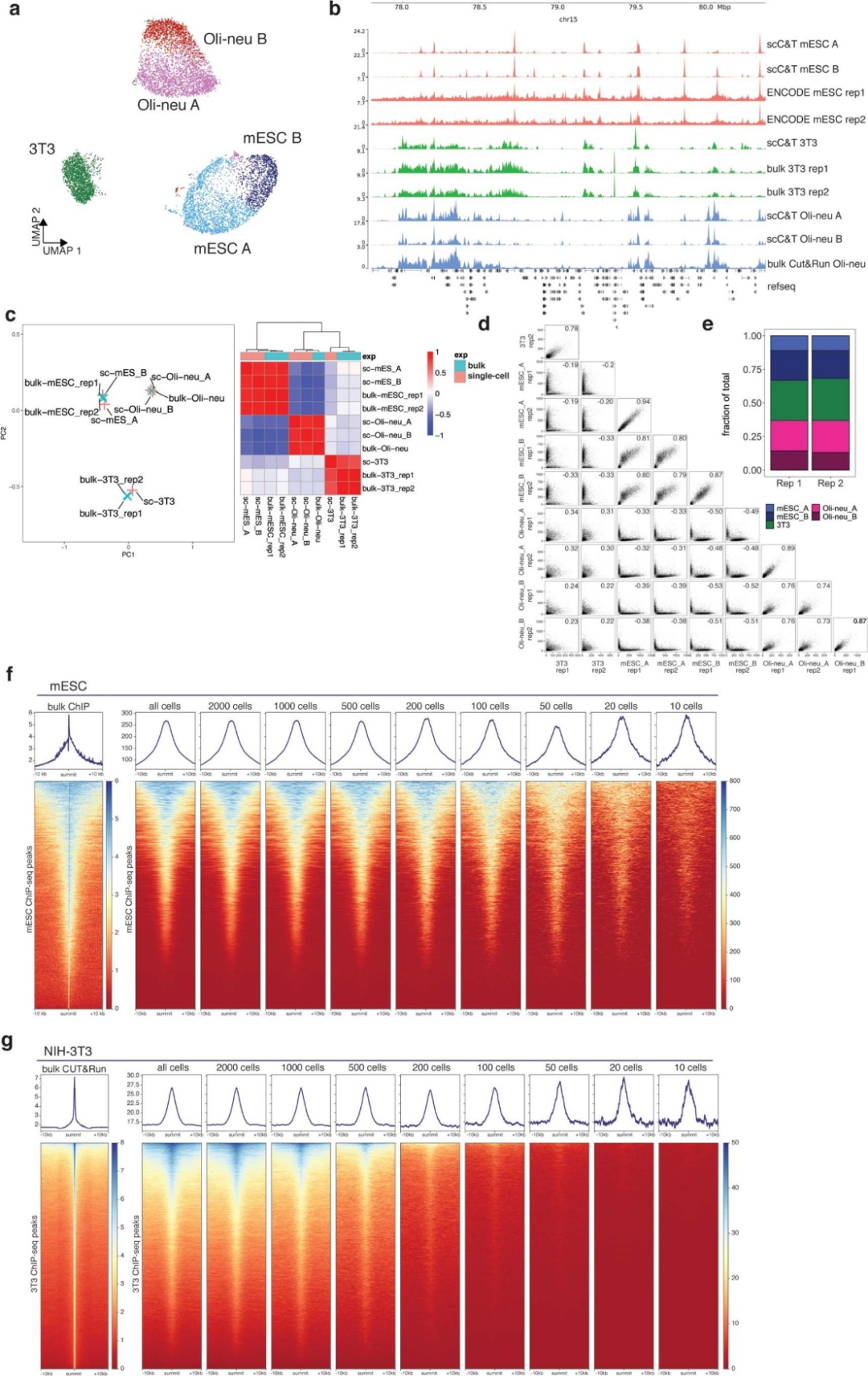
附图1:小鼠细胞系混合物的 scCUT&Tag
结果
1、小鼠大脑中几种组蛋白修饰的单细胞图谱及评估
对所有细胞的合并数据集的伪bulk分析显示了不同组蛋白修饰技术的特异性。特别是H3K4me3主要存在于转录起始位点的侧翼区域,H3K27ac同时占据这些区域和邻近的基因间区域(很可能是增强子),H3K36me3分布在整个基因体中。而H3K27me3与其他活性标记缺失的基因相关,或与H3K4me3相一致。
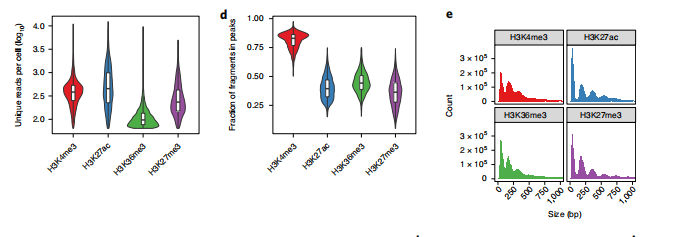
接下来,作者主要进行了单细胞层面的分析,作者基于每个barcode中的reads数及peak区域的reads比例作为参数来筛选真实细胞(PS.10x ATAC中判定peak和细胞很值得一看)。作者获得了 47,340 个单细胞的各种组蛋白修饰的 scCUT&Tag 谱,每个细胞的中位数在 98个(H3K36me3)和453个(H3K27ac)unique fragments左右(图c)。其中有39.4% 至 85.6% 的片段落在窄峰区域内(图d),表明背景水平较低。片段长度分布与亚核小体片段以及所有修饰的单核小体、双核小体和三核小体的捕获一致。
同时作者将数据与iCell8 scCUT&Tag与scChIP–seq m比较,指纹图谱显示,与scChIP-seq相比,scCUT&Tag具有更高的特异性和更好的信噪比,其特异性水平与iCell8 scCUT&Tag相似。
2、单个组蛋白修饰的 scCUT&Tag 可用来识别小鼠大脑中的特定细胞群
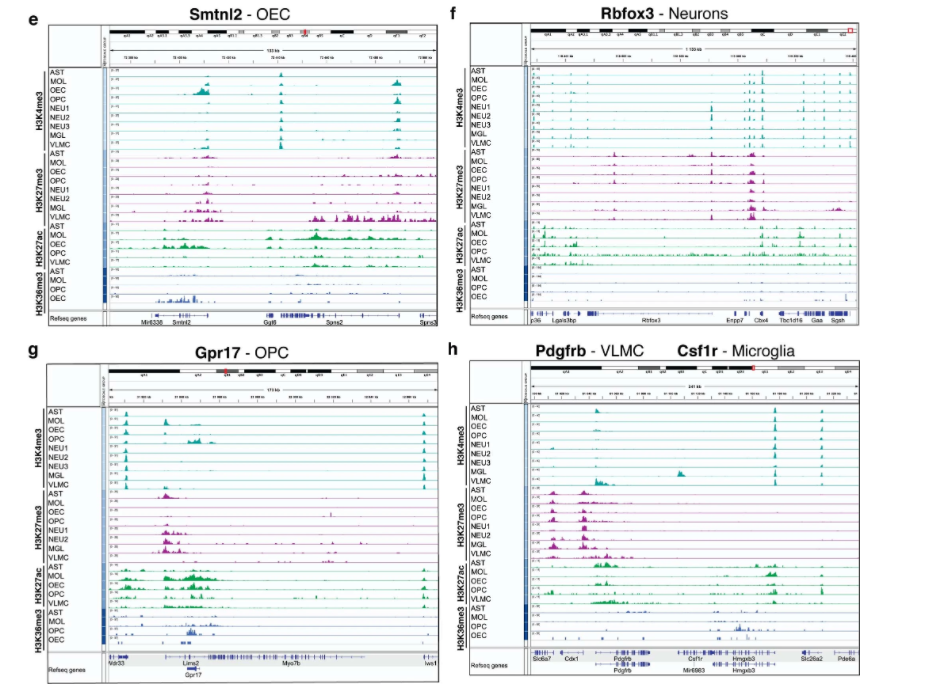
为了对细胞进行解卷积和聚类,作者使用 5 kb(H3K4me3、H3K27ac 和 H3K27me3)以及 50 kb 的分箱大小(H3K36me3)为所有数据集生成了细胞特征矩阵,并在分析 scCUT&Tag 中鉴定了所有主要的 CNS 细胞群。(图 1f-i 和图 2a-f)。通过识别标记基因启动子附近的特定峰(图 1j、k 和图 2a-f),我们手动将亚群注释为成熟少突胶质细胞(MOL、Mbp+、Mog+ 和 Cldn11+)、星形胶质细胞(AST、Slc1a2+、Rfx4+ 和Aqp4+)、嗅鞘细胞(OEC、Alx3+、Alx4+ 和 Frzb+)、血管细胞(VAS、Nes+、Tbx18+ 和 Foxf2+)以及少突胶质祖细胞 (OPC)、定型 OPCs (COP) 和新形成的内突细胞 (oligo NFOLs)(Pdgfra+、Neu4+ 和 Gpr17+)在 GFP+ 部分(图 1j、图 2a-f)。GFP− 细胞主要包括神经元(NEU、Rbfox3+ 和 Neurod2+)、兴奋性(Exc、Slc17a7+)和抑制性(Inh、Gad1+ 和 Gad2+)、星形胶质细胞(Slc1a2+、Rfx4+ 和 Aqp4+)和小胶质细胞(MGL、C1qa+ 和 CDpr4+) (图 1j、图 2a-f)。我们可以在 H3K27me3 scCUT&Tag 中识别相似的种群,并使用在标记基因区域附近缺乏抑制标记的标记组合对它们进行注释(图 1j、图 2b、d-f)。
在完成了细胞类型鉴定之后,作者发现聚类在生物学重复中具有高度可重复性,来自 P15/P25 年龄的细胞在聚类内很好地混合。OLG 谱系的细胞状态反映了小鼠的年龄,大多数 OPCs 来自 P15,分化的 OLG 来自 P25。有趣的是,我在 Sox10:Cre/RCE 小鼠的 GFP+ 部分中检测到一个主要的、可能是瞬时的 AST 群体,最有可能来自腹侧区域(ventral regions)。
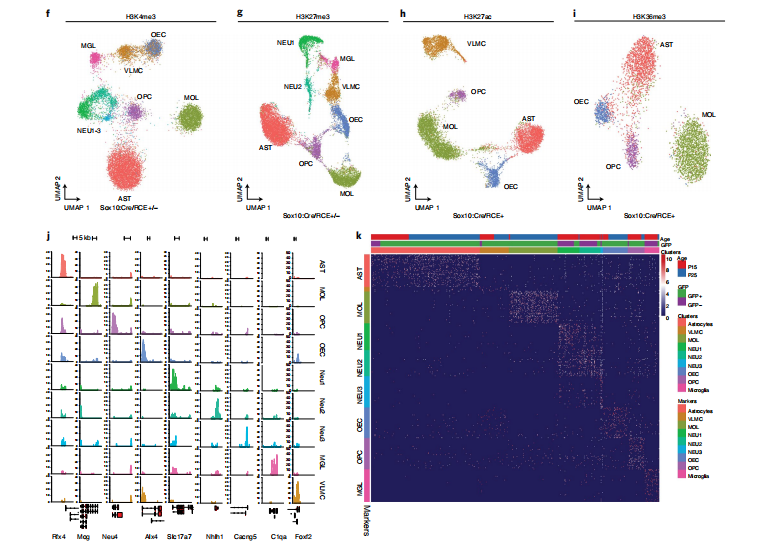
图1:细胞类型鉴定注释
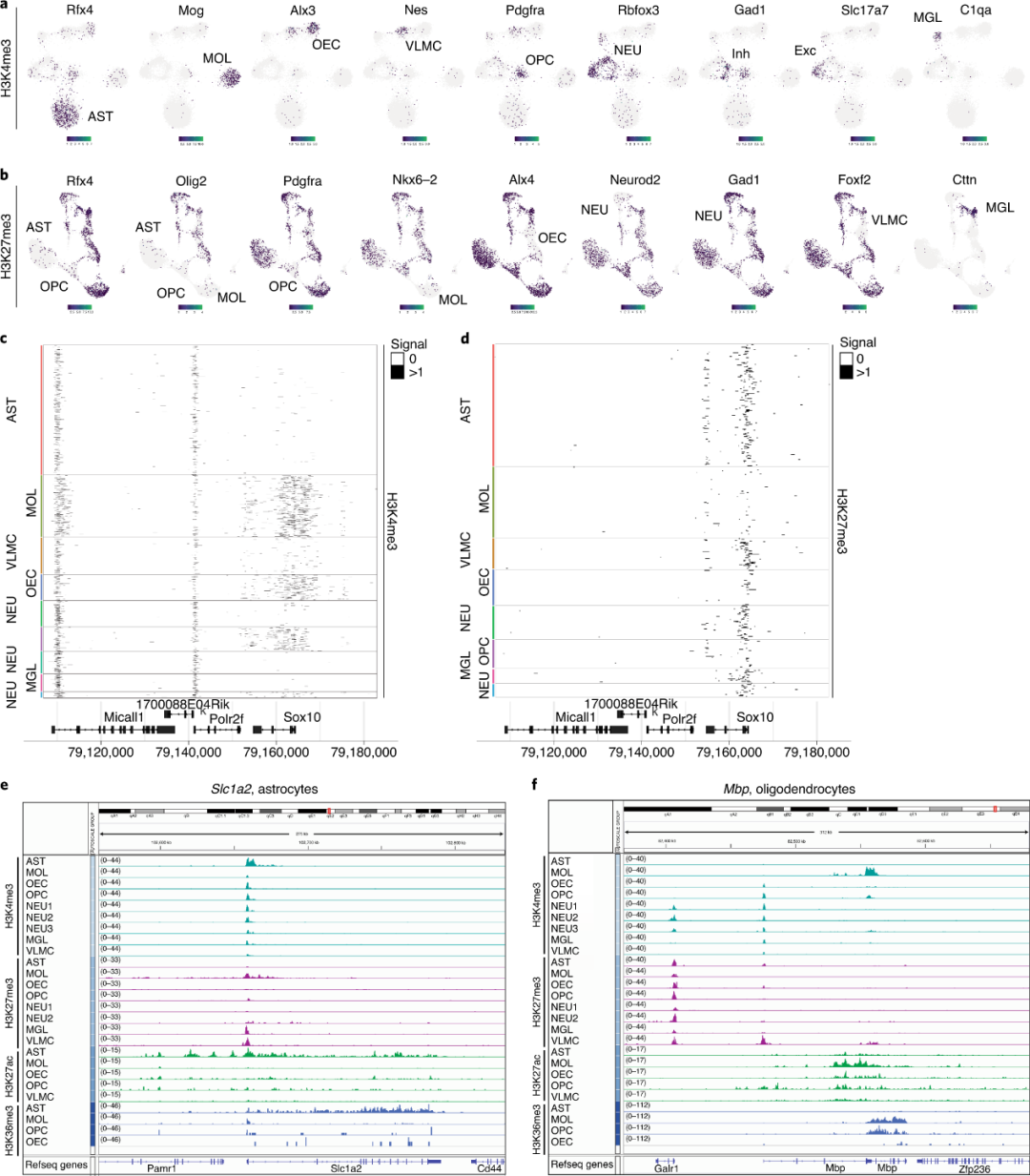
图2:基于scCUT&Tag标记区域的细胞亚群从头鉴定
3、scCUT&Tag 数据与单细胞基因表达的整合
为了验证亚群的手动注释的准确性,我们使用了青春期小鼠大脑 scRNA-seq atlas1。我们为选定的群体挑选了100个最特异表达的标记基因,生成元基因模块,然后计算模块内的基因活性评分(基因体和启动子中的 scCUT&Tag 信号)。我们发现特定细胞簇显示出主动修饰的元基因信号富集,并且在 H3K27me3 数据集中耗尽,证明了亚群注释的准确性。此外,作者在单细胞水平上使用典型相关分析 (CCA) 将 H3K4me3 scCUT&Tag 与 scRNA-seq 数据进行整合,发现主要细胞群与相应的 scRNA-seq 群聚在一起(图3a)。最后,使用基因本体 (GO) 术语分析对 H3K4me3 scCUT&Tag 簇进行功能注释,并发现了 不同的GO通路富集,例如星形胶质细胞分化和激活 (AST)、髓鞘形成 (OLG)、髓鞘形成调节 (OEC)、参与血管生成的细胞迁移(VAS)、神经胶质细胞发育 (OPCs)、神经元发育、神经元成熟和轴突生成 (NEU) 以及参与免疫反应的小胶质细胞激活 (MGL)在各自的亚群中被富集到。
之前的一项研究表明,可以在少突胶质细胞群中检测到更多的细胞亚型。作者想到是否可以通过将 scCUT&Tag 数据与现有OLG scRNA-seq 数据集集成来解决这种异质性。为此,作者使用 CCA 整合了H3K4me3 scCUT&Tag和 scRNA-seq,获得了较为理想的整合结果,并且保留了 scRNA-seq 聚类结果(图3b)。然后作者使用 OPC、MFOL、MOL1、MOL2 和 MOL5 的元基因评分来揭示 H3K4me3 scCUT&Tag 数据中的细胞亚型特征,有趣的是,作者发现看起来同质的少突胶质细胞群可以进一步解卷积为富含模块特异性基因的亚群(图 3c),表明少突胶质细胞异质性反映在表观遗传水平。
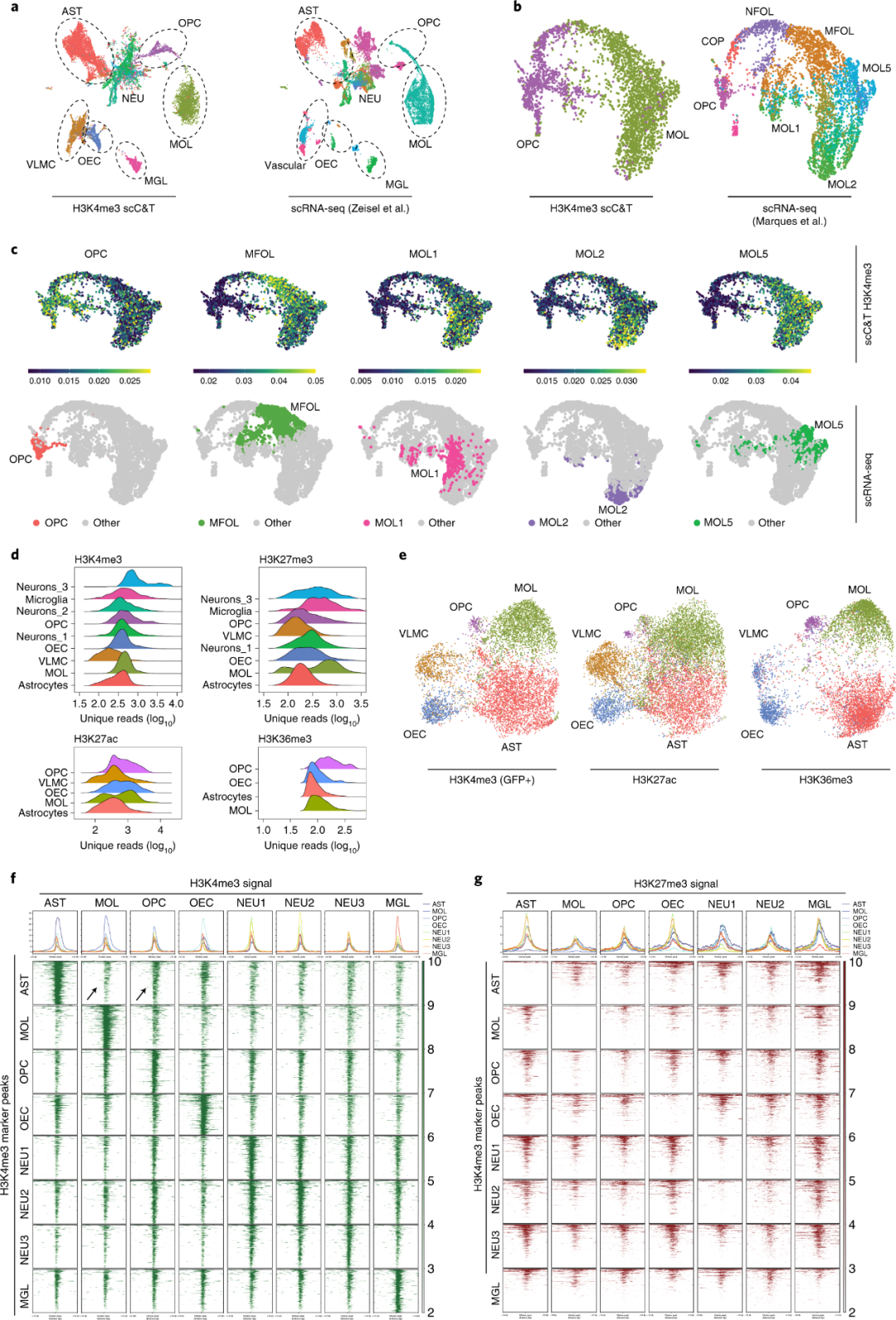
图3:H3K4me3 scCUT&Tag数据与小鼠脑图谱 scRNA-seq数据的整合分析
4、单细胞群中组蛋白修饰的全局差异和全基因组模式
由于 scCUT&Tag配置文件是针对所有现有亚群同时生成的,因此可以对其全局和全基因组的组蛋白修饰模式进行定量分析。作者使用每个细胞的唯一reads数作为单个细胞中组蛋白修饰绝对量的代表。并且发现在不同亚群间这方面存在很大差异(图3d)。这对于H3K27me3最为突出,相对于其他群体,它富含少突胶质细胞、小胶质细胞和一部分神经元(图3d)。少突胶质细胞中H3K27me3的富集与最近的发现一致,即H3K27me3在发育过程中驱动少突胶质细胞 - 星形胶质细胞的转换。有趣的是,我们还在未成熟少突胶质细胞群(OPC/COP-NFOL 阶段)中观察到相对较高量的 H3K36me3(图3d)。尽管细胞类型中抗体特异性信号全局水平的异质性可能是由不同的渗透性和/或标记引起的,但我们没有注意到特定细胞类型中所有组蛋白修饰的信号富集一致。因此,信号异质性不太可能是由细胞类型之间的差异标记效率引起的,而是由细胞类型之间的不同修饰水平造成的。作者接下来好奇是否可以在不同的活性修饰中分配细胞群并将它们相互关联。由此,作者使用CCA以基因分辨率整合数据。引人注目的是,所获得数据的二维即可以高精度重现了原始的非监督聚类(图3e),并且在不同数据集中用相同细胞类型注释的亚群在空间上的位置也类似(图3e)。为了进一步检查活性标记和抑制标记之间的相互作用,作者确定了对由H3K4me3标记的个体群体特异的所有活性启动子,并绘制了所有群体的每个簇的 H3K4me3(图3f)和 H3K27me3(图3g)的信号。正如预期的那样,当启动子富含 H3K4me3 时,可以观察到 H3K27me3 耗尽(图3g)。有趣的是,作者注意到星形胶质细胞特异性基因在OPCs中比在 MOLs 中具有更高的H3K4me3信号(图3f)。此外,H3K27me3信号从MOL中的 OPC特异性基因中耗尽,但在星形胶质细胞中没有(图3g),表明H3K27me3在MOL分化过程中不需要抑制OPC基因。相比之下,H3K27me3信号存在于OPC和MOL的AST特异性基因中(图3g)。最近有报道称,H3K27me3的中断会损害 OPC向MOL的分化,并触发向星形细胞命运的转变。此外,所呈现的表观遗传图谱表明AST在表观遗传上(H3K4me3)与 OPCs相关。
5、少突胶质细胞分化后H3K4me3的宽度增加
H3K4me3标记的广度之前与各种细胞类型、基因表达和转录一致性相关联。我们在H3K4me3堆积分析中注意到,与其他亚群的标记基因启动子相比,亚群特异性标记基因启动子的H3K4me3信号的幅度和宽度都增加了(附图2)。为了量化广度,作者专门研究了从scRNA-seq数据中识别出的标记基因的启动子。作者发现已鉴定种群的标记基因平均具有更高的H3K4me3宽度(图4a)。此外,单个细胞类型的宽度大小不同,AST和少突胶质细胞的启动子上的H3K4me3峰最宽,血管和软脑膜细胞 (VLMC) 和OPCs上的峰最窄(图4b),这可能表明从祖细胞状态到完全分化状态的转变过程中H3K4me3的宽度增加。为了进一步研究H3K4me3传播的动态,作者基于scCUT&Tag,以单细胞分辨率可视化H3K4me3的传播,并在从OPCs分化MOL的过程中研究了H3K4me3的广度。作者围绕MOL特异性标记基因生成了单细胞H3K4me3元基因图谱。然后我们根据来自MOL中表达的基因的H3K4me3 信号(MOL签名)对矩阵中的细胞(OPC和 MOL)进行排序,并通过拟时序分析进行验证(图 4c、d)。引人注目的是,可以观察到H3K4me3信号在MOL启动子处的宽度逐渐增加,具有单细胞分辨率(图4e),这与H3K4me3随着细胞向分化的少突胶质细胞特征进展时的扩散一致。
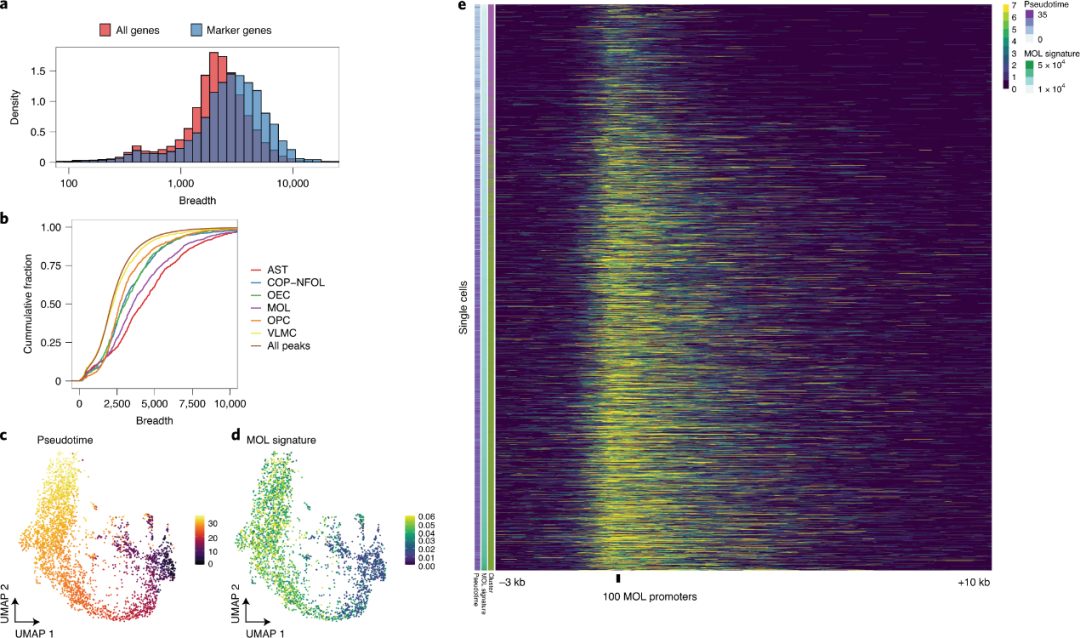
图 4:在单细胞分辨率下H3K4me3标记在启动子上的传播
附图2:四种组蛋白修饰的亚群的scCUT&Tag表达
6、scCUT&Tag of TFs
众所周知,在low input样本中使用ChIP-seq很难分析 TF 结合。因此,作者好奇scCUT&Tag是否能够以单细胞分辨率揭示 TF 的结合,于是他们选择了TFs OLIG2,其对神经胶质群体具有特异性,另外选择了RAD21,其是一种通用染色质结构因子和cohesin复合物的亚基。作者对出生后第 25 天的小鼠的大脑的GFP+分选细胞进行了scCUT&Tag。与组蛋白修饰相比,TF scCUT&Tag 的每个细胞的独特读取数较低。尽管如此,在OLIG2和RAD21 scCUT&Tag获得唯一reads的中位数依然在48 and 240。使用 LSI和UMAP进行降维,并获得了RAD21和OLIG2的特定集群(图 5a-d)。可以看到OLIG2的两个亚群的唯一reads的差别交大,作者将具有低唯一reads的集群注释为“low binders”(图 5a)。由于在TF CUT&Tag中基于标记手动注释种群具有挑战性(图 5e),基于亚群注释的假设是特定细胞类型中的OLIG2/RAD21结合与增强子/启动子活性相关。因此,作者分析了OLIG2/RAD21 在scCUT&Tag数据中被H3K4me3特异性修饰的基因的启动子区域中的结合,并确定了RAD21 scCUT&Tag中的 AST、OLG 和OEC群以及OLIG2 scCUT&Tag 中的OLG和非OLG(“low binders”)。OLIG2 scCUT&Tag中的OLG种群可能由成熟的OLG和OPC组成,它们似乎在OLG 集群中形成了一个亚种群。为了进一步分辨亚群注释,作者将RAD21/OLIG2与另一种组蛋白修饰(使用CCA的H3K27ac)整合,发现识别的簇始终与相应的H3K27ac簇合并。有趣的是,“low binders”中非OLG 细胞随机与H3K27ac定义的OEC、AST 和血管细胞簇群合并,而OLG簇与OLG H3K27ac 信号特异性合并。这一发现与Olig2 在整个细胞类型中的表达一致,它在整个OLG 谱系中高度表达,而 OEC和VLMC不表达Olig2,只有一小部分AST表达Olig2(图 5f)。
为了验证scCUT&Tag的特异性,作者在 RAD21和OLIG2的合并伪批量数据集中使用 MEME 套件在结合位点中搜索了富集的基序。作者发现染色质结构因子CTCF的基序在RAD21数据集中最高富集(图5g),这与CTCF和 cohesin 之间的协同性一致。我们发现了几个富含OLIG2 scCUT&Tag的基序,包括CAGMTG基序,类似于先前在小鼠 和大鼠中分别对OLIG2特异的 CAGMTG/CAGCTG基序(图 5h)。连同先前确定的OLIG2基序,作者发现了多种通用真核增强子和启动子特征(GC框和CAAT框)的富集。有趣的是,作者还发现了一个类似于来自SOX家族的TF的基序(ACARWR,扩展数据图 9f),这与OLIG2和SOX家族TF(SOX8、SOX10)成员之间的物理相互作用和协同性一致.
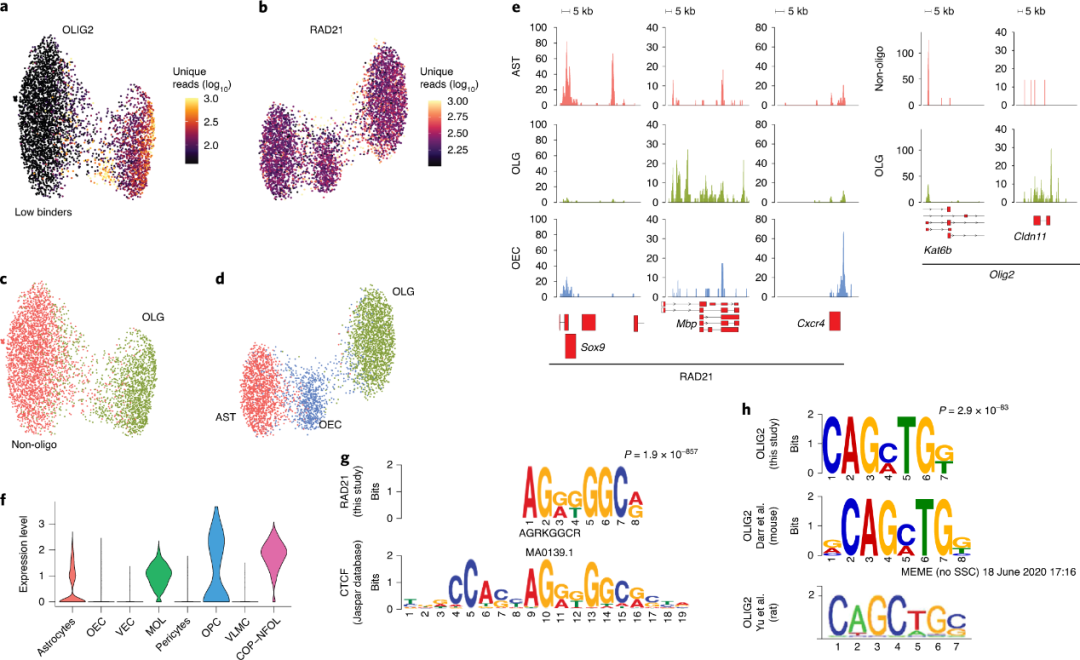
图 5:转录因子结合的 scCUT&Tag 分析
总结
篇幅原因,今天就先介绍到这里,这篇文章的亮点颇多,基于微流控的单细胞CUT&Tag可以更加特异的分析单细胞水平的各种修饰情况;与不同的单细胞数据的结合分析为探究更精细的细胞异质性提供了思路,对于研究复杂样本与组织的老师同学们值得借鉴。