2022-01-14
细胞的状态和行为会受到遗传和环境因素的影响。特别是肿瘤进展是由潜在的基因异常以及肿瘤微环境的构成决定的。量化这些因素的贡献需要新技术来准确测量基因组序列的空间位置。
为了同时测量遗传和局部因素,来自哈佛大学干细胞与再生生物学系(HSCRB)、麻省理工学院Broad研究所及哈佛的研究人员开发出一种空间分辨DNA测序的新技术,称为slide-DNA-seq。作者证明该方法准确地识别了局部肿瘤结构,并能够从头鉴别不同的肿瘤克隆及其拷贝数变化。
通过将slide-DNA-seq应用于转移性和原发性肿瘤的小鼠模型,揭示肿瘤克隆局限于不同的空间区域。此外,通过与空间转录组学的整合,发现了与克隆特异性遗传畸变、局部肿瘤微环境或两者相关的不同的基因。
这种多模式空间基因组学方法提供了一个多功能平台,用于量化细胞内外因素如何促进基因表达、蛋白质丰度和其他细胞表型。
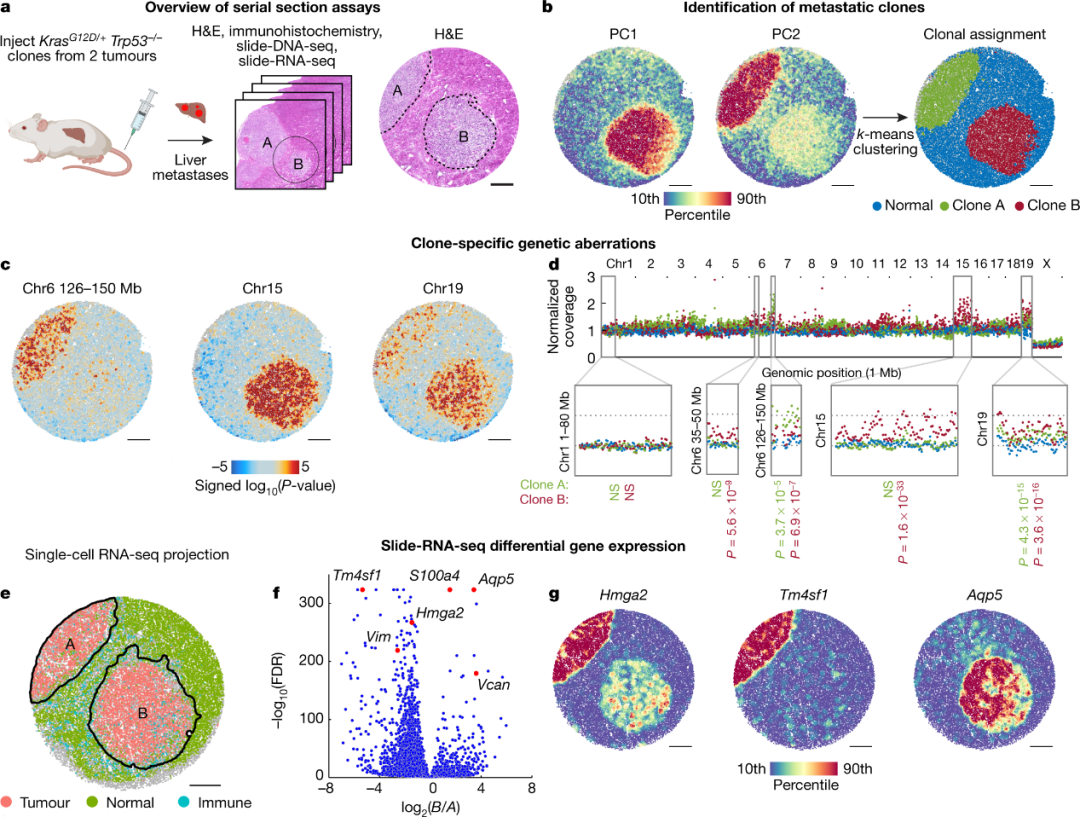
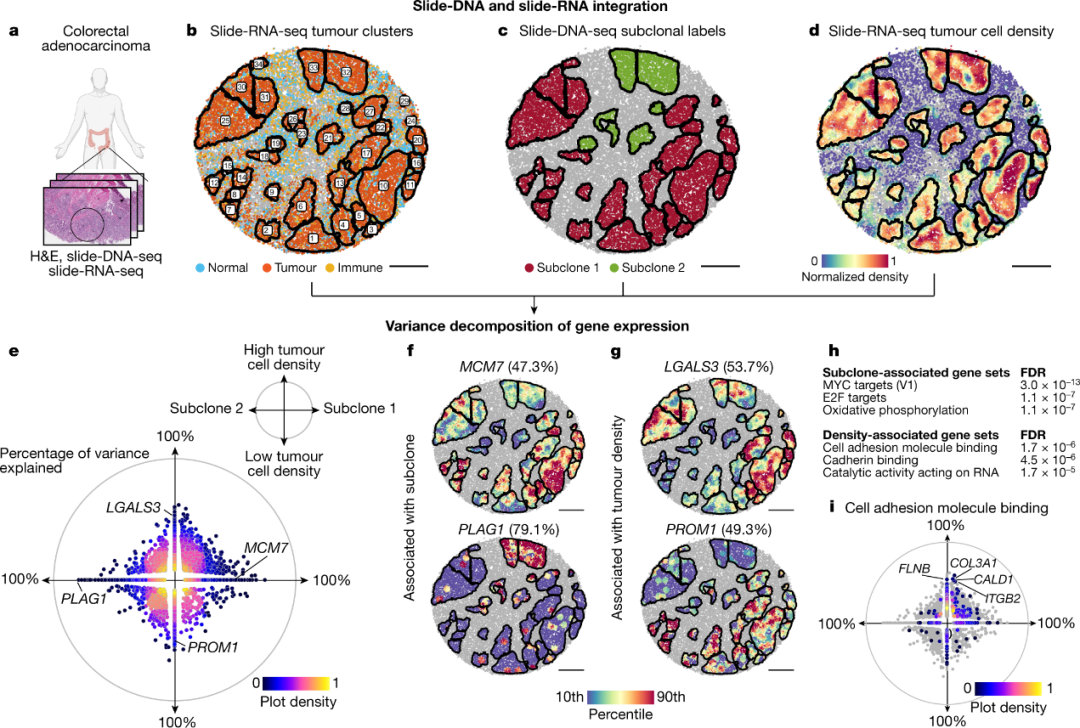
摘要 组织功能与精确细胞类型的空间位置相关,其状态受细胞内在遗传因素和外在环境线索的影响。在癌症中,肿瘤细胞的克隆群体进化出多种多样的DNA突变、拷贝数改变(CNA)和大的染色体重排,导致肿瘤内遗传异质性,这与耐药性、转移的风险增加有关和复发。同时,构成肿瘤微环境的周围正常细胞相互交流,形成具有不同生化和生物力学特性的空间邻域,影响细胞迁移和侵袭以及药物渗透性。解耦和量化肿瘤内的这些遗传异常和环境线索对于了解癌症进展和改进治疗至关重要。 目前用于描述肿瘤内遗传异质性的方法包括深度测序以量化等位基因突变频率和单细胞全基因组测序。作者认为当前人们对肿瘤克隆是如何在组织内分布,以及癌症进展在多大程度上是由克隆特异性遗传畸变或环境因素驱动的认知知之甚少,这突出了对可以大规模整合基因组、转录组和空间测量的新方法的需求。 空间分辨DNA测序 开篇明义,作者构建了一种叫做slide-DNA-seq的方法,可以实现能够对完整组织进行空间分辨DNA测序。 作者首先制作了一个3毫米beads的空间索引的序列,和slide-RNA-seq类似。每个10微米的聚苯乙烯珠包含一个独特的DNA条形码,该条形码对应于一个空间位置,并且连接接头引物等序列。然后将组织切片冷冻,并将单个10微米的新鲜冷冻切片转移到测序珠阵列上。为了实现DNA的无偏捕获,组织切片用HCl处理以去除组蛋白并用Tn5转座以创建两侧有定制接头序列的基因组片段。然后从bead上光断裂空间条形码,将它们连接到近端基因组片段,并通过PCR扩增得到的DNA测序文库(图1b)。在文库构建之后,使用高通量双端测序,并使用DNA条形码将每个基因组片段与磁珠阵列上的空间位置相关联。这些关联使后续分析能够重建组织中DNA的空间位置,而无需在显微镜下对样本进行成像。同时,作者针对组织固定、组蛋白去除和桥接寡核苷酸杂交进行了优化,均一的最大化文库大小,使Tn5对染色质的切割均匀,同时保留了组织结构。在完成最初的优化之后,每个阵列包含20,000到40,000个bead,每个bead的中位数为165到421个片段。此外,作者开发了一种概念性的修改版的方法,它使用重复的Tn5标记来提高产量,可以使基因组捕获片段增加十倍。 为了确定这种方法的空间和基因组分辨率,作者将slide-DNA-seq应用于小鼠小脑,该小鼠小脑包含不同的核密集(体细胞)和富含线粒体,作者使用DAPI和针对线粒体蛋白TOMM20的抗体染色(图1e),图1d图是基于线粒体的百分比进行着色,两者之间具有很好的相似度。为了测量基因组上的分辨率,作者使用相同组织的bulk测序校正了序列偏差和归一化覆盖率的数据。基于这种方法,99.78%的非重叠1Mb基因组bin的标准化拷贝数在1.5和2.5之间(图1f)。总之,这些数据表明,slide-DNA-seq可以在空间上定位正常组织内的基因组信息。 检测空间位置上的CNA分布 接下来,作者将slide-DNA-seq来应用于测量肿瘤切片中拷贝数改变(CNA)的空间分布,使用已知具有染色体扩增和缺失的肺腺癌基因工程小鼠模型,作者从KrasG12D/+Trp53-/-(KP)小鼠肺肿瘤中分离并扩增了单个肿瘤克隆,并将该克隆注射到小鼠的尾静脉中,从而在肝脏中引起大的转移,构建肿瘤转移模型。 在分析方法上,作者开发了一个幻灯片-DNA-seq分析工作流程,包括两个主要步骤:(1)克隆群体的从头识别和空间定位(2)每个克隆的基因组CNA的表征。 首先,为了检测和定位组织内的肿瘤克隆,我们根据空间接近度(k=50个最近的beads,直径在110微米)对beads数据进行平滑处理并执行主成分分析(PCA)以找到在整个组织中具有可变覆盖率的共同相关基因组区域。然后,通过k-means聚类为slide-DNA-seq阵列上的每个beads分配一个克隆型。基于这种方法应用于来自肝转移瘤的slide-DNA-seq阵列时,主成分1(PC1解释了2.89%的方差)显示出空间图案(图1h)与对晚期肿瘤标志物HMGA2在连续切片上的免疫荧光染色一致。同时为了验证这种方法是否可用于识别遗传上不同的肿瘤克隆,作者对4个肿瘤细胞系的bulk测序进行了下采样分析,发现于每个样本仅有1000个片段也显示出较好的稳健性。表明该策略中数据量对于slide-DNA-seq是足够的。 分析工作流程中的第二个任务是表征每个肿瘤克隆中存在的CNA。作者以1Mb的分辨率可视化了每个集群的基因组覆盖率。当应用于肝转移队列中,肿瘤相关簇显示出显着的CNA,包括染色体chr6的扩增,这是KRAS诱导的肺肿瘤的特征,而正常簇显示出相对均匀的覆盖(图1j)。 基于以上的结果,表明了slide-DNA-seq分析工作流程能够以大约1-Mb 的基因组分辨率从头发现和定位肿瘤区域。 图1 转移性克隆的空间基因组学 为了证明该实验和计算方法可以区分组织内的克隆,作者将源自两个独立衍生的转移性 KP 肿瘤的多个克隆注射到小鼠的尾静脉中,这会在肝脏中产生大的转移。然后作者进行了 H&E 染色并确定了一个组织区域,该区域具有两个空间上不同的转移灶(图2a)。同时作者也是用了免疫组化进行了进一步的确认。作者使用slide-DNA-seq对第三张切片进行了测序,发现使用上面描述的方法后,基于PC1与PC2可以明显的分辨出两个肿瘤转移灶(图2b)。作者开发了一种置换检验的方法以在空间定位一个或两个转移灶中存在的具有统计学意义的 CNA gain或loss,并检测到 chr6、chr15 和 chr19 上的差异区域(图2c)。作者在全基因组上比较了选定区域覆盖率的统计显着性(双边Wilcoxon秩和检验;图2d中的P值),进一步证明它们是由不同的克隆播种的。此外,作者观察到一个克隆可能是三倍体,并且使用流式细胞术独立证实了这一点。 为了测试两个克隆之间的遗传差异是否反映在细胞状态中,作者在第四个切片上使用了slide-rna-seq进行单细胞核转录组测序,基于单细胞核转录组的无监督聚类与在slide-rna-seq上的空间映射,显示出两个转移在转录水平上也是不一致的(图2e),分析发现在两个克隆间有3,732 个基因差异表达(图2f)。接下来,作者分析了一下两个克隆差异表达的明星基因。克隆 A 具有较高的晚期肿瘤标志物表达,包括 Hmga2(肺转移瘤)、Tm4sf1(JAK-STAT 信号传导)和 Vim(细胞运动),而克隆 B 的中高表达的包括 Aqp5(谱系身份缺失)和上皮细胞间质转化标志物 S100a4和Vcan25(图2f,g)。 通过以上结果,作者表明了配对的slide-DNA-seq和 slide-RNA-seq能够对遗传上不同的转移性肿瘤克隆及其相关细胞状态进行空间表征。 图2 人结肠癌中的亚克隆检测 由于结直肠癌是全世界癌症相关死亡的常见原因之一,84% 的此类肿瘤表现出染色体不稳定性。同时作者也想确定slide-DNA-seq是否可以在原发性人类肿瘤中从头检测克隆异质性。基于此,作者从IIIB期结直肠肿瘤中选择了一个样本。和之前一样,作者在连续切片上进行了H&E染色、多重免疫组织化学和slide-DNA-seq(图3a)。首先,检查H&E染色发现许多直径约100-500微米的局部肿瘤细胞聚集体。假设这些聚集体中的每一个都可能来自单个克隆谱系,可能是由于迁移受到限制,作者猜测也有可能是每个聚集体包含来自不同谱系的细胞的混合物,表明是细胞混合的结果。 那为了验证上述可能,与上文所述方法类似,作者进行了PCA和无监督聚类,产生了三个不同的基因组图谱(图3b)。其中一个簇的空间分布在视觉上与H&E染色中的正常组织一致(图3b右,蓝色区域),其中也包括中等PC1评分的区域,表明含有CNA的癌细胞丰度较低。相比之下,其他两个集群显示出较高的PC1分数,并且在空间上仅限于不同的肿瘤聚集体,这支持了每个聚集体起源于单一谱系的假设。与之前的报道类似,单个结直肠肿瘤细胞在腺体组织中播种,其中相邻细胞共享最近的共同祖先。作者通过slide-DNA-seq、H&E 染色和肿瘤标志物MKI67、免疫标志物CD45的免疫组织化共同分析验证了slide-DNA-seq检测到的肿瘤结构(图3c)。 接下来作者对不同的克隆型中的遗传畸变进行了描述。作者发现了几种遗传畸变,包括 chr8q 扩增和chr15 和chr18的缺失,它们在所有肿瘤区域中共享,表明它们在肿瘤进化的早期出现并且可能在肿瘤起始中起重要作用。chr8q扩增包含已知促进肿瘤进展的基因,包括原癌基因MYC和MYBL134,而chr15的缺失导致基因组稳定性所需的多个基因丢失,包括TP53BP135、RAD5136和FAN137。与这些共同的畸变相反,作者观察到chr1q、chr7和chr20的亚克隆扩增,这可能发生在进化的后期(图3d,e)。值得注意的是,先前对结直肠癌的分析将chr7p扩增归类为典型的克隆(而不是亚克隆)事件,而chr20p的丢失和获得都被确定为频繁的亚克隆畸变。这些事件的检测和时间分类证明了slide-DNA-seq在研究克隆异质性进化方面的实用性。 为了验证这些遗传畸变,作者对同一结直肠肿瘤进行了单细胞全基因组测序 (scWGS)。这种方法从整个肿瘤中进行细胞取样(比slide-DNA-seq组织切片多100倍的样本量),由此可能会识别出额外的亚克隆。与这一预期一致,对2,274个高覆盖率单细胞CNA谱的分析产生一个正常簇和五个肿瘤簇,其中一些类似于幻灯片-DNA-seq CNA谱(图3f)。然后,作者试图将高覆盖率测序映射到slide-DNA-seq阵列上,以增强分辨率识别CAN。空间区域主要匹配两个单独的scWGS簇仅支持使用slide-DNA-seq进行的分析,同时作者还发现了一个具有明显遗传畸变的小区域,该区域仅在 scWGS数据覆盖率较高时才显示。在展示了改进的空间分辨率后,作者以100kb的基因组分辨率重新分析了匹配的scWGS簇,揭示了第8号染色体中复杂的CNA图谱(图3g)。 图3 克隆异质性的多模态分析 最后,为了展示多模态空间测序方法的独特能力,作者试图量化肿瘤转录程序如何受遗传和环境因素的控制。作者首先对来自结直肠肿瘤附近区域的连续切片进行H&E染色、slide-DNA-seq和slide-RNA-seqV2(图4a),并整合病理、基因组和转录组信息。然后作者确定了肿瘤细胞的不同空间区域(图4b),并继续为每个区域分配亚克隆身份(图4c)同时并量化局部肿瘤密度(图4d)。 考虑亚克隆身份(细胞内在)和肿瘤密度(细胞外在)水平,作者使用去卷积的方法解析这些因素如何促进结直肠肿瘤的转录程序。基于方差分解方法,对于每个基因,计算亚克隆同一性解释的基因表达方差、肿瘤密度和无法解释的方差的百分比。slide-RNA-seq检测到的25,074个基因中,412个基因与亚克隆同一性显著相关,638个基因与肿瘤密度相关,1,098个基因与两者的组合相关(P<0.05,方差解释 >30%,图4e)。与亚克隆同一性相关的基因包括位于扩增区域的已知癌症基因,例如PLAG1,chr8q39上的癌基因和MCM7,chr7q上参与DNA复制起始的MYC靶基因(图4f)。值得注意的肿瘤密度相关基因包括 LGALS341(也称为galectin-3),它有助于肿瘤微环境中的免疫抑制,以及PROM1(也称为CD133),它对肠道稳态、再生和肿瘤起始起重要作用(图4g)。 除了表征单个基因,我们还进行了基因集富集分析以确定哪些分子途径与亚克隆或肿瘤密切相关(图4h)。该分析表明,亚克隆差异主要改变了参与细胞生长和增殖的基因的表达,其中MYC和E2F靶基因代表了亚克隆1的最高hallmark基因集。相比之下,与高肿瘤密度相关的基因最富集细胞粘附分子和钙粘蛋白结合特性(图4i),包括细胞外基质成分基因COL3A1、肌动蛋白调节基因FLNB和CALD1,以及机械转导调节基因ITGB2(也称为CD18)。值得注意的是,细胞外基质僵化和重塑被认为可以促进细胞增殖和肿瘤进展,这可能导致高肿瘤细胞密度。总体而言,这些分析证明了这种多模态方法在解耦和量化遗传和环境因素对基因表达的贡献方面的实用性。 图4 总 结 至此,我们已经为大家介绍了单细胞转录组,单细胞ATAC,单细胞cut-tag,单细胞基因组等方法应用。不难看出,多组学应用以及从bulk纷纷过渡到单细胞层面。特别是多模态这种多种组学联合分析的方法,在解析复杂疾病是十分有效的。欢迎各位老师同学联系我们,共同探究单细胞多组学的应用。