
2022-05-14
在之前文章推送中,我们对RNA提取结果进行分析,并对部分特殊结果的案例进行了简短的总结和展示。虽然小伙伴们已经知道如何分析结果,也能看懂质检图,但一直以来询问较多的,还是对RNA质量差如何才能继续建库比较感兴趣。下面我们就针对RNA质量较差的样本(样品降解、DNA污染、蛋白污染、色素残留、多糖污染、浓度低等)提供一些思路。
一、降解的RNA
大多数RNA是单链结构,碱基之间没有氢键,不稳定易降解。除了其本身含有丰富内源酶的特性外,其他外源因素,如样品制备情况、取样情况、抽提方法的选择等也可能会导致RNA的降解。针对降解的RNA主要有以下3种解决方案。
1.1
原样有剩余的,可以尝试提取方法的改变
由于有些样本组织结构特殊、代谢产物极其丰富,可能会影响到RNA提取质量。另外,由于RNA酶的广泛存在并难以消减的顽强特性,使得RNA的抽提纯化更为困难。
1.2
可以再次制备原样的,需要在制备、收集、保存、运输等方面更加注意
收集样品时需保持低温、无RNA酶等;制备样品时化学、物理等胁迫的程度不同可能导致RNA有不同程度的降解;短时间内解剖(切割,摘取)的组织样品,其处理及切割过程应在冰上尽可能快速进行;样品离开活体或原生长环境后,样品中的内源酶会开始降解RNA,降解速度与内源酶含量及温度有关。
收集好的样品,应该立即液氮速冻15min以上,随后-80℃保存,之后可以用干冰寄送。路程较远或运输时间较长的,可适当加大干冰用量。从样本收集到送样最好不要超过3个月。
1.3
原样珍贵,难以再次获取的,可以尝试风险建库
二、有杂质污染的RNA
针对不同的杂质污染类型,可以选用不同纯化思路,以下为小编总结的几种常见的杂质污染及解决方法。
2.1
蛋白污染、色素残留
产生蛋白污染的原因可能有:1)样本本身(如豆类,动物肝脏等)富含蛋白;2)取样组织过多或裂解液较少,导致裂解不充分;3)提取过程中(如Trizol法)吸到少量蛋白层等。
色素残留常与样品的特性有关。甲壳类动物、某些昆虫的外骨骼、真菌细胞壁及某些绿藻中含有多糖甲壳素,是RNA中色素残留的主要来源。
以上情况,我们可以利用磁珠或吸附柱对RNA再次进行纯化回收,经适当漂洗,即可得到较高纯度的RNA。
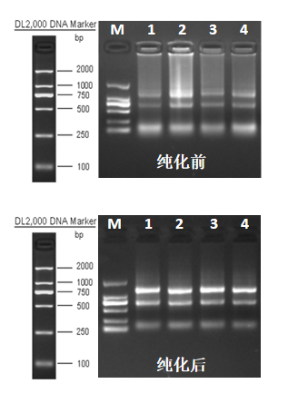
图1 胶孔蛋白污染纯化前后对比

图2 色素残留纯化前后对比
2.2
DNA污染
有些物种富含蛋白、多糖(如淀粉、当归多糖、软骨多糖)等,裂解过程中这些物质和核酸一起被释放,核酸易被这些粘稠的物质包裹,RNA提取中就会出现有DNA污染的情况。
我们可以选用DNA酶对样品进行DNA消化处理,之后再用磁珠或吸附柱对RNA进行回收,并将引入的DNA酶进行纯化去除,即可得到高纯度RNA。
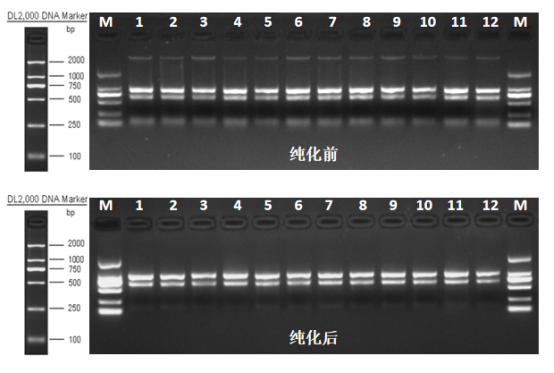
图3 DNA污染纯化前后对比
2.3
多糖污染
小麦、水稻种子、某些药用植物根茎的RNA中容易出现多糖污染的情况,由于多糖与核酸的性质相似,所以不建议用纯化的方式进行去除。派森诺拥有发明专利的提取方法,可在前处理阶段对多糖进行有效的去除。
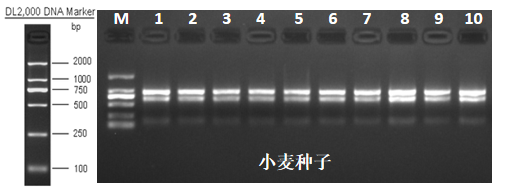
图4 小麦种子RNA胶图
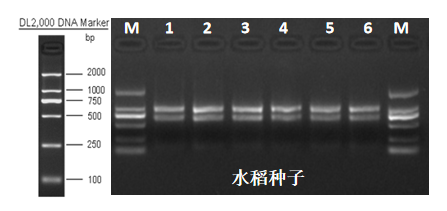
图5 水稻种子RNA胶图
三、浓度低的RNA
3.1
浓度低(检测有条带)、体积大、总量相对较高的RNA样品
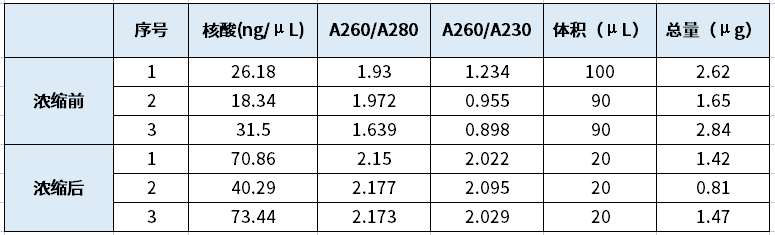
我们可以通过浓缩回收的方法来提高RNA的浓度。常用的方法还是选用磁珠或吸附柱对RNA进行吸附,经适当漂洗、晾干后加入适量的无酶水进行洗脱。这种方式不仅达到了升高浓度的目的,而且可以去除一部分杂质。下表为经吸附柱浓缩回收的对比表,RNA的回收效率在49%及以上,且纯度更高,RNA建库的成功率也相对更高。
表1 RNA浓缩前后对比
另外,利用真空离心浓缩仪也可以对RNA样品进行浓缩,其原理是在真空状态下溶剂可在低温中沸腾蒸发,这种方法虽然能有效的提高RNA的浓度,但样品中的杂质浓度也会随之升高,且RNA有较大的降解风险,对后续RNA建库也会带来困难,所以一般不推荐使用。
3.2
浓度低(检测有条带)、体积小、总量相对较小的样品,我们是否可以进行风险建库或是微量建库?
很多受到来源限制的样品,如医院的临床样本、极端环境下样本,此类样品一般制备困难、送样量较少,提取的RNA总量低并且质量较差;还有些样品本身的RNA丰度低,如动物软骨组织、脂肪及眼角膜等;经胁迫处理的样品一般细胞状态较差,mRNA易降解且含量较少,如经高温/干旱/高盐处理的植物、细菌等。以上种种情况都有可能导致最终的RNA难以满足建库要求。
对于此类样品,派森诺有着丰富的建库经验,样品浓度低至4ng/μL的RNA样品可用于建库,建库成功率接近100%。
3.3
浓度低(检测无条带)的RNA样品
由于琼脂糖凝胶检测条件的限制,RNA条带无法显示,使用安捷伦2100高灵敏芯片能够检测到核酸,也可以进行微量建库。若样品组织量足够,可以考虑在提取阶段适当进行多管合并提取,以增加核酸得率;或考虑在样品处理及收集中是否有处理不当的地方,优化后再进行提取建库工作。
除了对以上质量较差RNA的补救措施外,某些降解、浓度总量低的RNA样品,也可以进行后续的建库及上机测序。
A、浓度总量极低样本微量建库
针对浓度总量较低的样本,推荐微量转录组文库。我们选取部分样本进行结果展示,图6为RNA的琼脂糖凝胶电泳图,RNA样品浓度较低,胶图上两条主带清晰,但亮度微弱。实验室对其进行微量转录组建库,qubit文库浓度在10-15ng/μL之间,labchip质检结果见图11,文库峰型正常。
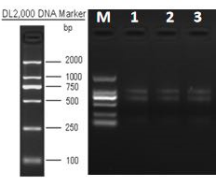
图6 浓度总量低样本琼脂糖凝胶电泳图
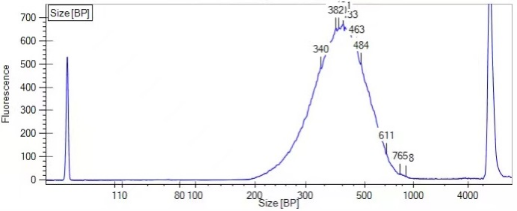
图7 文库片段大小labchip检测结果
将文库进行上机测序,其中上机模式为Novaseq PE150,挑选其中2个样本的下机数据质量进行统计及评估,表2中为下机各项数据统计,N(%)及Q30(%)正常;将下机数据过滤成高质量序列后的过滤统计见表3,高质量序列占比正常,数据可信度高。
表2 下机数据统计
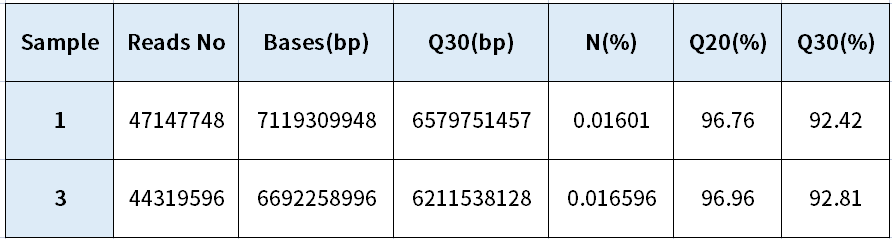
注:Sample:样品名;
Reads No.:Reads总数;
Bases(bp):碱基总数;
Q30 (bp):碱基识别准确率在99.9%以上的碱基总数;
N(%):模糊碱基所占百分比;
Q20(%):碱基识别准确率在99%以上的碱基所占百分比;
Q30(%):碱基识别准确率在99.9%以上的碱基所占百分比。
表3 数据过滤统计
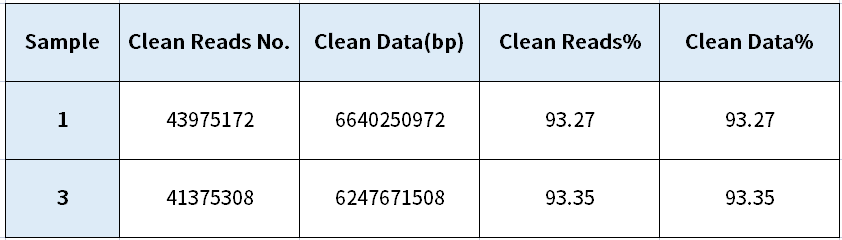
注:Sample:样品名;
Clean Reads No:高质量序列read数;
Clean Data (bp):高质量序列碱基数;
Clean Reads %:高质量序列reads占总reads的百分比;
Clean Data %:高质量序列碱基占测序碱基的百分比。
单碱基质量分布见图8,大部分序列的碱基质量在20以上,代表测序质量较好。图9为用于评估测序结果的均一性(或是否有偏向性)的基因上测序深度分布图,理想条件下,Reads在所有表达的基因上的分布应该呈现均一化分布,从图9上看,基因覆盖均一度均匀,测序结果的偏向性不强。
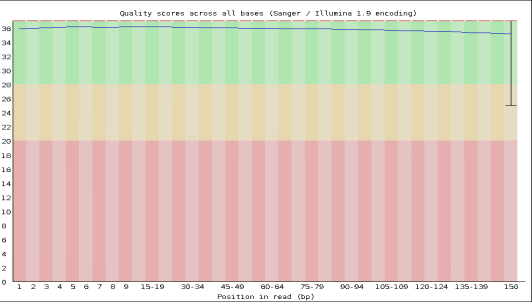
图8 单碱基质量分布图
注:横坐标是 Reads 中碱基位置(5’->3’),纵坐标是对应位点碱基 Q 值。红线代表中位数,蓝线代表平均数,黄色区域代表 25%-75%区间(按照四分位数划分),触须表示 10%-90% 区间。
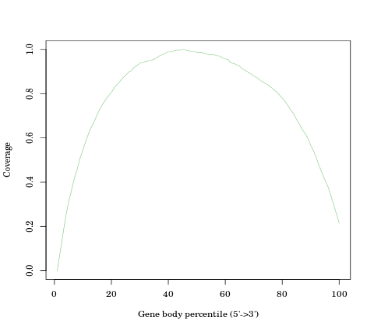
图9 微量转录组文库在基因上测序深度分布
注:横坐标为单个基因的碱基长度占总碱基长度的百分比,0表示基因的5'端,100表示基因的3'端;纵坐标为比对到所有基因的横轴位置上相应区间内的序列条数的总和。图中体现了所有基因覆盖情况的叠加结果,曲线中每个点的纵坐标表示所有基因在该相对比例位置上所有序列的数量;曲线反映了测序所得序列是否在基因上均匀分布。若无明显偏向锋,则说明测序无偏向性。
B、降解类样本风险建库
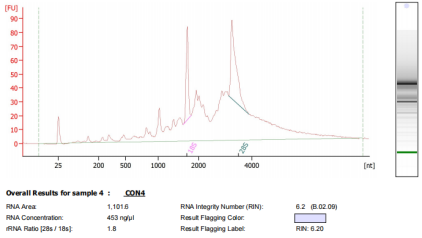
我们可以看到图10中是物种为小鼠的RNA样品琼脂糖凝胶电泳图及2100质检图(其中1个),质检结果为rRNA的完整性,可从侧面反映mRNA的质量。从质检结果上看,5个样品总RNA有不同程度的降解,RIN值在5.5-6.5之间。根据不同样品的降解程度,实验室对该批样品的建库流程进行优化和调整,文库浓度及总量结果见表4;labchip对文库片段大小进行检测,部分结果如图11所示,文库峰型正常,可用于上机测序。
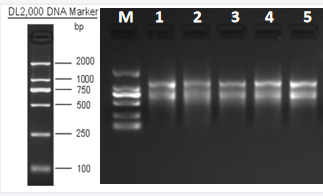
(a)
(b)
图10 降解样本RNA质检图
注:(a)降解样本琼脂糖凝胶电泳图;(b)安捷伦2100质检图
表4 文库qubit浓度及总量
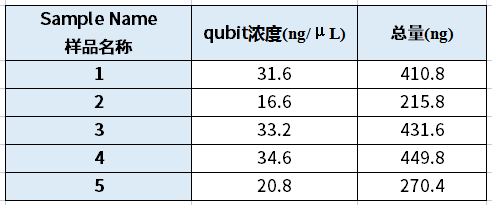
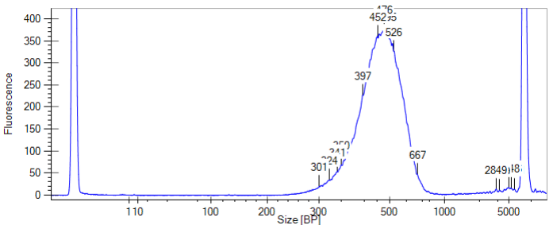
图11 文库片段大小labchip检测结果
下机数据质量统计见表5,表5中为下机各项数据统计,N(%)及Q30(%)正常。下机数据Reads平均质量分布见图12,该图主要用来检测测序数据的平均质量分布情况,从结果来看,峰型集中且无前拖尾峰表示测序数据的数据质量整体较好。另外与参考基因组的比对情况见表6,总的基因组比对率在95.6%及以上。
表5 下机数据统计
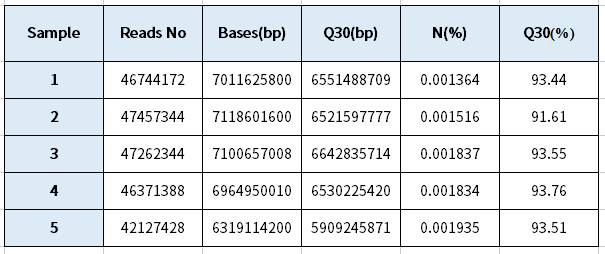
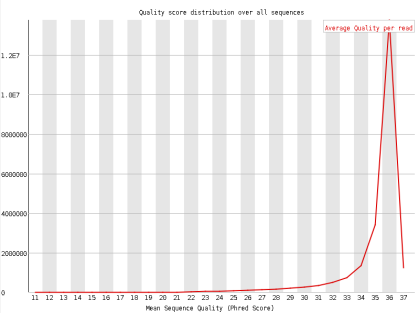
图12 Reads平均质量分布
注:横坐标表示 reads 的平均质量,纵坐标为对应平均质量值的reads数目。
表6 对比结果统计表
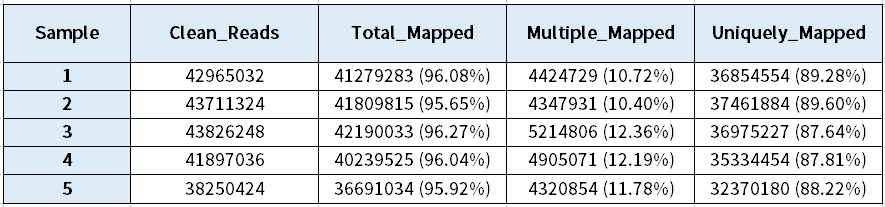
注:Sample:样品 Clean Reads:用于比对的序列总数
Total Mapped:比对上参考基因组的序列总数,百分比为 Total Mapped / Clean Reads
Multiple Mapped:比对到多个位置的序列总数,百分比为 Multiple Mapped / Total Mapped
Uniquely Mapped:只比对到一个位置的序列总数,百分比为 Uniquely Mapped / Total Mapped