2022-06-20
目前陆生动物的群体多样性多数局限于传统的调查方法:其一是直接的方法,主要通过摄像机记录观察或人为现场观察;其二是间接的方法,主要观察动物自身留下的东西,例如脚印、粪便或血迹等。但是这些方法需要消耗大量人力以及时间成本,甚至还需要一定的运气。
哥本哈根大学研究团队提出:“捕获来自脊椎动物在空气中传播的环境DNA(eDNA),有可能使我们探测到那些看不到的动物。“在2022年1月7号的细胞出版社(Cell Press)旗下的期刊 Current Biology上,此团队发表了一项独立的概念验证研究,表明通过当地动物园采集空气样本,他们就可以收到足够的eDNA来识别附近的动物。
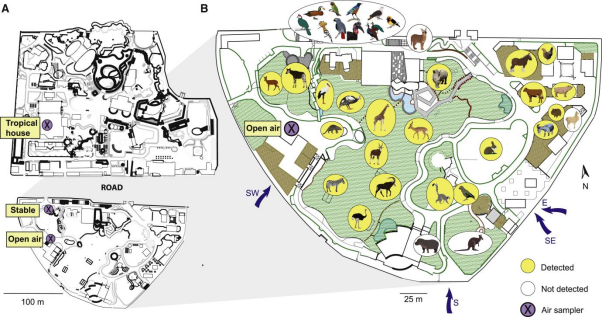
图1
实验设计
实验地点
丹麦的哥本哈根动物园
采集时间
2020年9月与12月
采集点选择
1、Stable:室内-155平米,居住着Okapi johnstoni与Cephalophus natalensis(图1A)
2、Open air:其中包含多种哺乳动物和其他陆地脊椎动物的多个室外围墙(图1A/B)
3、Tropical house:室内-442平米/2200立方米,其中居住着多种小型脊椎动物(图1A)
仪器
1、真空水过滤-吸尘器:8.8立方米/min,滤膜孔径:0.22um
2、F8级百褶纤维颗粒过滤器的鼓风机:0.8立方米/min,电压:24V
3、F8级百褶纤维颗粒过滤器的鼓风机:0.03立方米/min,电压:5V
标记基因选择
线粒体标记基因-95bp:
5’-CGGTTGGGGTGACCTCGGA-3’
5’-GCTGTTATCCCTAGGGTAACT-3’
12S基因-97bp:
5’-TTAGATACCCCACTATGC-3’
5’-TAGAACAGGCTCCTCTAG-3’
human blocker:
5’~3’—GCGACCTCGGAGCAGAACC-spacerC3
5’~3’—TACCCCACTATGCT TAGCCCTAAACCTCAACAGTTAAATC-spacerC3
实验流程
通过在不同的地点使用空气采集器进行收集其周围环境中的空气,其中包含来自各种来源的遗传物质,例如唾液、尿液以及粪便等小到足以漂浮在空气中的颗粒。接着,对空气收集器当中滤膜进行DNA提取,再利用特异引物进行PCR扩增获得对应物种的特定DNA(同时去除人源DNA)进行建库上机测序,最终将测序的数据与DNA的参考数据库进行比对分析,以鉴定检测到的物种信息(图2)。
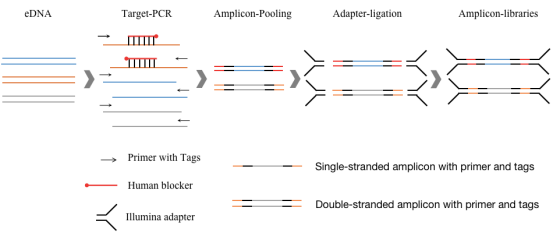
图2
实验结果
在三种地点,共收集了40份样本,检测到了49种非人类脊椎动物的DNA,主要包括:30种哺乳动物,13种鸟类,4种鱼类,1种两栖动物以及1种爬行动物(图3所示)。在这49种中有38种是动物园内已有的物种,3种是用于动物饲料的鱼类,3种动物园内已知害虫,其余5种是野生动物或是已知的动物园周边的物种。而进一步分析,发现风向是影响实验结果因素之一,例如在动物园的南部采集位置仅检测到北部的一种物种,而当时的风来自西南,南,东南和东部,北部没有吹来的风(图1);同时,也发现了动物园内人员流动也是会影响动物园动物的eDNA,例如在Tropical house,经常被投喂食物的动物(袋鼠或山羊等)并未被检测到。
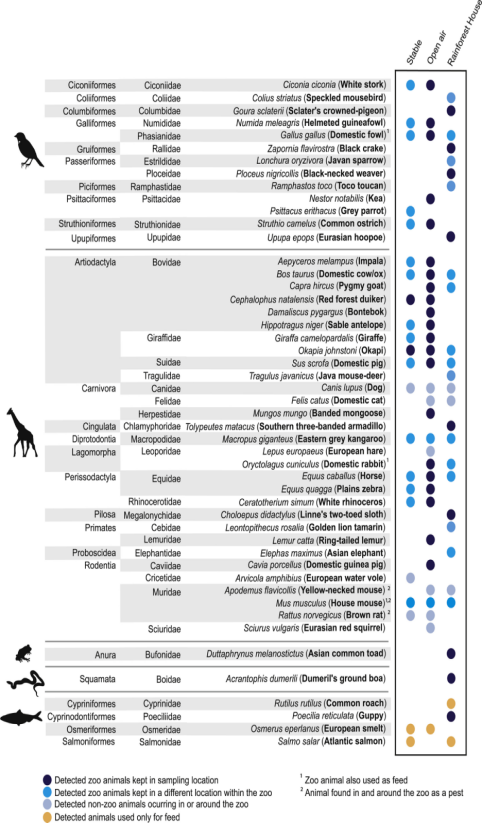
图3
讨论
在2022年同月,伦敦大学玛丽皇后学院也使用相同的技术手段,在Hamerton Zoo ParK内检测到25种哺乳动物和鸟类的DNA,甚至包括在英国濒临灭绝的欧亚刺猬的DNA(IF:10)。虽然此项技术方案的结果,容易受到多种环境因素的影响,但是整体结果还是比较接近自然情况,甚至还可以检测到未被发现的物种。而鉴于目前全球气候以及生物多样性的危机,通过空气采集物种DNA的方法,可以彻底改变陆地生物监测的方式,并且能够时时跟踪动物群落的组成以及及时发现非本地物种的入侵。
文献:Lynggaard, C., et, al. (2022). Airborne environmental DNA for terrestrial vertebrate community monitoring. Current Biology.https://doi.org/10.1016/j.cub.2021.12.014


