
2022-08-31
随着单细胞测序技术的迅速发展,其在临床研究、基础科研等领域的应用成果已经得到了认可,在探究细胞异质性问题中发挥了不可替代的作用。为了将单细胞测序技术更广泛地应用到各领域研究中去,人们逐渐将目光锁定到了微生物领域,以微流控为基础的微生物高通量单细胞基因组学技术——Microbe-seq应运而生,正式开启微生物单细胞应用新时代。
2022年6月3日哈佛大学和麻省理工学院的研究团队在Science上联合发表了题目为High-throughput, single-microbe genomics with strain resolution, applied to a human gut microbiome的研究长文,文中介绍了该团队在微生物群落研究方法学上的重大突破性技术:Microbe-seq。
运用这项技术,作者团队从来自人类肠道的复杂微生物群落中分离出单个微生物的基因组:用微流控技术将单个微生物包裹在液滴中裂解细胞释放DNA,用液滴中含有的特异性barcode进行标记扩增、测序,共得到了20,000多个微生物单扩增基因组,组装成了近100个细菌物种的基因组,其中包括几种具有多个亚种菌株的细菌,利用这些基因组来探索微生物之间的相互作用,重建了水平基因转移(HGT)网络并发现了92个物种对之间的HGT,并发现了噬菌体与一株普通拟杆菌的体内宿主-噬菌体关联关系。
接下来,我们带大家解析这篇文章:
分辨菌株与单菌基因组的重要性
目前在人类肠道中已经探察到近千种肠道菌群,然而每个个体的肠道菌群组成都是特异的,即使是同一菌种的不同菌株间也会因基因组的变异表现出不同性状,这些差异性状导致它们在生化反应、代谢产物的产量、耐药性以及与宿主免疫系统的相互作用等方面有着截然不同的表现,会直接影响个体健康情况。不仅如此,菌株之间的相互作用也同样会影响到肠道微生物群落中的微生物行为,比如噬菌体调节细菌组成,微生物细胞之间遗传物质的转移等等。
受现有技术水平限制,现阶段人们仅能够在物种水平上分析微生物菌群组成,并不能全面解析特定微生物的基因和功能途径,因此,急切地需要一种菌株水平的解析方法来填补技术空白,为人们理解肠道微生物行为及其对健康的影响提供帮助。
现有技术短板
一直以来,学者们一直在尝试使用各种方法来探索菌株水平的基因组问题,但得到的结果总是不理想:
1、鸟枪法宏基因组学尽管已经有部分技术进步(如长读长测序、read-cloud测序和Hi-C)开始提供一些物种的菌株水平信息,但还是不能有效地区分单个样本中多个分类群共有的DNA序列,例如当一个物种有多个菌株或多个分类群的基因组中出现同源序列时。
2、传统的培养方法只能培养出少数易于生长的微生物菌株,且成本偏高,耗时长,不能还原样本微生物菌群的全貌。
3、基于微孔板的单细胞测序虽然可以测出菌株水平的基因组,并已被用于探测噬菌体和细菌之间的关联,但由于技术及成本限制,只能依赖于在滴度板上的孔中分离和裂解单个或大约十几个微生物,再扩增其全基因组进行测序,涉及细胞数量过少。
对于所有这些现用方法,技术手段严重限制了生成源自同一群落的菌株分辨基因组的数量,从而限制了对人体肠道微生物组的基因组结构和行为的了解。
技术原理
微生物高通量单细胞基因组学技术——Microbe-seq很好的解决了以上问题,制备高通量样本步骤如下所述:
1、利用液滴微流控技术,将微生物单独地包裹在含有裂解液的油包水液滴中;
2、在每个液滴中裂解微生物释放基因组DNA,这个过程必须保证每个液滴中只含有单个微生物;
3、将含有裂解后微生物的液滴转移到第二个微流控装置中,与含有扩增试剂的液滴在电场帮助下合并(并未破裂)。使用最为广泛应用的MDA扩增法进行全基因组扩增;
4、将液滴转移到第三个微流控装置,将之前的液滴与第三个装置中含有转座酶等试剂的液滴合并(并未破裂)。此步骤可以将扩增出来的DNA片段化并连上接头,随后在液滴中用带有特定序列的标签标记液滴内DNA;
5、使用第四个微流控装置将之前的液滴与另一个含有barcode引物的磁珠液滴合并。这些引物包含两部分:一部分是每个液滴唯一的barcode序列。另一部分则是对先前添加的接头进行退火的序列,通过PCR将这些barcode引物连接到每个液滴内的片段DNA分子上。
6、最后打破液滴,将所有液滴内的DNA合并,添加测序接头使用Illumina平台进行测序。流程示意图如下:
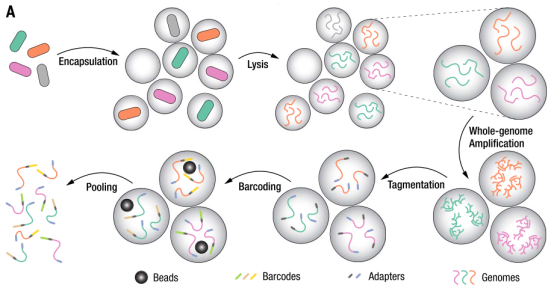
由于每一个液滴内DNA标记的标签是唯一的,因此将测序得到的序列按照标签进行归类即可得到每一个液滴内单个微生物的基因组信息。这些具有相同barcode的测序序列的集合即称为单扩增基因组single amplified genome (SAG)。
验证Microbe-seq的可靠性——性能评估实验
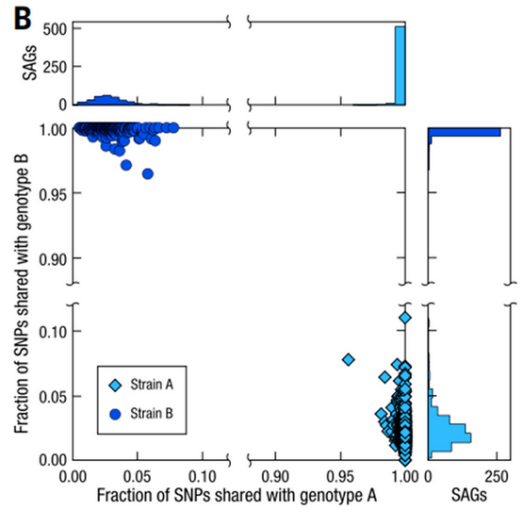
为了验证每一个SAG所包含的基因组信息是否可信,作者应用Microbe-seq方法对四种已知菌株构建了模拟微生物群落样本进行测试。实验共得到了5497个SAG,用纯度来代表每个SAG中单微生物基因组的比例,结果显示84%的SAG纯度超过95%,表明绝大多数SAG都来自单一微生物,如下图B所示。
作者统计单个SAG包含的基因组信息的比例后发现,对于革兰氏阴性菌大肠杆菌和肺炎克雷伯菌,平均基因组覆盖率分别为8%和10%;而对于革兰氏阳性菌枯草芽孢杆菌和金黄色葡萄球菌,比例为17%和25%,这是由于革兰氏阳性菌基因组较小导致的基因组覆盖率差异。对于上述四种菌株,几十个SAG基本上包含了微生物基因组的所有信息,如下图C所示。
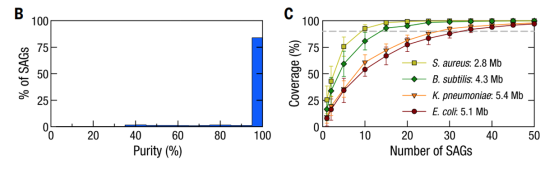
由于模拟样本实验中得知单个SAG涵盖的基因组信息一般在全基因组的50%以内,无法直接被用作该样本的参考基因组,为了获得高质量的微生物群落的参考基因组,识别单个SAG,作者团队设计了一种不参考外部基因组的生信分析方法:
1、将同一物种多个相似SAG进行基因组组装,然后将平均核苷酸同一性(ANI)值超过95%的基因组归为同一物种,并将它们的序列进行合并,进而获得物种水平的基因组作为参考基因组。
2、将单个SAG与得到的参考基因组进行比对,识别来自于不同菌株的单细胞微生物并进行基因组组装。
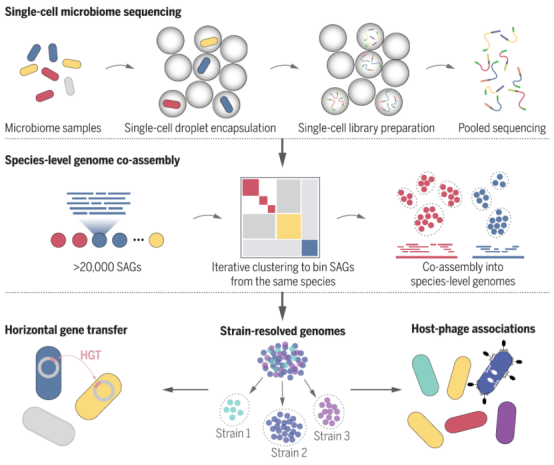
Microbe-seq技术流程示意图
人类肠道微生物菌群应用结果及分析
文中实验选取了从1名健康受试者收集的7份纵向粪便样本,获得了21914个单扩增基因组(SAG),其中从每个样本中得到了约1000-7000个SAG,每个SAG平均包含约70000条序列。与同源样本宏基因组结果相比,这些SAG捕获了相似水平的物种基因组。
1、构建高质量人体肠道微生物样本参考基因组
在这个健康人的肠道微生物样本中,通过Microbe-seq共组装得到76个物种的基因组,其中52个物种具有高质量基因组(完整性>0.9,污染<0.05),24个物种具有中等质量基因组(完整性> 0.5和污染<0.1)。超过四分之三的SAG属于这76个物种中的一个,表明Microbe-seq得到的大部分SAG的可以用于构建独立的参考基因组。
通过与同源样本分离培养细菌的基因组结果进行比较,发现76个物种中有19个物种被成功分离培养。在这19个物种中,有17个物种的组装基因组与分离培养得到的基因组高度一致,表明Microbe-seq可以获得与分离培养相似水平的可靠基因组。
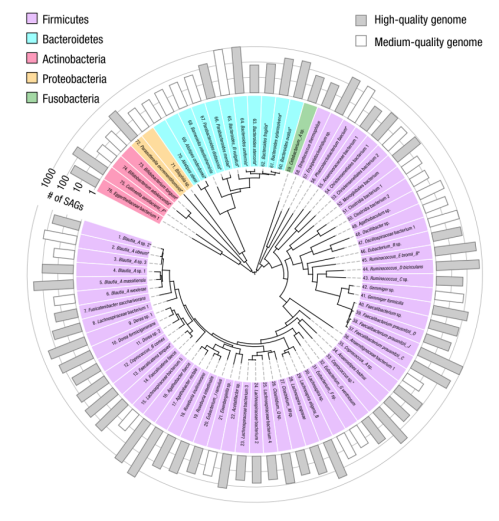
由核糖体蛋白序列构建的76种细菌的系统发育由圆中心的树状图表示
注:外圈柱子长度代表在其组装基因组过程中使用的SAG数目。星号标记的是19个具有相应分离株的基因组
将每个样本中所有 SAG的所有reads模拟宏基因组学中产生的数据与微生物基因组公共数据库进行比较,对每个样本中的每个read进行分类,与同源样本宏基因组微生物多样性结果做对比,发现通过Microbe-seq检测到的属与宏基因组结果重合度高达96.9%-99.8%。由于与宏基因组的裂解方式不同,导致革兰氏阳性菌(如厚壁菌门和放线菌门)的比例过高而革兰氏阴性菌(尤其是拟杆菌门和变形菌门)的比例相对较低,但同一个门内的微生物的丰度趋势在两种方法中一致,表明Microbe-seq可获得与宏基因组一致的微生物多样性。
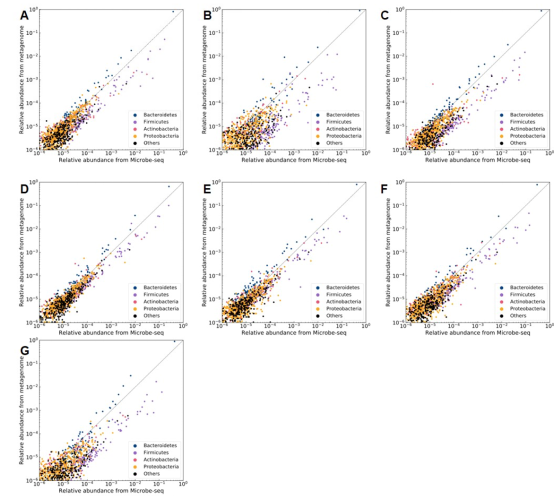
7个纵向样本使用Microbe-seq与宏基因组方法的微生物多样性比对结果
2、精准解析菌株水平的基因组信息,发现新菌株
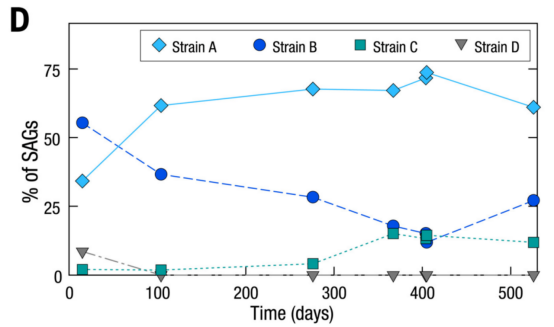
为了区分菌株,作者团队开发了一种利用 SAG之间同源序列差异的方法:将SAG重新比对到物种水平的组装基因组,可以得到基因组中所有SNP位点在该SAG的reads中出现的比例。同一菌株的微生物具有几乎相同的基因组,因此代表同一菌株的两个SAG几乎总是在两个SAG共享的每个SNP位点具有相同的碱基;相反,代表不同菌株的SAG显示出相当低的相似性。依照SNP信息对普通似杆菌的SAG进行层次聚类分析并进行降维可视化所有SAG对之间的SNP相似性,发现SAG分为四个明显分离的聚类簇:将SAG最多的两组高质量基因组其标记为候选菌株A和B;一个中等质量基因组C;以及另外一个质量较低的基因组D,如下图A:
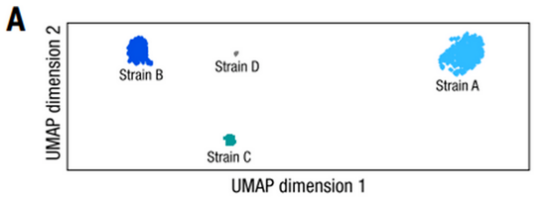
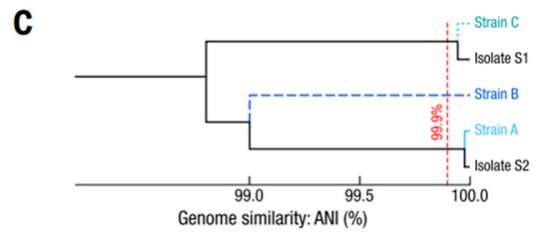
分析SAG最多的两个簇,发现每个普通似杆菌的SAG聚类簇具有一致的基因型,而与其它SAG簇的基因型重叠程度很低,这说明每个SAG聚类簇代表不同的菌株,如下图B。其中,菌株A和菌株C与分离培养的两个菌株的基因组一致,菌株B为鉴定到的全新菌株,这些结果强有力地证明了Microbe-seq能够正确鉴定出已知菌株和尚未培养的潜在新菌株,同时实现其基因组的精确组装。
进一步将这种基于 SNP的分析应用于具有高质量或中等质量物种水平基因组的剩余物种,在该个体的76个微生物物种中,作者团队总计发现了10个具有多个不同的菌株的物种,并且组装了对应的菌株水平的基因组。在前文中提到的19个已经被成功分离培养的物种中,有两个物种的共组装基因组与培养测序所得的基因组具有一定偏差,而这两种物种都具有多个菌株,这也解释了共组装基因组与培养测序基因组存在偏差的原因,如下图C。
识别每个 SAG菌株的能力能够辅助追踪这些菌株随时间的推移在人类供体中的相对丰度,从而深入了解细菌种群动态。在收集样本的一年半中,这些菌株的丰度变化幅度不大。例如,作者团队在第400天左右连续收集的两个样本中观察到菌株B的丰度非常相似,如下图D所示。这些观察结果表明不同的拟杆菌门物种可以稳定地在人体肠道定殖数十年,并且相同拟杆菌门物种的不同菌株可以以稳定的相对丰度共存。
3、重建菌株的水平基因转移现象
微生物研究中一个特别值得注意的课题是研究微生物如何交换遗传信息,其中最著名的机制之一是菌株的水平基因转移(HGT)现象。
将来自两个物种的基因组之间的 HGT事件定义为存在至少5 kb且具有99.98%相似性的共同序列。作者将这些标准应用于所有57个高质量菌株分辨基因组,过滤掉SAG合并造成的潜在污染,并观察来自不同物种的90对菌株之间的265个HGT序列,共发现了66个单个个体肠道微生物群中HGT现象,且同一门中的菌株之间的转移明显大于不同门中的菌株之间的转移:65对菌株在厚壁菌门内,25对菌株在拟杆菌门内。如下图:
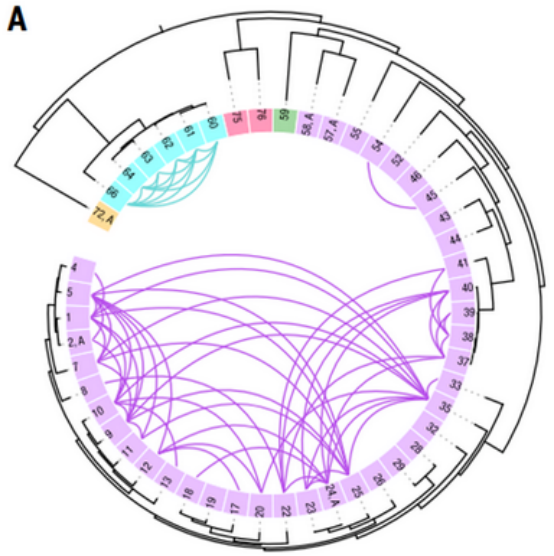
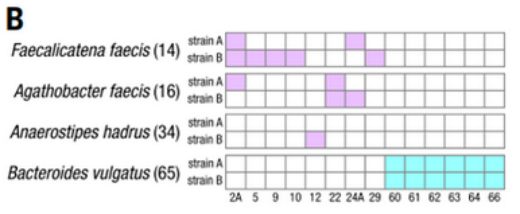
Faecalicatena faecis菌株与不同的菌种交换了基因,而两株普通芽孢杆菌只与其他六个相同的类杆菌菌种交换基因
4、菌株水平的宿主-噬菌体关联
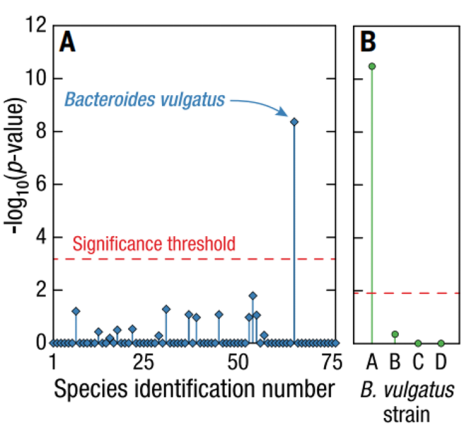
基于微流控液滴的方法不仅封装了单个细菌,还封装了与之物理共处的噬菌体。本次实验检测到了crAssphage的存在,crAssphage是目前从人体肠道微生物组中发现的最丰富的噬菌体。为了探索微生物与噬菌体之间的关联,作者团队将每个SAG中的reads与crAssphage基因组进行比较,发现数十个SAG包含很大一部分reads可以与crAssphage匹配,且这些SAG还包含大量不与crAssphage基因组匹配的reads,而是与细菌分类群匹配,将这些reads与76个物种的组装基因组进行比对,以确定哪些细菌物种可能与该人类供体中的crAssphage菌株相关。
在这几十个包含相当一部分与crAssphage匹配的序列的SAG中,有14个SAG只与一个物种--普通拟杆菌相关。证明普通似杆菌是该个体中噬菌体crAssphage的体内宿主物种,如下图A。图B则更精确地显示了普通似杆菌菌株A与噬菌体crAssphage存在明显的关联。
总结
Microbe-seq技术将微流控技术应用于微生物领域,配合特殊的生物信息学分析手段,无需传统手段或前参考即可以精准地组装出高质量的菌株水平基因组。同时,该技术还可精确解析菌株水平基因组、发现未培养的潜在的新菌株,在菌株水平探究水平基因转移现象、宿主-噬菌体关联、功能基因和代谢通路问题。
单细胞技术在微生物领域中具有广泛的研究前景,无论是土壤还是海洋,亦或是是植物与动物中的微生物,该文章为广大微生物学者们打开了新思路、提供了新方法,Microbe-seq有望成为单细胞微生物研究的主流技术,从自然菌群中完成单细胞测序及分析研究的那一天已经指日可待了。