
2022-10-09
细胞调控其基因表达的一种方法就是在DNA中添加化学修饰,从而决定哪些基因表达,哪些基因沉默,DNA甲基化就是其中一种化学修饰。DNA甲基化在原核生物的病毒防御、错配修复、基因调控和发病机制等方面起着重要作用。
随着表观遗传调控机制研究的不断发展,人们发现在原核生物中有三种不同形式的DNA甲基化:第一种是标记DNA碱基腺嘌呤(6mA);另外两种是标记DNA碱基胞嘧啶(4mC和5mC)。虽然有证据表明甲基化在转录调控中的功能作用,但其如何协调基因表达以确定表型仍然难以捉摸。就是由于缺乏全面基因组甲基化组分析方法。目前大多数DNA甲基化分析测序技术都是针对5mC开发的,例如亚硫酸氢盐测序,只有少数方法可以同时检测三种DNA甲基化,如单分子实时测序技术(SMRT-seq)和纳米孔测序技术(Oxford Nanopore),虽然它们已被用来检测DNA甲基化,但SMRT-seq缺乏可以交叉验证结果的开源生物信息学工具,此外有结果表明SMRT-seq可能高估了细菌基因组中的4mC;而Oxford Nanopore测序检测DNA甲基化信号,尤其是6mA会存在噪声,仍需要完善。
因此,为了帮助全面了解微生物表观基因组学,2022年5月30日吴涛教授实验团队在《Genome Biology》期刊上发表了一种基于化学试剂的测序方法—NT-seq(亚硝酸盐处理后进行下一代测序),该方法可以同时检测并量化单个细菌物种和宏基因组环境中的三种不同类型DNA甲基化标记,并且可以与DIP-Seq结合在单碱基分辨率下以高保真度检测6mA,并消除DIP-seq中的假阳性位点。
接下来,带大家解析这篇文章:
技术原理
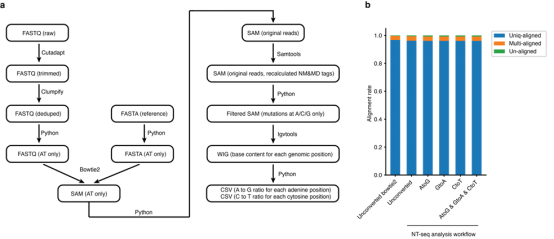
通过对亚硝酸盐和2’-脱氧腺苷和2’-脱氧胞苷的反应产物进行HPLC分离和质谱分析,结果表明了在亚硝酸盐处理条件下,腺嘌呤、胞嘧啶和5mC脱氨,分别产生肌苷(I)、尿嘧啶(U)、胸腺嘧啶(T),在PCR扩增时聚合酶会将肌苷(I)读作鸟嘌呤(G),尿嘧啶(U)读作胸腺嘧啶(T),碱基发生改变,此外5mC在亚硝酸盐处理中的脱氨率比胞嘧啶高4.5倍,可借此区分胞嘧啶和5mC;而6mA和4mC脱氨基会产生亚硝基化6mA(6mA-NO)和亚硝基化4mC(4mC-NO),在PCR扩增时聚合酶会将6mA-NO和4mC-NO分别读作腺嘌呤(A)和胞嘧啶(C),碱基未发生改变,如图1-a所示。 在此基础上设计了一个实验流程来验证亚硝酸盐处理可用于开发一种可以同时检测全基因组三种类型的甲基化的测序方法。首先杂交了两种保护性寡核苷酸,它们与单链DNA的引物区域反向互补,以此保护碱基免于脱氨,而未保护的序列中的碱基在亚硝酸盐处理下,腺嘌呤、胞嘧啶和5mC碱基会发生变化,而6mA和4mC碱基不会发生改变,如图1-b所示。并在此实验过程中确定了2.3%乙酸、1M亚硝酸钠(pH=4.187),并在37℃下孵育为最佳处理条件以达到最高的脱氨效率。同时为了对基因组DNA进行甲基化分析,需要将NT-seq读数与参考基因组对齐,因此开发了一个NT-seq分析流程,该分析管道可以容忍亚硝酸盐处理引入的所有可能的碱基变化(A到G、C到T或G到A),如图2-a所示。 图1 NT-seq实验原理图 图2 Nt-seq分析流程 使用NT-seq检测寡核苷酸中的6mA、4mC和5mC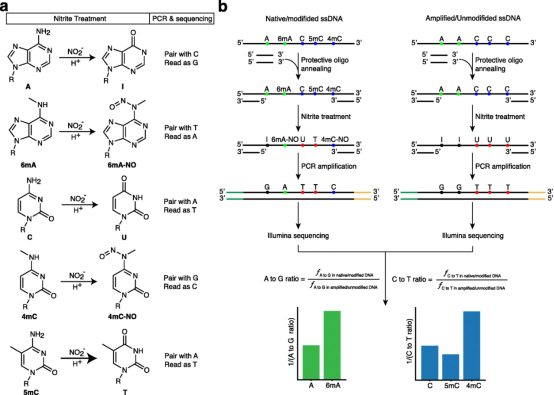
采用2021年发表的亚硝酸盐介导的DNA测序方法对修饰的6mA、4mC和5mC和未修饰的寡核苷酸进行了初步试验。发现6mA位置,A与G比率约为18倍低于其他腺嘌呤位置,并且发现6mA位置归一化的A到G频率与6mA百分比线性相关,表明NT-seq可以精确量化6mA频率。而4mC和5mC的分析结果与6mA类似,除了5mC位置的C到T比率比其他胞嘧啶位置高约40%,这与先前的研究一致,表明5mC比C更容易脱氨基,如图3所示。证明了NT-seq可以检测DNA寡核苷酸中所有三种类型的DNA甲基化。 图3 NT-seq检测寡核苷酸中的腺嘌呤和胞嘧啶甲基化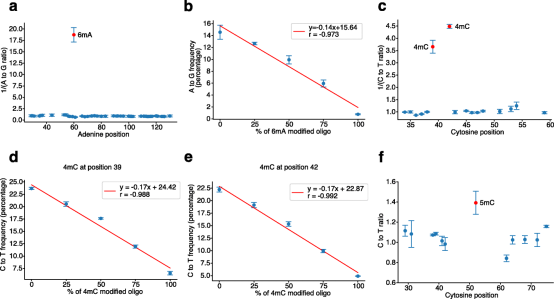
NT-seq检测细菌中的甲基化基序 以大肠杆菌MG1655基因组为实验材料,来验证NT-seq检测基因组DNA中三种甲基化基序的能力。实验结果显示,与未甲基化的腺嘌呤位点相比,已知6mA位点(Dam和M.EcoKI基序)的A到G频率显著降低,而以M.EcoKII基序为阴性对照(甲基转移酶M.EcoKII在实验室条件下不表达),结果显示M.EcoKII基序的A到G比率与未甲基化的腺嘌呤位点没有区别,以上结果表明NT-seq检测的是真正的6mA基序。为了进一步验证该结果,采用基因敲除技术获得hsdM KO(M.EcoKI)和dam/dcm突变菌株进行实验,发现与野生型大肠杆菌菌株相比,hsdM KO和dam/dcm突变菌株的基序与未甲基化腺嘌呤位点的A到G比率没有差异,再次验证NT-seq检测的是真正的6mA基序。 接下来验证NT-seq检测的是真正的4mC和5mC基序。以幽门螺杆菌JP26基因组为实验材料,采用与检测6mA基序相同的方法,结果表明NT-seq确实可用于同时检测细菌基因组中多种类型的DNA甲基化基序。 图4 NT-seq同时检测细菌中的三种甲基化基序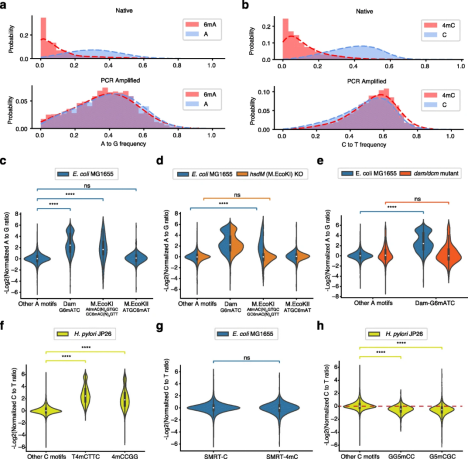
NT-seq可以发现新的甲基化基序 在证明NT-seq可以检测细菌中已知甲基化基序的基础上,作者准备验证NT-seq是否可以发现新的甲基化基序。以幽门螺杆菌JP26基因组为实验材料,收集了所有可能的甲基化的腺嘌呤基序,根据这些基序的A到G比率分为两组:上组中大多数是已知的6mA基序,下组主要包含未甲基化基序,其中有少数例外。在对例外情况中的基序(GMRG6mA)基序的一个子基序(GAGG6mA)进行分析时发现是由于之前的SMRT-seq工作流程不精确地将GAAG6mA和GCRG6mA归类为GMRG6mA基序。 此外,NT-seq也鉴定了一种新的6mA基序(GGWCN6mA),并且该继续未被SMRT-seq鉴定。在对该基序的亚基序分析时发现其与未甲基化基序相比,所有亚基序的A到G比率显著降低。在SMRT-seq IPD比率量化中也观察到类似趋势,表明GGWCN6mA是真正的6mA基序。除此之外也对幽门螺杆菌JP26中的4mC和5mC基序进行了检测,但未能发现新的基序,这可能是由于该基因组中已经没有新的其他胞嘧啶甲基化基序。 综合以上结果,表明NT-seq不仅可以验证报告的甲基化基序,还可以识别细菌基因组中的新的甲基化基序。 图5 NT-seq发现新的甲基化基序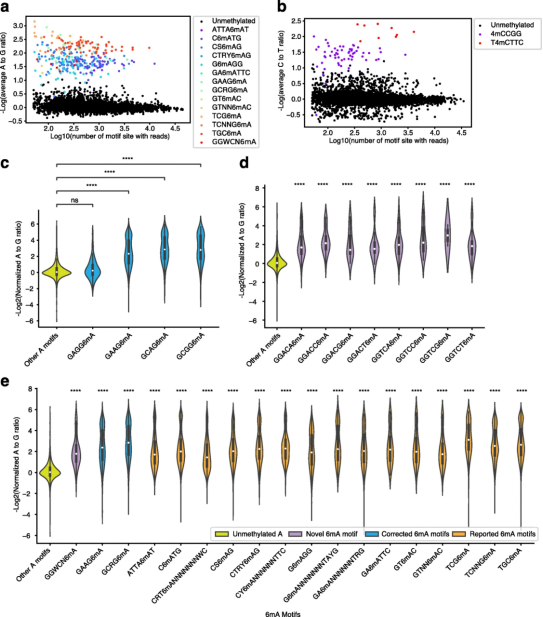
NT-seq鉴定微生物群落中的甲基化基序 以商业微生物群落标准品为实验材料,来确定NT-seq是否可以检测微生物群落中的DNA甲基化。该商业微生物群落中包括八种细菌和两种真菌,由于两种真菌没有报道的甲基化基序,因此将6mA分析集中在八种细菌上,而这八种细菌分别为枯草芽胞杆菌、大肠杆菌、粪肠球菌、单核细胞李斯特菌、金黄色葡萄球菌、肠杆菌和发酵乳杆菌。收集了所有可能的甲基化腺嘌呤基序,发现了在7种细菌菌株中,发现之前SMRT-seq数据与NT-seq数据结果有所差异,例如粪肠球菌和发酵乳杆菌,其中粪肠球菌在SMRT-seq分析中具有微小差异的两个6mA基序,而在NT-seq检测中却没有差异,因此证明这两个基序不一定是甲基化6mA;在发酵乳杆菌中,SMRT-seq检测中没有可用数据,而在NT-seq检测中发现AG6mAGG基序与未修饰的6mA基序相比,A与G比率有所降低,且降低程度相似,同时AG6mAGG的大多数亚基序A到G比率分布显示出与未甲基化基序的明显分离,表明其是真的6mA基序。并且在对总6mA基序丰度分析中发现AG6mAGG基序占75%,是最显著富集的基序,综上结果证明NT-seq检测到的AG6mAGG确实是发酵乳杆菌基因组中真正的6mA基序。 在NT-seq可以检测到微生物群落参考中的6mA的基础上,验证是否可以检测到4mC和5mC,但是在这些细菌基因组中没有检测到4mC,可能是由于这些细菌基因组中没有4mC基序;而对于5mC的话,在对8种细菌的亚硫酸氢盐测序数据分析中在枯草、大肠、金黄色葡萄球菌和肠杆菌中鉴定出6个胞嘧啶甲基化基序,而在NT-seq的测序中这些基序的C与T比率均高于未甲基化基序的比率,这表明这些胞嘧啶甲基化基序是5mC甲基化而不是4mC甲基化,因为5mC的脱氨率比未甲基化胞嘧啶要高4.5倍。因此以上结果可以表明NT-seq可以检测微生物群落中的甲基化基序。 图6 微生物群落参考中的甲基化基序检测 DIP-NT-seq可以单碱基分辨率分析6mA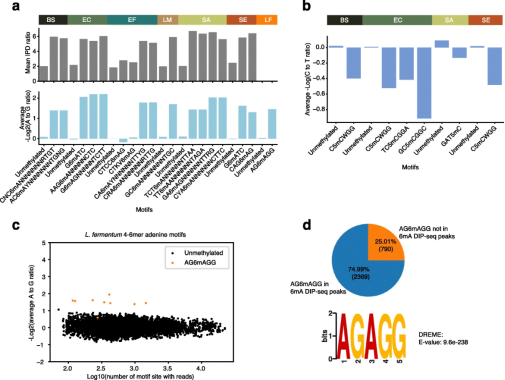
为了评估NT-seq在单碱基分辨率下检测不同类型DNA甲基化性能,作者以幽门螺杆菌基因组中的每种甲基化类型生成了ROC曲线。与寡核苷酸结果一致,NT-seq在单碱基分辨率下检测6mA和4mC的性能相似,5mC检测的性能显著下降。为了探索NT-seq在单碱基分辨率检测甲基化的性能是否可以进一步提高,将6mA-DIP与NT-seq结合并在大肠杆菌基因组中进行了测试,同时为了全面评估NT-seq在检测6mA位点的性能,使用Dam/M.EcoKI基序作为6mA位点的标准,生成了未富集和 DIP 富集样品的 ROC 曲线。一致地,DIP 富集极大地提高了不同测序深度阈值下的 AUC 分数。 为了进一步验证DIP-NT-seq的性能,将DIP-seq与成熟的SMRT-seq方法在6mA单碱基检测上进行比较,我们发现DIP-NT-seq和SMRT-seq结果是一致的。此外,使用Dam/M.EcoKI介导的6mA作为实验对象,DIP-NT-seq可以检测到SMRT-seq中高达76.8%的假阴性位点,而SMRT-seq中仅检测到0.3%的假阳性位点,表明DIP-NT-seq能够有效过滤从SMRT-seq中识别出的假阳性6mA。除了SMRT-seq,我们还将DIP-NT-seq与其他一些基于高通量测序的大肠杆菌6mA 检测方法进行了比较,例如6mA DIP-seq和6mACE-seq。比较结果显示DIP-NT-seq 不仅可以将传统的 DIP-seq 提高到单碱基分辨率,还可以提高 6mA 检测的灵敏度。 为了确定 NT-seq 是否可以提高 DIP-seq 的特异性和分辨率,我们在野生型和MTA1突变的伪尖毛虫中进行了DIP-NT-seq。与6mA DIP-seq和SMRT-seq 结果一致,MTA1 突变体中的 6mA 水平比野生型伪尖毛虫低约 50%,表明 DIP-NT-seq 可以稳健地检测 6mA 变化在真核基因组中以单碱基分辨率。DIP-NT-seq 检测到的 6mA 的 SMRT-seq IPD 比率显着高于 DIP-NT-seq 未检测到的 6mA,表明 DIP-NT-seq 检测到的 6mA 位点可以在 SMRT-seq 中交叉验证。 图7 在细菌和真核基因组中以单碱基分辨率进行6mA分析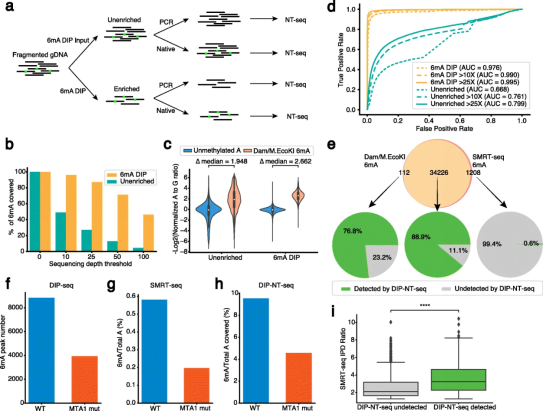
讨论与总结 NT-seq可用于同时检测基因组中所有三种主要类型的甲基化基序,并且NT-seq不仅可以检测已知的甲基化基序,还可以发现新的基序。并在此基础上证明NT-seq可用于在单个细菌物种和微生物群落中从头发现甲基化基序。此外,实验结果表明耦联甲基DNA免疫沉淀(DIP)和NT-seq可以在原核生物和真核生物中以单碱基分辨率分析6mA,并且在大肠杆菌中,DIP-NT-seq与SMRT-seq检测性能相当,另外DIP-NT-seq可以从DIP-seq或SMRT-seq测序中过滤掉假阳性的6mA位点,提高了6mA检测的准确性。 除了以上这些优点之外,NT-seq还有一些限制。首先,目前NT-seq中的亚硝酸盐处理会导致DNA损伤,这给文库生成带来了麻烦,使我们无法将A和C完全脱氨以达到更好的性能。其次,NT-seq读数偏向于G-poor区域,这可能会限制NT-seq在富含GC的细菌基因组中的应用。最后,NT-seq的5mC检测效率低于亚硫酸氢盐测序。这些限制条件可以通过寻找温和的亚硝酸盐处理条件来克服,以实现接近完全的脱氨率,同时减少DNA降解。 综上所述,吴涛教授实验团队开发了一种NT-seq方法,可以同时检测原核基因组中所有三种主要类型的DNA甲基化。可以准确检测单个物种和微生物群落样本中的甲基化基序。与SMRT-seq相比,NT-seq为细菌甲基化作图提供了一种经济高效的解决方案,这将促进细菌表观遗传学的研究。通过耦联甲基 DNA 免疫沉淀 (DIP),我们发现 NT-seq 可以在细菌基因组中以单碱基分辨率准确分析 6mA,这也可以应用于真核基因组,以帮助消除 6mA DIP-seq 中的非特异性信号。NT-Seq 可以交叉验证 SMRT-Seq 结果并生成更多训练数据集,用于开发用于甲基化分析的机器学习工具。