
2022-10-20
空间转录组分析可以揭示组织结构和环境依赖性细胞-细胞或细胞-环境相互作用的分子景观。受限于当前的技术手段,获取单细胞分辨率的全基因组空间转录组信息仍具有挑战性。在这里,我们介绍了一篇Science Advances上发表的方法文章,文章开发了iSpatial这一新算法,通过整合空间转录组和单细胞转录组信息,推导整个转录组的空间模式。
摘要 iSpatial算法通过整合空间转录组和单细胞转录组信息,推导整个转录组的空间模式。与现有的其它方法相比,iSpatial在预测基因表达和空间分布具有更高的准确性。此外,它减少了原始数据中的假阳性和假阴性信号。通过对多个空间转录组数据集测试iSpatial,证明了它对来自不同组织和不同技术的数据的广泛适用性。因此,这项研究提供了一种以单细胞分辨率揭示整个转录组空间组织的计算方法。伴随着许多高质量数据集的产生,iSpatial为了解复杂组织和疾病过程的结构和功能提供了独特的方法。
iSpatial概述 FISH和基于原位测序的ST技术,例如MERFISH、seqFISH、osmFISH以及STARmap,可以在单细胞水平同时揭示基因表达和位置信息,但是预定义的基因靶点有限(图1左)。另一方面,scRNA-seq可以无偏地分析整个转录组,但是不提供空间信息(图1中)。通过整合两种方法的单细胞基因表达谱(逐个细胞的基因矩阵),可以根据scRNA-seq数据推断出每个空间图谱细胞中非目标基因的缺失信息,从而得到图谱细胞的全基因组空间表达信息(图1右)。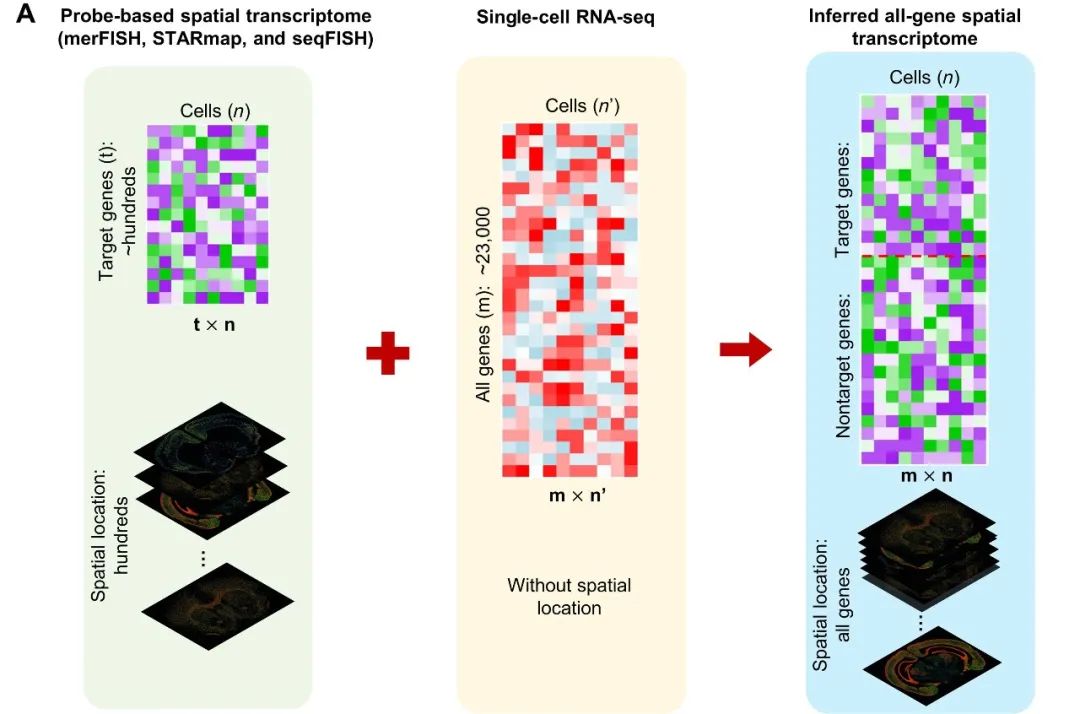
iSpatial设计原理 iSpatial是一种基于R开发的工具,整合了scRNA-seq和ST谱数据,以高空间分辨率推断每个基因的表达模式。①首先对scRNA-seq和ST数据分别进行降维处理;②进行表达稳定化处理:根据主成分分析(PCA)空间中的相邻细胞的表达水平,去除潜在的噪音/背景表达;③两个数据集的归一化处理,并通过RPCA+Harmony两轮连续的整合嵌入到一个共同的空间;④在共嵌合的基础上,使用加权的K-邻近(KNN)模型推断出ST数据集中每个细胞中的每个基因的表达值;⑤细胞的物理空间位置由ST数据确定;⑥形成一个新的单细胞基因表达谱,同时具有全基因组覆盖和单细胞空间分辨率;⑦新的表达矩阵可用于下游分析,包括识别空间可变基因(SVG),空间表达谱分析等。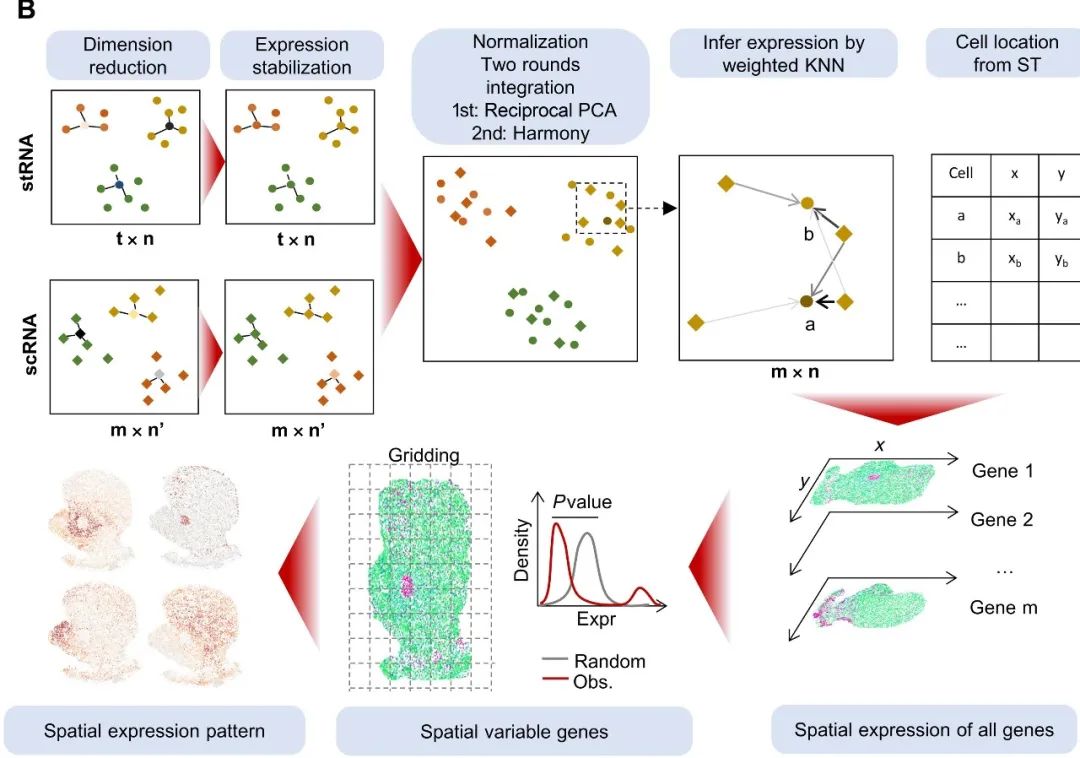
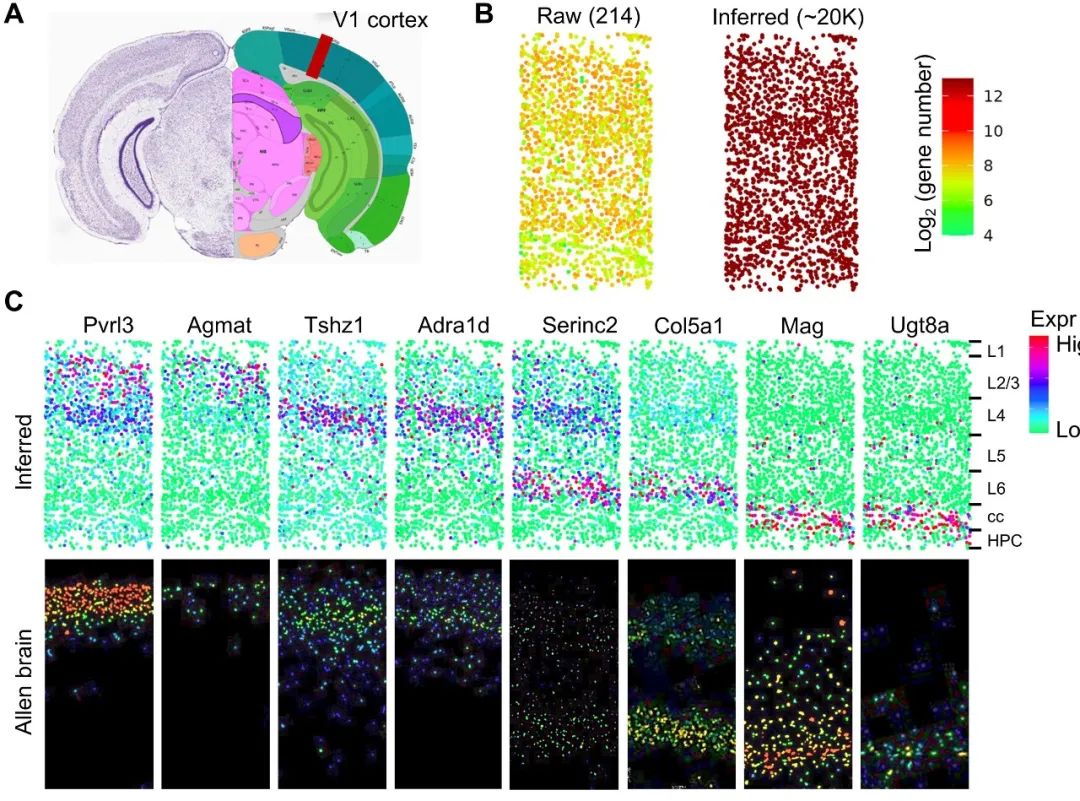
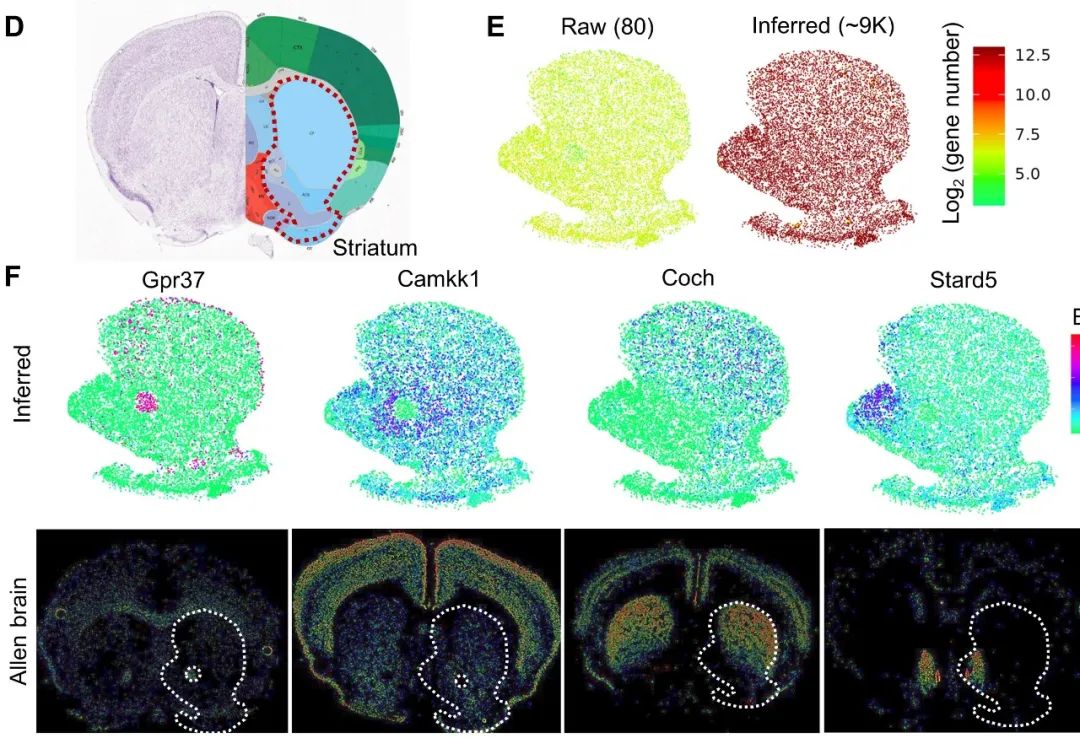
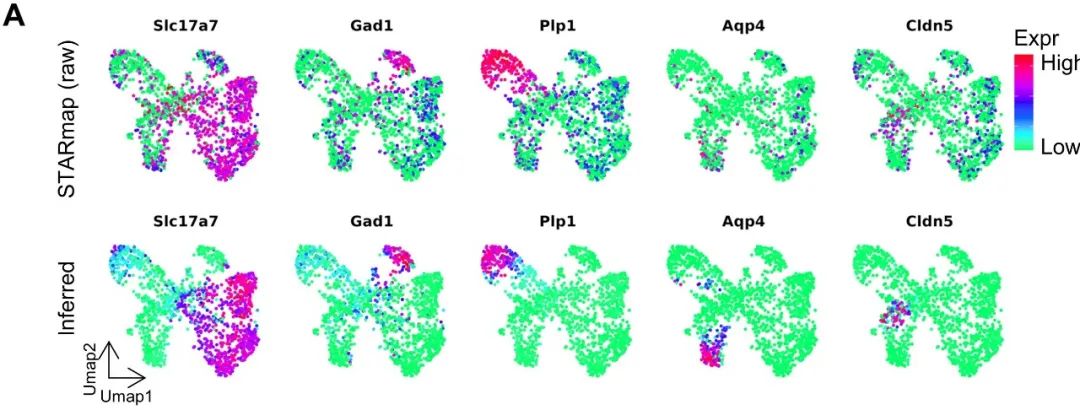
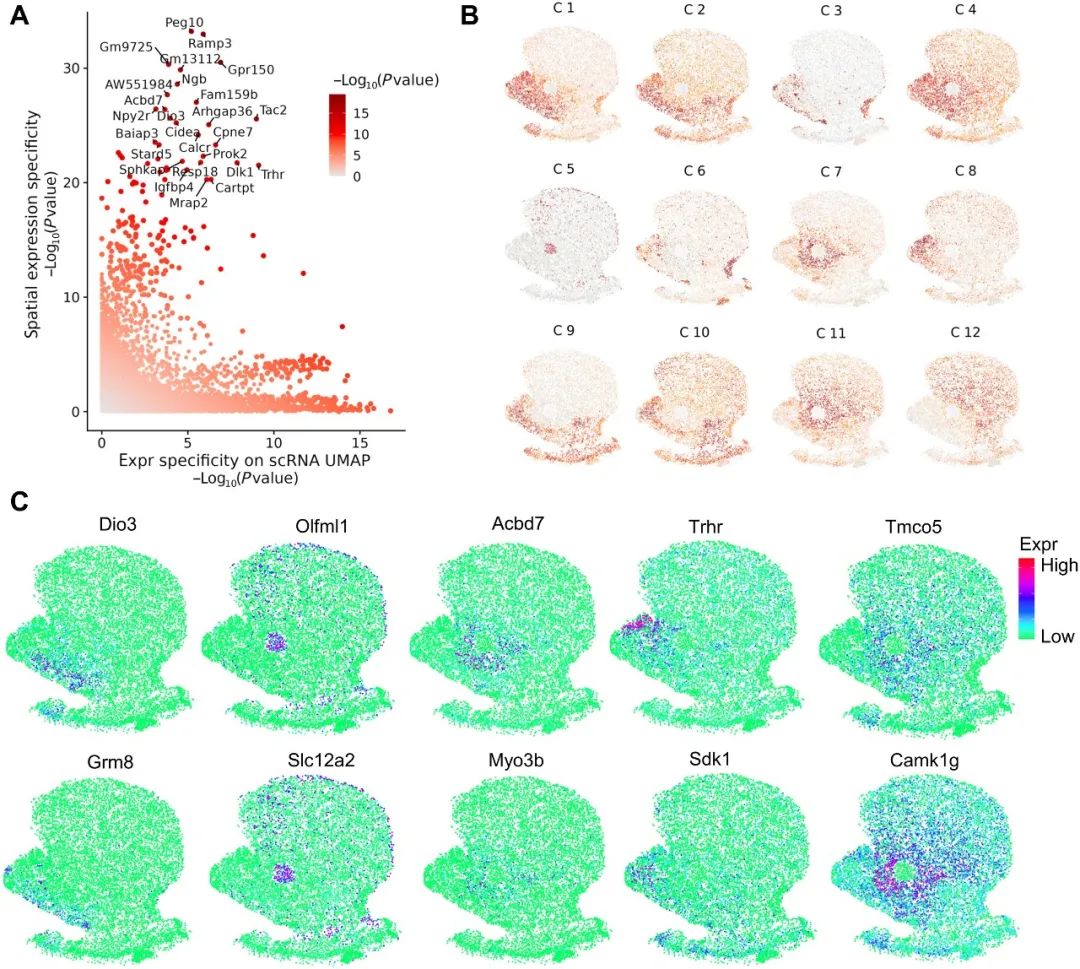
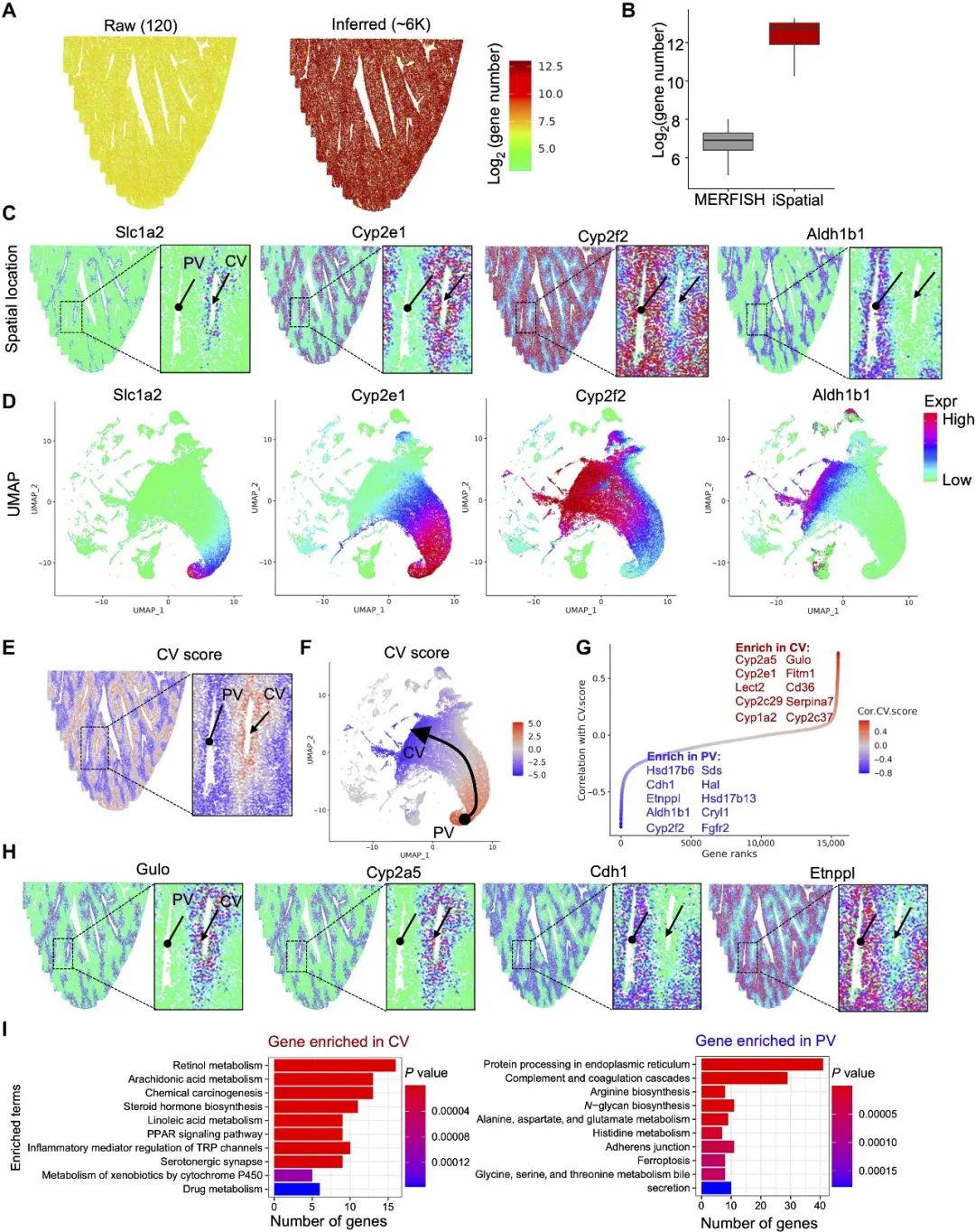
iSpatial性能测试 研究团队使用不同组织和技术产生的多个ST数据集测试了iSpatial。与其它现有方法相比,iSpatial在预测基因表达和空间分布方面具有更高的准确性。此外,它还减少了原始数据集中的假阳性和假阴性信号。同时它对来自不同组织和不同技术的数据集具有广泛适用性。 1.iSpatial在预测空间表达模式的准确性方面优于现有工具 测试数据:从Slide-seq V2生成的小鼠海马体空转数据集。随机抽取3000个基因作为训练数据集,其他基因作为验证数据集。然后与另一个配套的单细胞数据集整合,使用iSpatial推断形成新的数据集。 通过将推断的表达模式与Slide-seq(验证数据组)确定的“真值”进行比较,我们发现iSpatial可以高精度地预测空间表达模式。例如,iSpatial推断出Atp2b1,Prox1和Fibcd1的表达。iSpatial可以“增强”原始数据中未被很好地检测到的信号。例如,Slit1,Tspan18,Efnb2,Car12等几乎无法在海马细胞中通过Slide-seq检测到。 随后开发团队使用Slide-seq数据集进一步比较了iSpatial与另外两个现有工具Liger和Seurat在同一任务上的性能:与iSpatial相比,从Liger和Seurat获得的空间模式在较高背景下更模糊;在不同表达水平的所有基因组中,iSpatial表现出明显高于其他方法的相关系数和较低的RMSE,而且预测的准确性与基因表达水平呈正相关。此外,细胞类型特异性表达的基因表现出更高的预测准确性。这一结果表明,iSpatial在功能相关的基因上实现了更高的预测准确性。当比较从原始Slide-seq数据识别的SVG和从不同方法推断的数据识别的SVG时发现iSpatial在三种方法中对SVG具有最高的预测能力。 开发团队还使用了成年小鼠冠状半脑切片的Stereo-seq数据来衡量iSpatial的性能。结果显示,iSpatial在验证数据集上取得了比其他方法更高的相关性。 2.iSpatial广泛适用于不同的ST数据集 测试数据:使用iSpatial来分析覆盖小鼠大脑初级视觉皮层(V1)的STARmap数据集。原始的STARmap数据仅包含1020个基因靶点,但是iSpatial推断出超过20,000个基因的表达。重要的是,不仅检测到这些在STARmap没有检测到的基因,还能定量其表达量。 测试数据:小鼠纹状体MERFISH数据集。原始的MERFISH数据集包含253个靶基因,可以识别纹状体中的九种主要细胞类型,其中175个靶基因在某些细胞类型中表现出显着的富集。通过将该数据集与相应的scRNA-seq数据集整合,iSpatial可以在单细胞分辨率下推断出约9000个基因的表达和位置,其中超过2200个基因被鉴定为细胞类型特异性表达基因。推断基因的空间模式与ISH确定的空间模式基本一致。 通过将iSpatial应用于来自不同组织(海马、大脑半球、皮层、纹状体和肝脏)和用不同技术(Slide-seq、Stereo-seq、MERFISH和STARmap)生成的数据集,在每个数据集中都发现了已知和以前未知的空间表达模式,表明iSpatial广泛适用于分析不同ST数据集。 3.iSpatial减少来自空间转录组的FP和FN信号 尽管相比于基于测序的ST技术,基于图像的ST技术具有更高的检测效率,但是这种技术的性能表现高度依赖于基因探针的特性。例如,一些一些转录本太短,无法被足够的探针靶向到,因而产生假阴性(FN,dropout)。另一方面,一些基因由于具有相近的同源物,因此可能很难通过杂交区分,因而导致假阳性(FP,background)。作者认为iSpatial可以在进行表达预测时,通过给scRNA-seq细胞更高的权重来减少这些错误信号。单细胞测序获得的细胞对检测基因的长度不敏感,而且可以根据序列差异明确区分相似的转录本。 测试数据:比较原始STARmap数据和iSpatial推断数据之间的UMAP上一些成熟细胞类型标记基因的表达模式。我们发现,尽管这些细胞类型特异性标记物在相应的细胞类型中表现出高富集性,但在STARmap分析时,通常存在FP信号(例如,Slc17a7,Gad1,Plp1和Cldn5)。在某些情况下,没有观察到预期的表达模式,可能是由于FN(Aqp4)。而iSpatial推断的结果可以减少FP和FN的发生。 4.iSpatial 支持整个转录组水平的空间分析 ST分析的一个主要目标是识别SVGs,这是不同组织结构/功能异质性的分子基础。将iSpatial应用到STARmap皮层数据以识别SVG,发现iSpatial可以显著增加检测到的SVG数量(从21个增加到2122个),iSaptial可以帮助识别生物学相关的空间基因表达模式。svg的聚类分析揭示了6种主要的空间模式,它们类似于已知的小鼠皮层层组织。值得注意的是,即使我们将分析局限于STARmap的目标基因,iSpatial仍然识别出更多的SVGs(推断数据162个,原始数据21个),这可能是由于原始数据中FP和FN信号的校正。 5.iSpatial改进了肝脏ST数据的分析 为了评估iSpatial在其他组织(除脑组织之外)数据集中的性能,开发团队分析了Vizgen MERFISH小鼠肝脏图谱数据集,iSpatial成功地推断出每个单个细胞中平均6000多个基因的表达,比原始数据增加了20倍以上。推断出的空间模式与现有知识基本一致。在iSpatial推断表达谱的基础上进一步生成了UMAP,发现细胞在UMAP上的位置与它们沿CV-PV轴的原位分布密切相关,显示了沿CV-PV轴的梯度表达谱。值得注意的是,尽管Liger和Seurat也能揭示类似的梯度表达模式,但三种方法的比较表明,iSpatial取得了更高的特异性和准确性,特别是对具有更多空间限制的表达模式的基因。总之,iSpatial可以克服各种ST分析的目标基因数量有限的问题,在不同组织中达到全转录组水平的高精确度。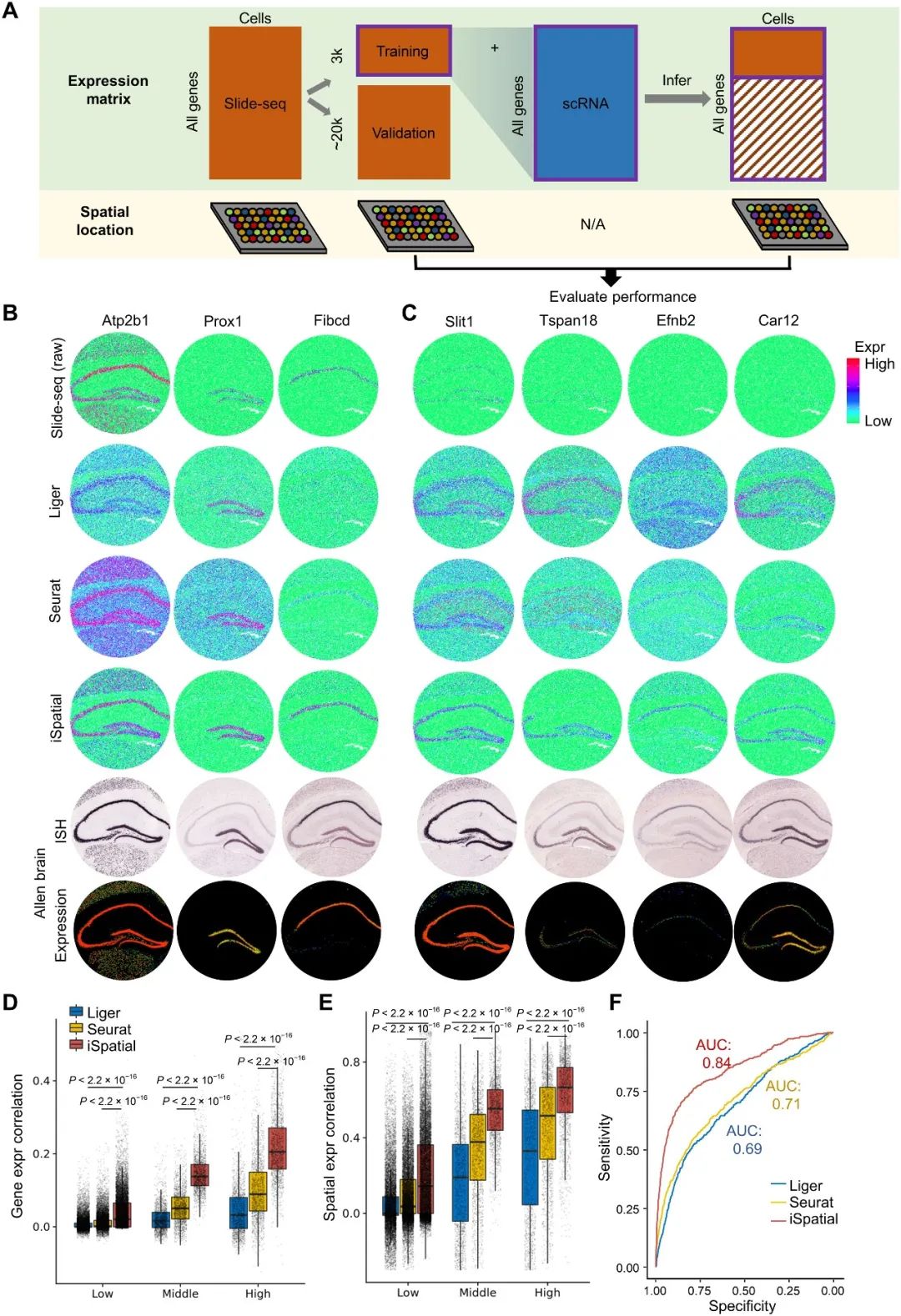
派森诺生物可以提供单细胞转录组和空间转录组以及联合分析等精准服务,欢迎联系我们销售小伙伴们垂询~~~