2022-12-02
宏基因组(Metagenome)和宏转录组(Metatranscriptome)是通过鸟枪法测序技术(Shotgun sequencing),结合全微生物组关联分析(Microbiome-Wide Association Studies,MWAS)的策略,分别从DNA/RNA水平,全面精细地展示整个微生物群落的物种组成谱、功能代谢谱、表达谱,进而从原理上阐明微生物群落在生态系统中发挥作用的根本机制。
对于数据量和信息量都非常庞大的宏组学研究而言,选对分析软件和数据库至关重要。历经多年发展,目前能用于宏基因组和宏转录组分析的软件已然数不胜数,对于分析者而言,可谓“甜蜜的烦恼”!那么,在琳琅满目的分析方法中,究竟哪些才是当前宏组学的“网红”呢?本期,我们为您推荐以下软件和数据库,大家走过路过千万不要错过哦~
MMseqs2
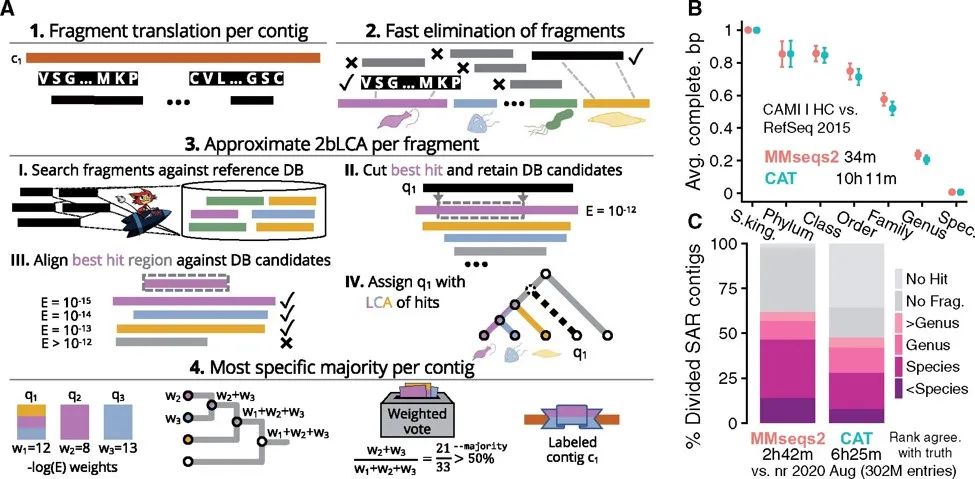
MMseqs2 (Many-against-Many sequence searching,https://github.com/soedinglab/mmseqs2)是一款集核苷酸、蛋白质序列检索、回帖、聚类、注释等功能模块于一体的强大软件。今天,我们就来介绍MMseqs2 的taxonomy模块。作为宏基因组微生物物种注释的新工具,MMseqs2_taxonomy发表在2021年的Bioinformatics上(https://academic.oup.com/bioinformatics/article/37/18/3029/6178277?login=false)。

对于待注释的氨基酸/核酸序列,MMseqs2_taxonomy 首先通过其search模块提取数据库中所有可能的氨基酸序列片段,并快速保留有助于分类注释的片段,再通过加权方式确定其分类身份(如下图A)。其亮点是在于提出了2bLCA (dual BLAST based last common ancestor) 和 a-2bLCA (Approximate 2bLCA) 算法,来解决LCA算法在实际操作过程中如何确定备选序列数目的难题,在确保稳健性的同时,显著提高了宏基因组物种分类注释的精准度(如图C)。
为进一步展示a-2bLCA算法的优势,小编特意使用宏基因组的实际数据,将该算法与LCA和TopHit算法进行了比较(如下图),图中是s1-5表示采用的灵敏度等级。结果显示,TopHit算法虽然获得了精细的注释信息(更多注释到了species水平),但由于是单一选择匹配度最高的注释结果,其结果并不稳健;而将a-2bLCA的结果与LCA进行比较,可以发现2bLCA算法能精确到species水平的序列更多。
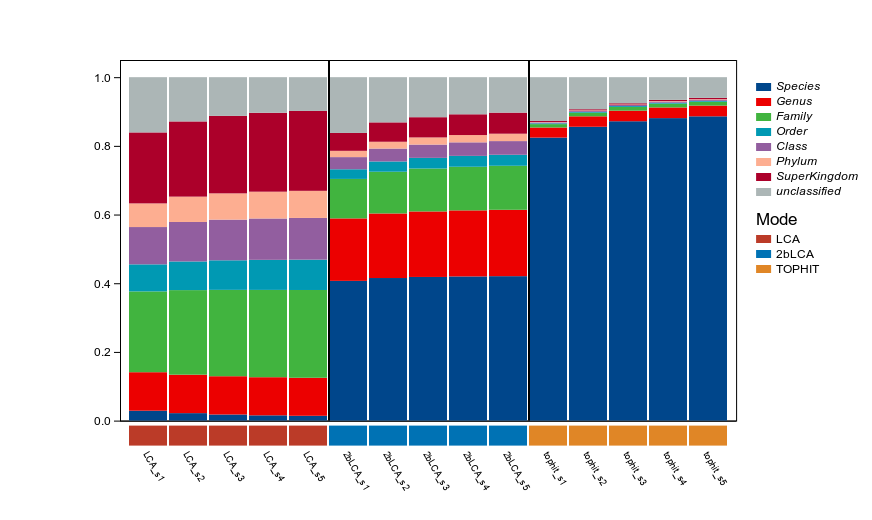
LCA vs. 2bLCA vs. TopHit算法在不同分类水平下注释到的序列比例
GTDB基因组分类数据库
GTDB基因组分类数据库(https://gtdb.ecogenomic.org/),全称Genome Taxonomy Database,它基于大量基因组系统发育分析构建的基因组分类标准流程体系,对现有的大量微生物基因组进行了重分类。该数据库已经于2018/2020连续发表两篇Nature Biotechnology,配套软件GTDB-tk于2019发表于Bioinformatcs,在提供微生物分类参考数据库的同时,也提供了微生物基因组物种分类、进化树构建的全套工具,绝对是服务到家的好工具。
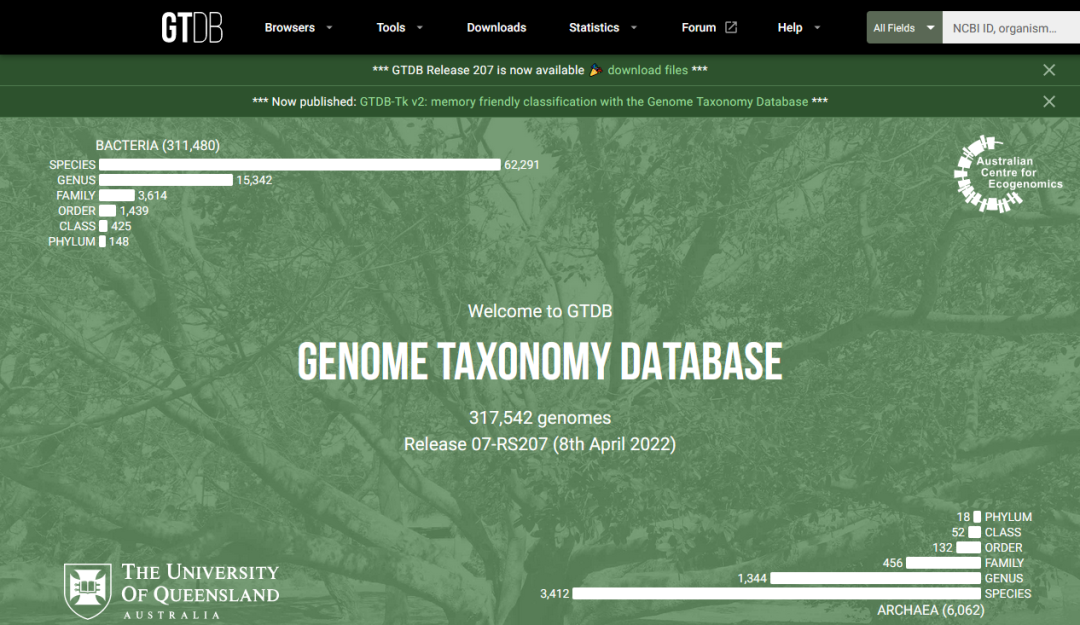
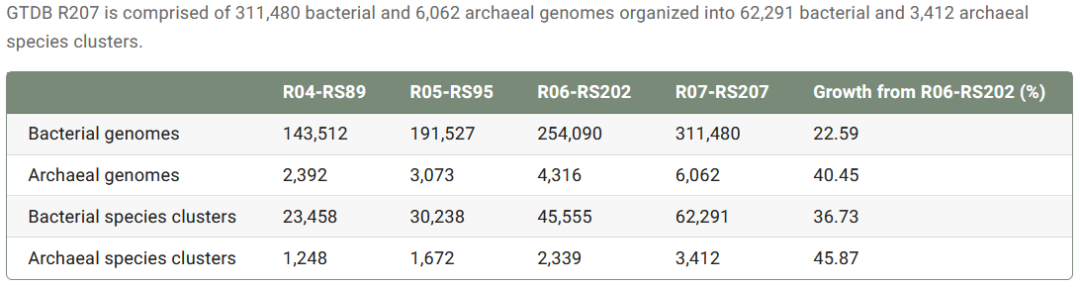
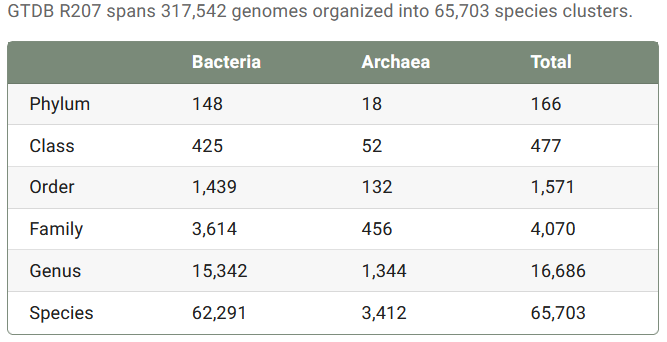
目前该数据库涵盖了317542个微生物基因组,由311480个细菌和6062个古菌基因组组成(如下图);并分为65703个种水平分类单元,由62291个细菌和3412个古细菌构成(如下图)。
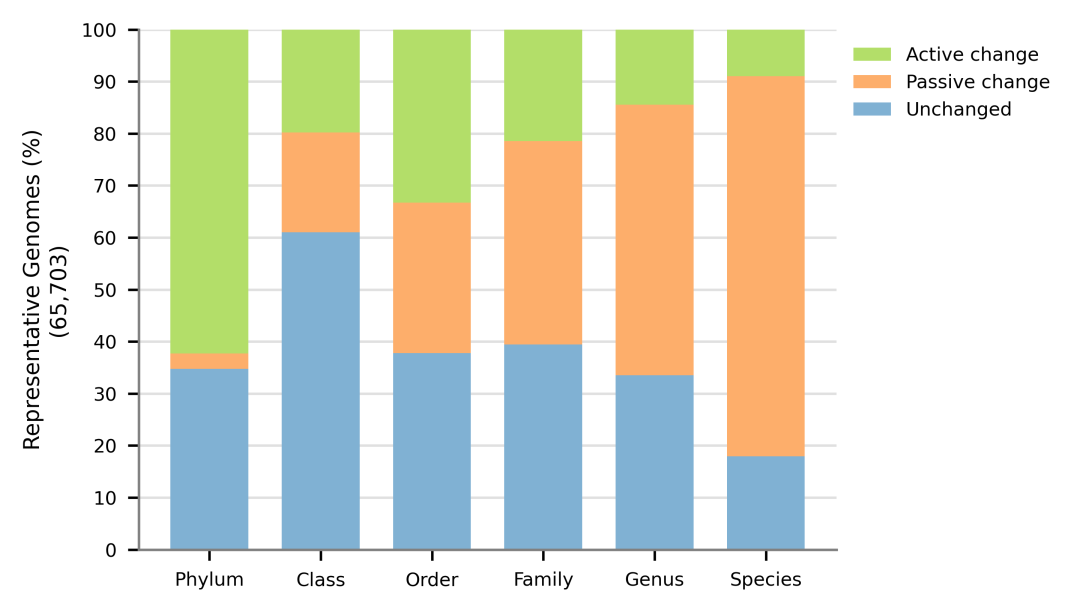
下图显示了GTDB的65703个代表基因组的新注释结果与其在NCBI分类系统中的差异分布。如果一个基因组的新物种名称与其在NCBI分类系统中相同,则标注为Unchanged(不改变其分类);如果NCBI中没有物种分配,则标注为Passive change(被动更改);如果其新物种名称与NCBI分类不同,则标注为Active change(主动更改)。
我们再使用数据实测一下,使用GTDB-prot(GDTB基因组数据库中提取的蛋白序列数据库)和NCBI-nr数据库进行蛋白序列注释的效果(如下图,注意,我们只统计了原核部分),可以发现GTDB与2bLCA的组合结果最为高效,且GTDB相比于NR数据库在原核微生物注释的精细程度方面更具优势。
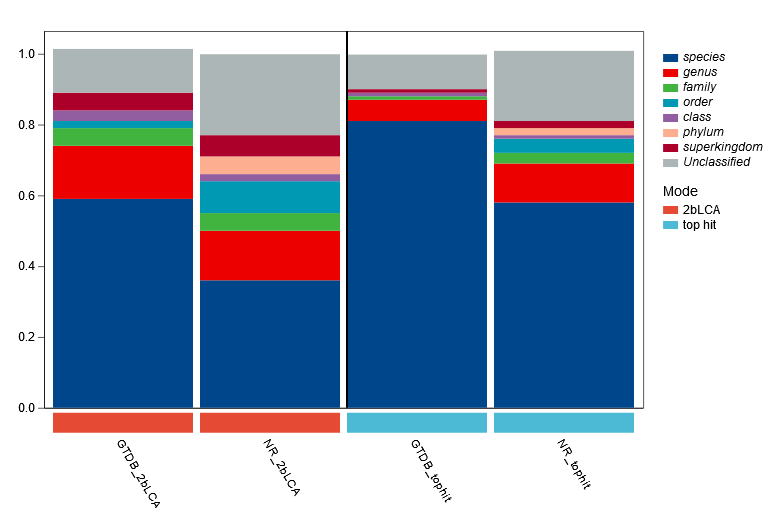
GTDB数据库与NR数据库2bLCA vs. TopHit算法的结果比较
MMseqs2在其发表的文章中声称比CAT工具快2-18倍,而CAT在今年推出的更快的diamond2 软件 (这个软件我们会在下一期具体评测) 的加持下,在速度上也许可以超过MMseqs2。尽管如此,为了使用2bLCA算法,以便获得更准确的注释信息,我们仍旧推荐使用MMseqs2_taxonomy作为宏基因组微生物物种注释的最佳方法。
此外,由于GTDB数据库中仅有原核生物的信息,若想获得更为全面的注释结果,或许可以考虑将NCBI-nr中的真核序列、病毒序列与GTDB-prot进行整合,这样运用于宏基因组物种注释将更为得心应手。
以上就是我们本期精心整理的宏组学软件和数据库推荐,在此给大家抛砖引玉啦~这些软件和数据库,都将纳入派森诺最新宏组学分析流程中,欢迎大家尝鲜体验!


