
2022-11-30
在这个飞速发展的测序时代,DNA和RNA测序已经逐渐成为“实验室中的家常菜”。若要评选出目前最受欢迎的一道菜,那恐怕非单细胞RNA测序莫属。
研究背景
以往,研究人员通常利用RNA测序(RNA-seq)来检测样本中的所有RNA转录本,以发现新型RNA,或开展基因表达分析。不过,测序的对象往往是组织样本或细胞群,这使得细胞之间的差异有可能被平均值所掩盖。作为独一无二的个体,细胞也许并不乐意自己的光芒被掩盖。于是,单细胞测序应运而生。自2013年被《Nature Methods》评为年度技术以来,这项技术正被迅速地采用,发表文献的数量也在逐年递增。单细胞RNA-seq(single-cell RNA sequencing)技术作为一项革命性工具,在单细胞水平上对转录组进行分析,已经被广泛用于多个方面的生物医学研究,包括肿瘤异质性研究、新细胞类型的鉴定、组织发育与细胞分化过程研究和基因调控网络研究等。
细胞外囊泡(EV)是细胞分泌的脂质双层膜结构的囊泡状小体,直径约为50~1 000 nm。EV广泛存在于细胞培养上清液、血清、血浆、唾液、尿液、乳汁等体液中,其携带蛋白质、脂类、DNA、RNA,参与细胞间信号传递、物质交换、免疫调节等。目前,EV被认为是疾病诊断中潜在的无创生物标志物和药物传递载体。EV是内源性细胞产物,需要亲代细胞来源才能获得,因此EV存在高度异质性。
传统的检测EV货物分子的方法(如PCR、RNA测序)通常借助于批量分析确定EV内物质含量,但缺失了单个EV分子量和EV间异质性的信息。即便是后来的数字PCR、结合qPCR的流式细胞术等可以检测单个EV的货物分子量,但仍缺乏单个EV RNA图谱。因此,在单个EV水平上探索转录组特征和异质性至关重要。
单细胞RNA测序(scRNA-seq)技术使得能够在单细胞水平检测基因表达,以及检测细胞异质性和细胞类型。在此基础上,此篇旨在建立一种实验和分析方法来进行单细胞RNA测序以揭示单个EV的转录组特征。
接下来,带大家解析这篇文章:
技术原理
单细胞 RNA 测序(Single cell RNA sequencing,scRNA-seq)是一种在单细胞水平上利用 RNA 测序对特定细胞群体进行基因表达谱定量的高通量实验技术。待测组织经过单细胞分离、RNA 提取、逆转录、文库构建和测序,便可利用数据分析获得多个细胞的基因表达谱。其技术原理主要包括单细胞分离、扩增测序和数据分析3方面。
10×genomics技术:首先凝胶微珠上连接上特定的DNA片段,DNA片段由三部分组成:Barcode、UMI、PolyT组成。Barcode是由16个碱基组成。一共有400万种Barcode,一个微珠是对应于一种Barcode,通过这400万种Barcode,可以把凝胶微珠给区分开。UMI是一段随机序列,也就是说每一个DNA分子,都有自己的UMI序列。10bp的UMI,有100万种序列的变化(4^10 = 1,048,576),UMI的作用是为了区分哪些reads是来自于一个原始cDNA分子,区分基因片段重复还是duplication及区分是真实的SNP位点还是PCR产生的突变。通过10×genomics仪器将单个细胞与单个凝胶微珠通过油相混在一起,形成油包水的小微滴,接下来把细胞膜破掉,让细胞当中的mRNA游离出来。游离出来的mRNA与小液滴中的水相混合,也就是和逆转录酶、结合在凝胶微珠上的核酸引物、以及dNTP底物相接触,进行后去反应。
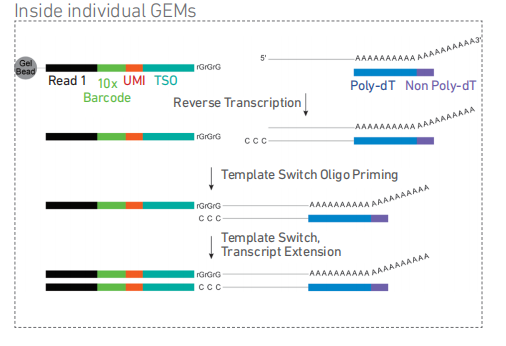
图1 单细胞RNA测序技术原理
单个EV的转录组特征分析
文中实验选取人慢性髓系白血病细胞K562、人脐带间充质干细胞MSC,并使用差速离心法从K562和MSC培养液中分离EVS。将分离出的EVS进行了鉴定;随后有使用流式细胞术进行计数,之后再进行scRNA-seq。
1、EVS的特性及浓度分析
首先采用差速离心法分离K562和MSC细胞来源的EVS。如图2a所示,通过透射电镜观察到具有膜结构的球形囊泡。纳米粒子跟踪分析证实EVS的粒径分布为100-1000nm,大多数在100-200nm范围内(图2b)。通过EV进行标记标记,包括跨膜蛋白(CD9)、胞浆蛋白(Alix和TSG101)和微囊泡特异性标记蛋白Annexin A1(图2 c)来验证EV的有效性。为了用流式细胞术测量EV浓度,用100、300、500和1000 nm的校准珠确定EV门控策略(图2d),并通过钙黄绿素-AM染色来确定完整的EV。钙黄绿素-AM标记完整的EV但不标记膜碎片或其他碎片。在P6点处记录K562-EV和MSC-EV样本的钙黄绿素阳性事件浓度(图2 e,f)。EVS被定义为钙黄绿素-AM阳性事件,直径在100~1000纳米之间(命名为K562-EV1和MSC-EV)。由于过滤磷酸盐缓冲盐水(PBS)中的颗粒大多小于300纳米,这可能导致通过10x基因组学捕获EVS的概率下降,然后将大小为300-1000纳米的钙黄绿素-AM阳性颗粒(命名为K562-EV2)作为对照(图2e)。为了进一步证明钙黄绿素AM标记的完整的EVS,用Triton X-100洗涤剂裂解EVS的磷脂双层。如图2g所示,经过2%的Triton X-100处理后,P6点的事件率显著降低,证实这些事件是EVS而不是碎片。
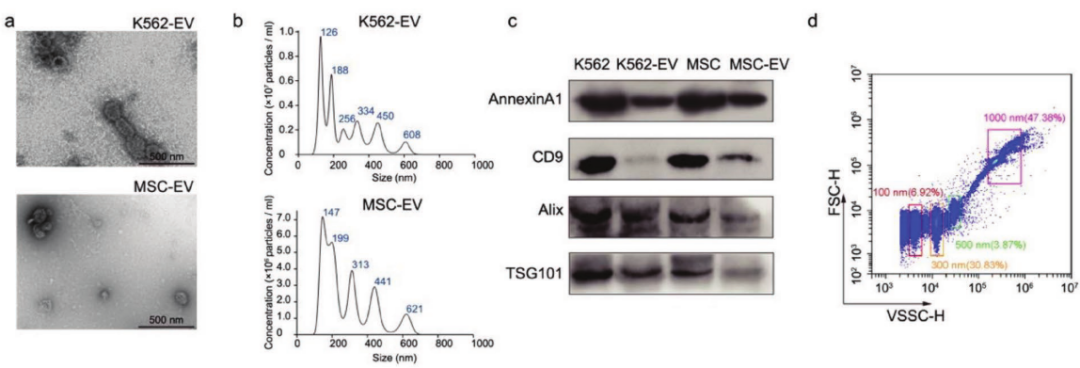
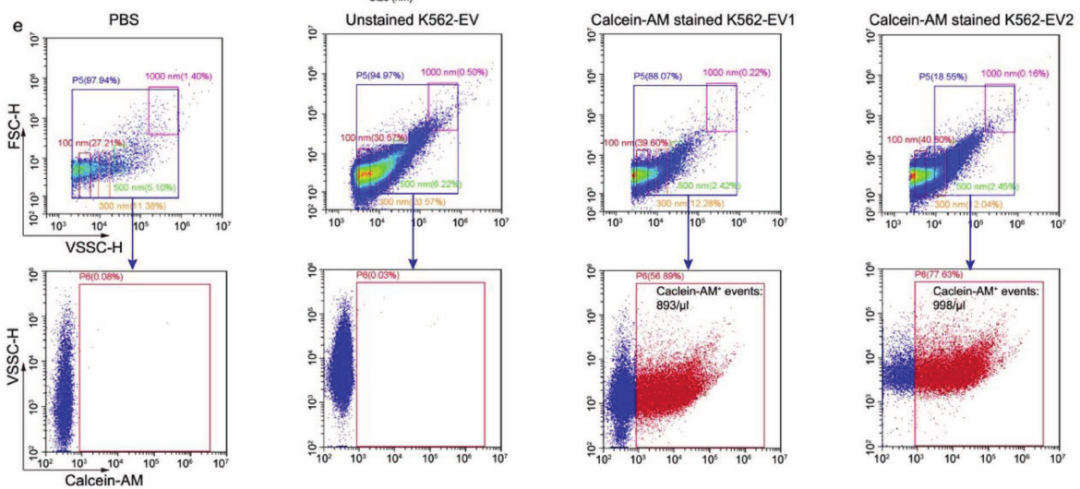
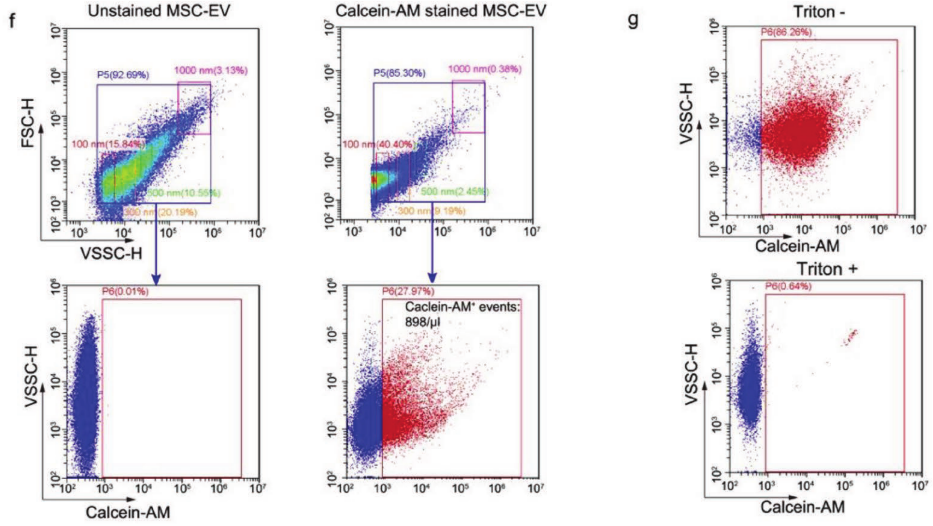
图2 分离的EVS的性质和浓度分析
2、从10x基因组学数据中鉴定单个EV
为了探索单个EV的转录组学特征,在10X基因组学平台上对3个EV样本(K562-EV1、K562-EV2和MSC-EV)进行了scRNA-seq检测。如图3a所示,在去除背景条形码、识别异常值和去除双重值后,使用三个原始的10x基因组输出数据集进行下游分析。虚线下面表示每个数据集在每个步骤中处理的条形码数。由于EV通常比细胞小且含量较低,cell ranger算法无法区分EV和背景(图3b)。经过尝试和改进,使用具有自适应阈值的CB2算法可有效区分真实的EV和背景。在去除背景barcode后,三个样本分别获得了2366、4684和1680个barcode,且拐点清晰(图3c)。然后,设置质控参数去除异常EV,并用DoubletFinder软件去除潜在的doublets。最终,共获得2088个K562-EV1、3935个K562-EV2和1603个MSC-EV的转录组数据。其中,在K562-EV1转录组数据中检测到3299个基因,在K562-EV2中检测到5660个基因,在MSC-EV中检测到2936个基因。K562-EV1和MSC-EV数据集中每个EV的基因中位数非常接近,分别为44.5和42,且UMI计数中位数也相似,而K562-EV2的基因中位数和UMI计数略高些(图3d)。之后,对MSC-EV和MSC的单细胞数据中检测到的基因数的频率分布进行比较,发现EVs的分布形状类似于细胞(图3e)。在K562-EV1中,每个EV检测到的基因数范围为6-97个,在K572-EV2中为11-148个,在MSC-EV中为7-90个,平均值为52。同样地,EV和细胞中UMI计数的分布曲线也显示了类似的结果(图3f)。在K562-EV1中每个EV的UMI计数范围为19-102个,K562-EV2中为33-152个,MSC-EV为19-94个,平均数为66。
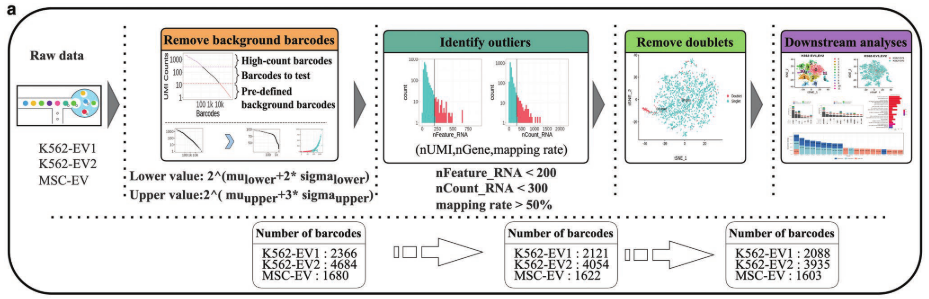
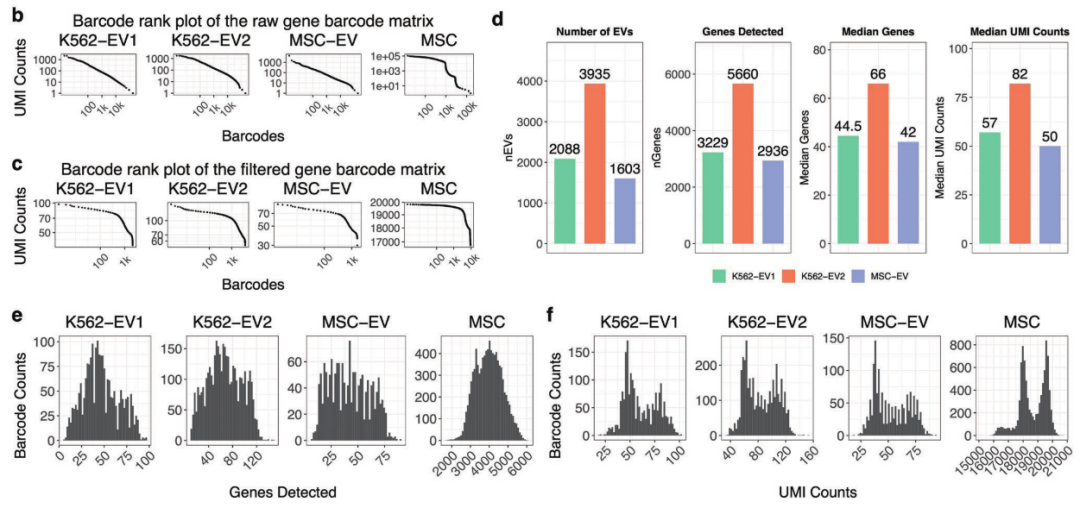
图3 单个EV转录组的特征
3、EV标本中高表达基因的研究
接下来,研究者对3个EV数据集中的前500个高表达基因进行分析。三个EV样本共有323个基因,其中包括78个核糖体基因和8个线粒体基因(图4a)。另外,仅在K562-EV数据集中发现了5个血红蛋白基因和4个线粒体基因。为了验证单个EV数据集的可靠性,将单个EV和批量EV RNA测序数据中表达最高的前500个基因进行比较。发现5个数据集中共有229个基因高表达,包括76个核糖体基因和7个线粒体基因(图4b)。与单个EV结果一致,5个血红蛋白基因也在3个K562-EV数据集中被发现。这说明单个EV测序数据的可靠性。
此外,研究者还分析了每个数据集中基因表达百分比最高的前10个基因(图4c)。HBG2在K562-EV1和K562-EV2中的表达百分比最高,分别为41.9%和49.8%。TMSB10在MSC-EV中表达百分比最高,为35.1%。EEF1A1在3个EV数据集中均为高表达基因,表达百分比分别为38.2%、44.8%和28%。然后,将上述筛选出来的20个基因比较分析,发现其中18个基因是核糖体基因,EEF1A1和FTL(铁蛋白轻链)除外。
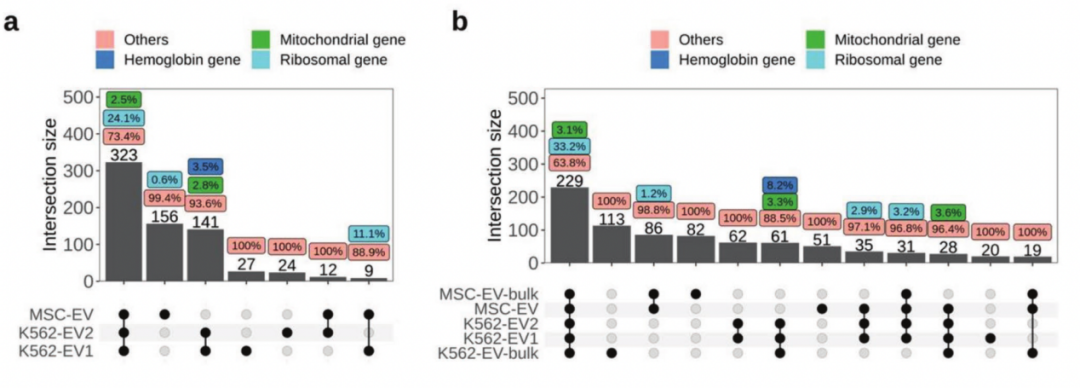
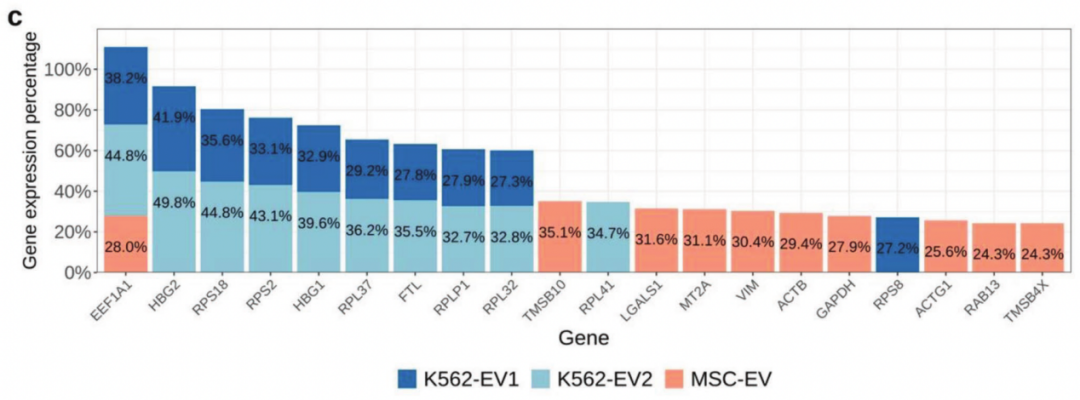
图4 EV样本中的高表达基因
4、单个EV转录异质性
为了探索单个EV的转录异质性,采用t-SNE和无监督聚类分析单个EV测序数据。Harmony包用来整合和校正K562-EV1和K562-EV2数据集上的批次效应,然后确定了12个clusters,而在MSC-EV数据集中确定了10个clusters(图5a-c)。同时,使用Seurat软件包中的FindAllMarkers功能来表征每个EV cluster中的标记基因。在整合的K562-EV数据集中,cluster 0的EV数占比最多,约为28.9%,但缺乏显著的标记基因;cluster 1检测到1个核糖体标记基因,cluster 2有2个核糖体标记基因;cluster 6和cluster 11的标记基因均为线粒体基因,cluster 8的标记基因都是血红蛋白基因(图5d)。而在MSC-EV数据集中,cluster 7的标记基因是线粒体基因(图5e);且与GO富集结果中显著富集的细胞骨架相关功能一致,存在多个细胞骨架基因,如原肌球蛋白基因(TPM1/2/4)、微管蛋白基因(TUBB)等。比较K562-EV和MSC-EV数据集中的cluster标记结果,除线粒体基因和核糖体基因外,还存在2个重叠基因,转移相关肺腺癌转录物1(MALAT1)和热休克蛋白90α家族A类成员1(HSP90AA1)。
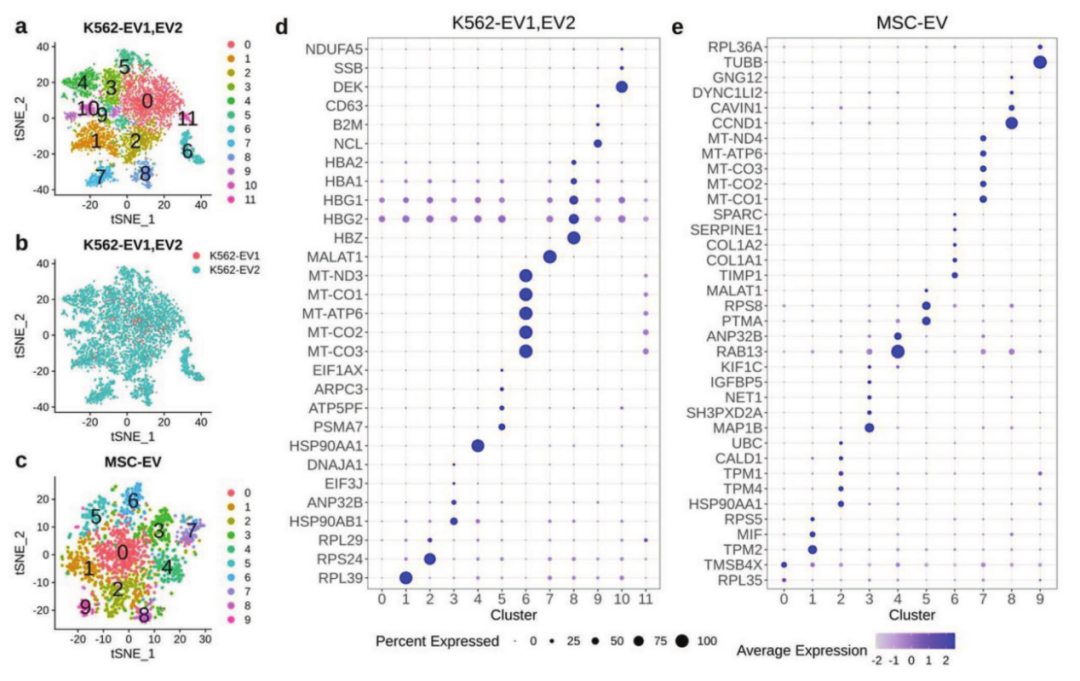
图5 单个EV的转录异质性
5、整合EV与亲本细胞的转录谱
为了比较EV与其亲本细胞的表达,将单个EV数据集与亲代细胞单细胞数据集整合并进行分析。结果表明,每个cluster均存在EV和细胞,说明不同的EV来自不同的细胞。在K562-EV1、K562-EV2和K562这3个数据集中,共确定了9个不同的clusters(图6a)。Cluster 6和cluster 7的数据大多数来自EV数据集,cluster 8的的数据主要来自K562细胞数据集(图6b)。Cluster 1的标记基因主要是血红蛋白基因,而cluster 2的标记基因是线粒体基因(图6e)。此外,整合MSC-EV和MSC数据集确定了12个clusters(图6c),单个EV分散在MSC中(图6c,d)。所有clusters的标记基因以散点图展示,包括许多细胞骨架基因,如TUBA1A/B/C和TUBB4B和肌动蛋白基因(ACTA2/G2)(图6f)。
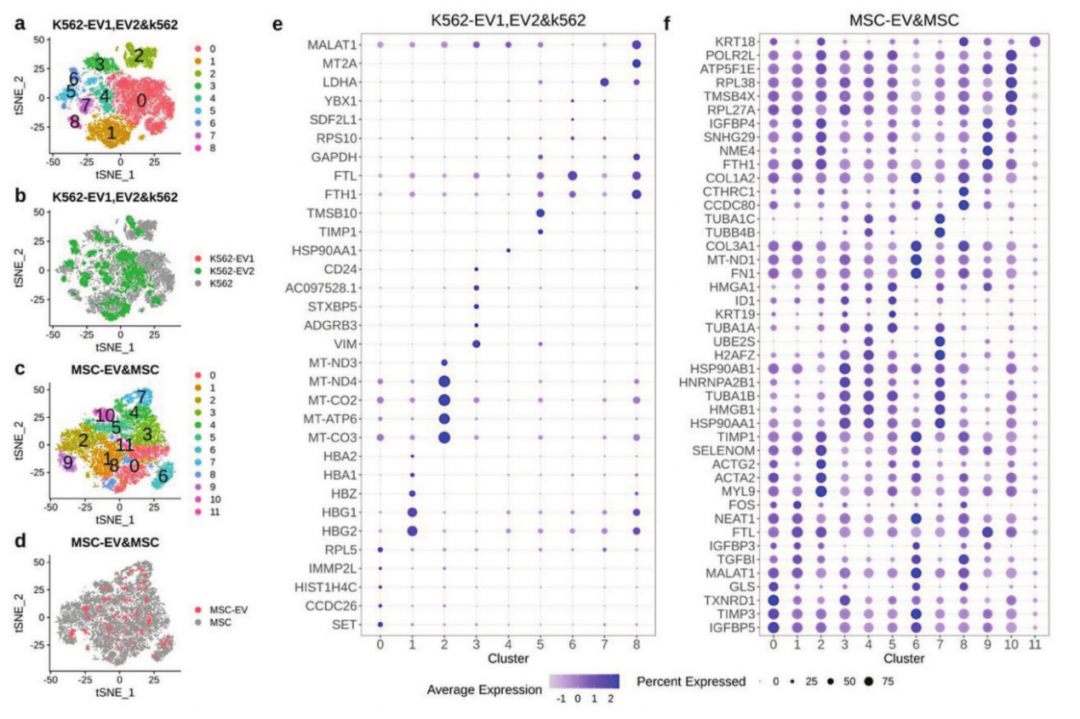
图6 整合EV与亲本细胞的团簇分布
总结
scRNA-seq技术的飞速发展使人们对组织中细胞种类、细胞的特殊状态有了深入认识。而scRNA-seq的优势是它能够检测出细胞之间的差异性,提高了基因表达研究的分辨率, 因此它常被应用于以下几个方面 :探索组织中存在哪些细胞类型或者亚群;识别未知或稀有的细胞类型或状态;阐述在细胞分化过程或者不同时间内基因表达变化;检测特定细胞亚群的差异表达;通过表达谱进行聚类,推断基因调控网络。
基于10x Genomics Chromium系统利用油包水的微反应体系,通过序列标签区别群体中的不同细胞,获得单细胞水平的数字化基因表达谱,可实现数千甚至数万个单细胞群体分析,解决常规scRNA-seq方法在通量或扩展性方面存在的不足,为单细胞研究开拓新的思路。其主要优势:超高通量;项目周期短;细胞捕获效率高;真正的单细胞测序;测序平台兼容度广。
然而,此篇作者首次使用10x Genomics Chromium平台和改进的实验和生物信息学分析方法对单个EV的基因表达进行高通量分析,揭示了K562和MSC的单个EV RNA表达谱和异质性。这种方法适用于所有类型的EV,为EV货物分子及其分类提供了途径。