
2022-12-05
此产品具有测序单碱基准确性高、成本低、测序通量大、DNA起始量要求低、组装效果好等等优点,说到这里你是不是很好奇我们今天的主人公是谁了,那快来和小派一起看看吧!
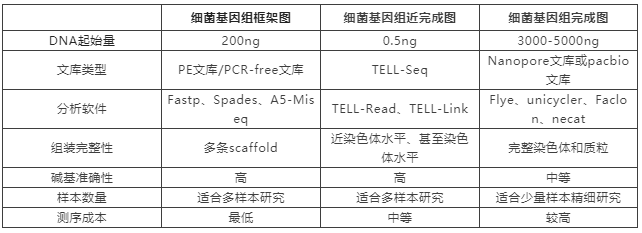
今天介绍的主人公名字叫做细菌基因组近完成图测序,它是细菌基因组de novo测序中的一员,听到这个名字大家是不是觉得熟悉但是又有点陌生呢?因为我们正常听说到的细菌de novo测序主要是细菌基因组框架图测序(或扫描图测序)和细菌基因组完成图测序,那细菌基因组近完成图测序又是什么呢?其实细菌基因组近完成图测序主要是在建库方法上进行了优化,采用常见的二代短读长测序仪器进行测序,进而得到接近染色体甚至染色体水平的高质量基因组。此方法对于DNA起始量要求极低且测序成本较低,是一款适合于大样本量研究且对组装结果有一定要求的老师。
细菌基因组近完成图测序和常规的de novo测序一样都需要经过样本准备、文库构建、高通里测序以及生物信息学分析这些过程,但是细菌基因组近完成图测序最大的区别就是在DNA起始量、建库方式和对测序数据的处理上。

图1:细菌基因组近完成图分析流程
1.DNA起始量要求不同:
近完成图测序DNA起始量要求非常低,单次建库仅需0.5ng,而常规的PE文库、PCR-free文库、三代文库的单次建库量一般在0.2-5ug之间。
表1:三款细菌基因组denovo测序产品比较
2.建库方式不同:
近完成图测序采用的是转座酶关联长读长测序(TELL-Seq)技术,该技术通过转座酶对来自同一条长DNA的小片段进行特定标记,利用标记对短读长序列进行拼接,让二代测序的短读长数据产生超长读长测序效果,等价读长达20kb-200kb。这个主要是由于在文库构建过程中添加了数以百万计的带有独特标签序列的磁珠到样本中与DNA发生反应,这些标签序列对DNA长片段进行编码标记,经过测序后,便可获得长片段DNA信息,从而精确地组装基因组。
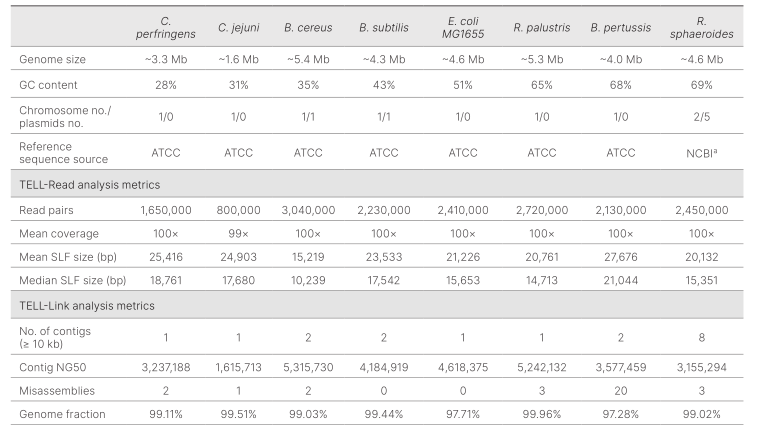
图2:TELL-Seq建库原理
3.分析软件不同:
TELL-Seq文库进行生物信息学分析的时候使用的是其配套的Tell-Read、Tell-Link软件分别进行数据质控、基因组组装,并对组装的基因组进行校验,进而得到一个高质量的细菌基因组。
4.组装得到的基因组效果不同:
细菌基因组de novo测序的多款产品的组装效果是有明显差异:(1)细菌基因组框架图测序得到的是多条scaffold,基因组相对来说比较碎;(2)细菌基因组完成图测序得到的是完整的染色体和质粒信息;(3)细菌基因组近完成图测序得到的是接近染色体甚至染色体水平的基因组。
表2:TELL-Seq技术组装得到的基因组信息
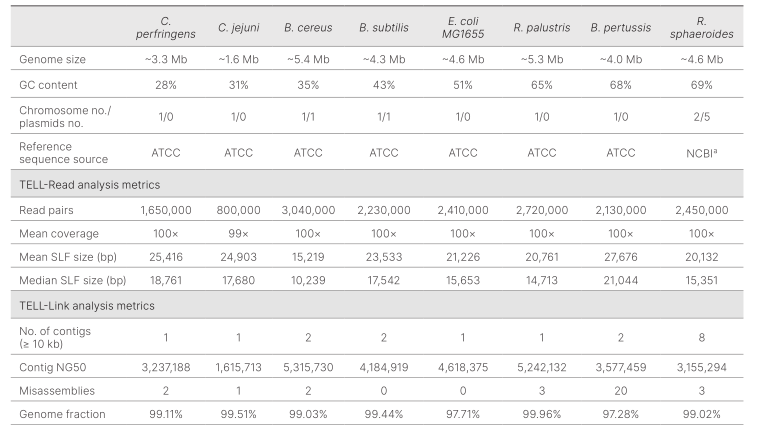
如表格中测试组装得到的基因组:此次测序使用的基因组大小在1.6Mb-5.4Mb之间,GC含量在28%-69%之间,包含了革兰氏阴性菌和革兰氏阳性菌。根据组装得到的N50、contig数量、错误装配的数量和基因组测序深度来看,基因组组装质量受基因组大小或GC含量的影响较小,此次测序的基因组几乎均组装成接近完整染色体或质粒的基因组。
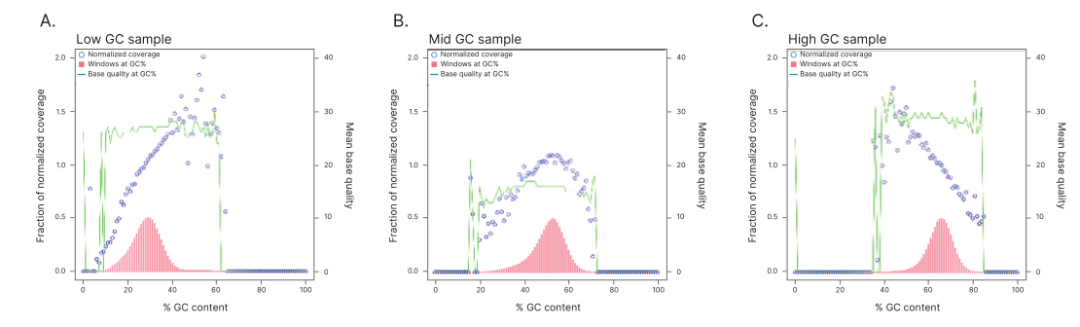
图3 不同GC含量的基因组测序深度比较
5.测序成本不同:
单次检测建库成本由高到低为:细菌基因组完成图测序>细菌基因组近完成图测序>细菌基因组框架图测序,所以如果您是希望花费较少的钱得到完整性更好的基因组的话,那选择细菌基因组近完成图测序准没错!
综上所述,细菌基因组近完成图测序是一个采用TELL-Seq关联长读长二代测序技术,结合二代高通量测序平台,打造出一款通过二代测序平台达到接近三代组装效果的全新产品。如果您也对此产品感兴趣,欢迎发邮件或者致电我们哟(邮箱地址:microsupport@personalbio.cn,联系电话:025-56165883-832)!
参考文献:
[1]Chen Z, Pham L, Wu TC, Mo G, Xia Y, Chang PL, Porter D, Phan T, Che H, Tran H, Bansal V,
Shaffer J, Belda-Ferre P, Humphrey G, Knight R, Pevzner P, Pham S, Wang Y, Lei M. Ultralow-input single-tube linked-read library method enables short-read second-generation sequencing systems to routinely generate highly accurate and economical long-range sequencing information. Genome Res. 2020 Jun;30(6):898-909. doi: 10.1101/gr.260380.119. Epub 2020 Jun 15. Erratum in: Genome Res. 2021 May;31(5):934. PMID: 32540955; PMCID: PMC7370886.
[2]Chiu, R., Rajan-Babu, IS., Birol, I. et al. Linked-read sequencing for detecting short tandem repeat expansions. Sci Rep 12, 9352 (2022). https://doi.org/10.1038/s41598-022-13024-4.
[3]Arun Sethuraman, Rosalina Stancheva, Ciara Sanders, Lakme Caceres, David Castro, Hannah Hausknecht-Buss, Simone Henry, Haven Johansen, Antolette Kasler, Sandy Lastor, Isabelle Massaro, Immanuel Mekuria, Andrea Moron-Solano, Niki Read, Gretchen Vengerova, Andrew Zhang, Xiaoyu Zhang, Betsy Read, Genome of a novel Sediminibacterium discovered in association with two species of freshwater cyanobacteria from streams in Southern California, G3 Genes|Genomes|Genetics, Volume 12, Issue 7, July 2022, jkac123, https://doi.org/10.1093/g3journal/jkac123