2023-10-24
上次(如何使用cytoscape玩转网络图?(一)点击查看)带大家了解如何用cytoscape绘制PPI网络图、以及使用MCODE插件来筛选核心网络,今天小派再给大家来详细介绍网络调整的参数和其它两个好用的小插件,助大家都能够玩转cytoscape。
Cytoscape和Java软件可通过官网下载,或下方网盘获取。 链接:https://pan.baidu.com/s/1HqAbflFfRZHveuRgJZ3rGA?pwd=lv05 提取码:lv05
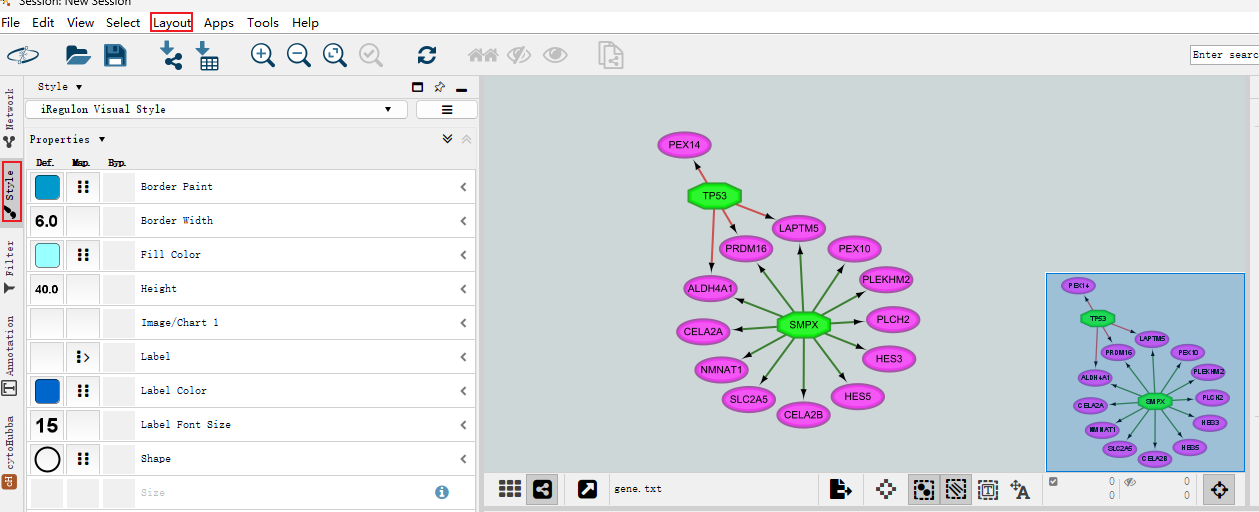
四、Style调整参数 首先可以在Style里选择一些预设的样式,如:Smple1样式。 然后可以对Node(节点)进行调整: Border Paint/Width:边框的颜色和宽度; Fill Color:节点填充的颜色; Width/Height:节点长宽; Image/Chart 1:自定义的节点图形(图像、图标或渐变); Label:节点标签; Label Color:节点标签颜色; Label Font Size:节点标签字体大小; Shape:节点形状; Size:节点大小; Transparency:节点填充颜色的透明度(0表示完全透明,255表示完全不透明)。 此外Properties里还有一些其它可以调整的选项,常见的有: Label Font Face:节点标签字体; Label Position:节点标签相对于边的位置; Label Transparency:节点标签的透明度(0表示完全透明,255表示完全不透明)。 还可以对Edge(边)进行调整: Color:边的颜色; Label:边的标签(其它的参数和Node里的标签类似); Line Type:边线条类型(实线,虚线等); Source/Target Arrow Shape/Unselected Paint:箭头/箭尾的形状/颜色; Stroke Color:箭头后面连线的颜色; Transparency:边填充颜色的透明度(0表示完全透明,255表示完全不透明); Width:边线条宽度。 备注:这些调整可以通过分析网络和attributes里的各种属性进行自定义调整,例如节点的颜色按照上下调区分、边的粗细按照score值渐变、节点大小按照Degree渐变等。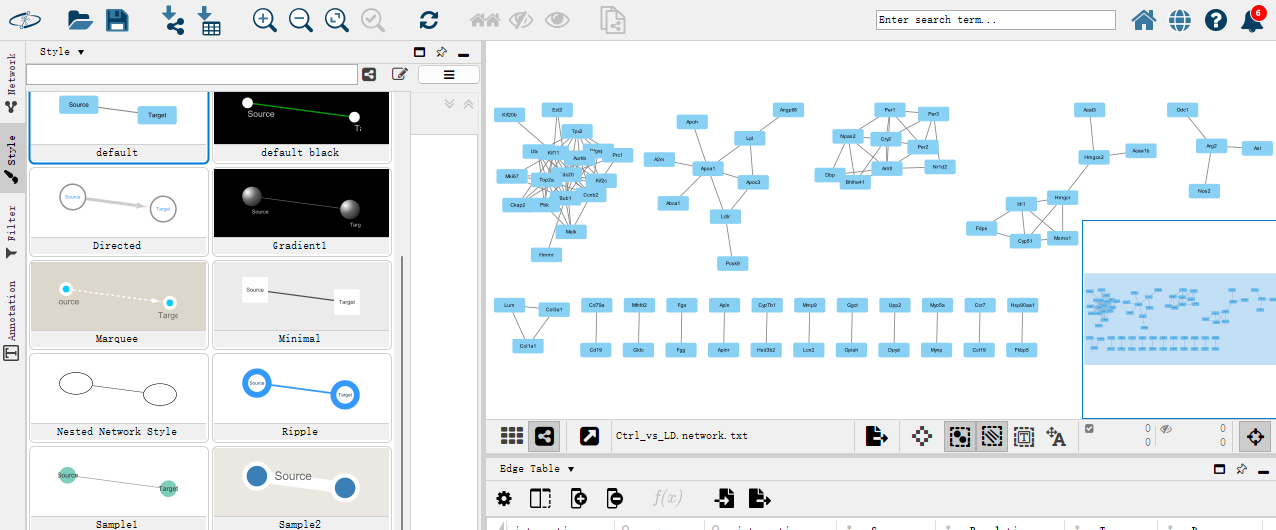
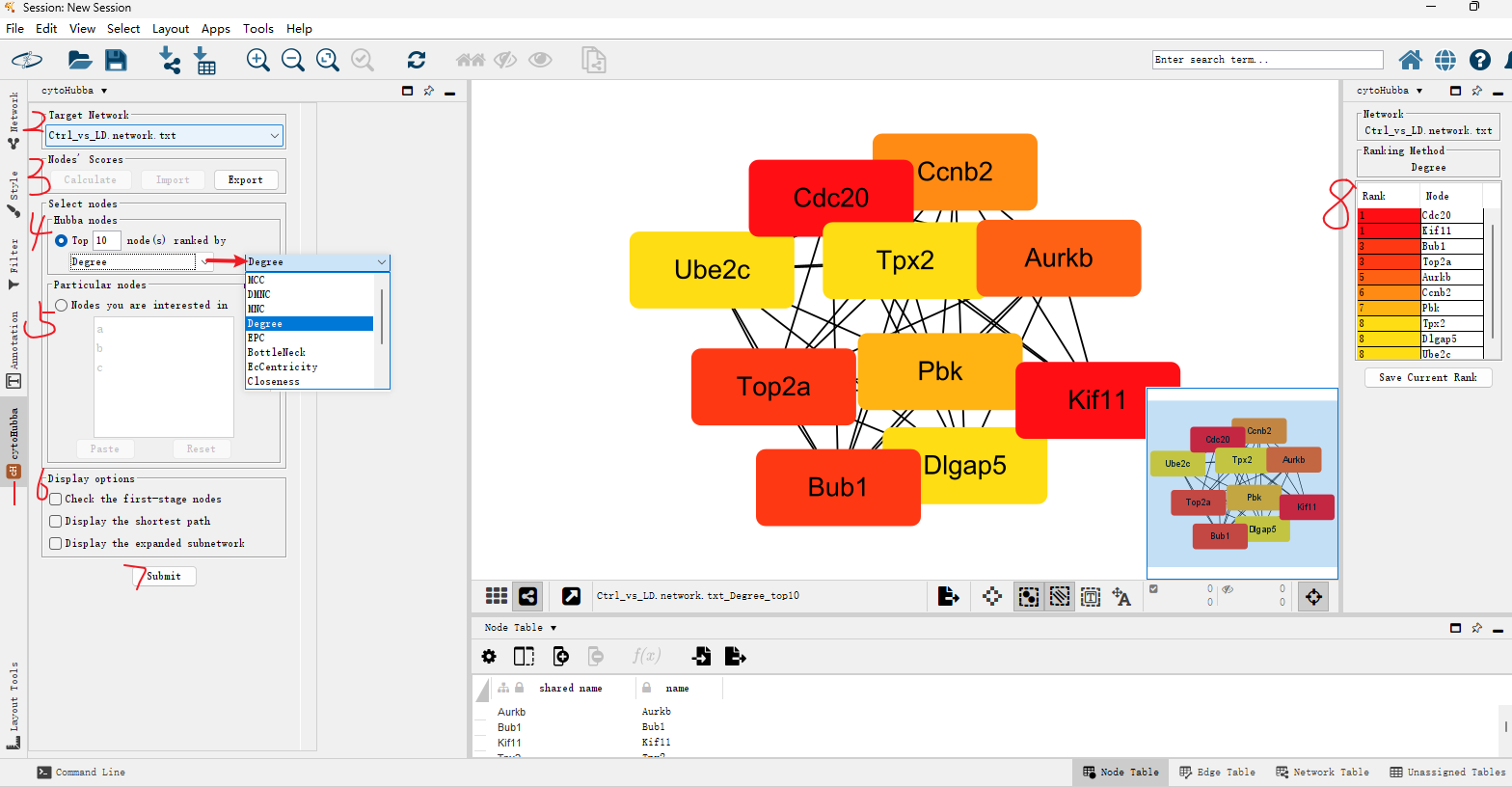
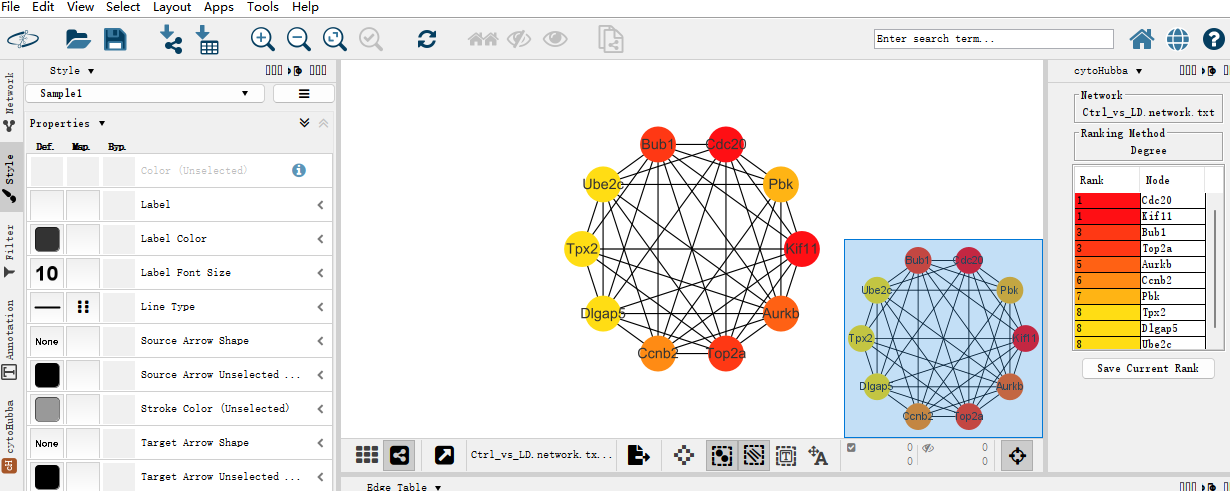
五、筛选hub基因--cytoHubba插件 当构建的PPI网络存在上百个基因的对应关系时,可以利用cytoHubba插件通过拓扑网络算法给每个基因赋值,排序发现其核心基因(hub gene)和子网络。 首先在Apps里的App Manager...里搜索并安装cytoHubba插件。 操作步骤: 1、选择左边的cytoHubba进入工作页面; 2、Target Network里选择需要分析的网络; 3、点击Calculate; 4、点击Top 10,可根据需求调整提取关键基因的数量;另外点击MCC可选择算法,它提供了11种拓扑分析方法,包括:MCC、Degree等; 5、Particular nodes为选择感兴趣的基因进行分析(一般是对整个网络分析的话,此项可以忽略); 6、点击左下方Display the shortest path:显示最短的路径,(Check the first-stage nodes:显示出hub gene连接到的第一个节点、Display the expanded subnetwork:显示出最长的子网络); 7、点击Submit提交; 8、提交后就是按照Degree筛选的Top10核心基因和对应的网络,右边的列表就显示核心基因的排名(颜色越深,证明分数越高,越显著); 9、最后可以回到Style界面对网络进行调整和导出。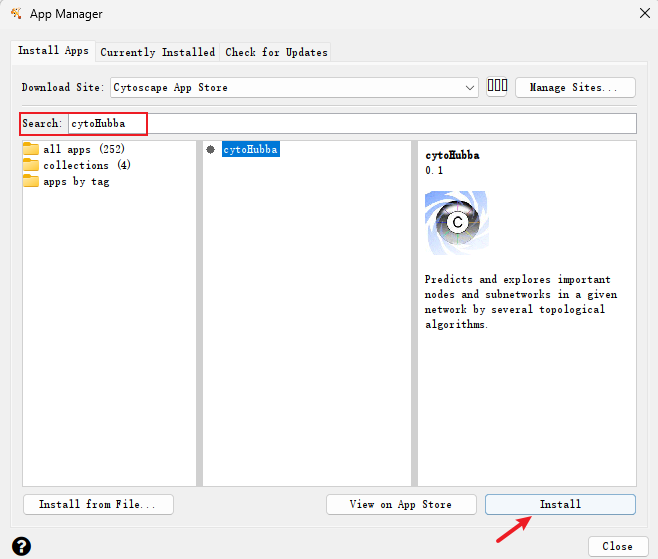
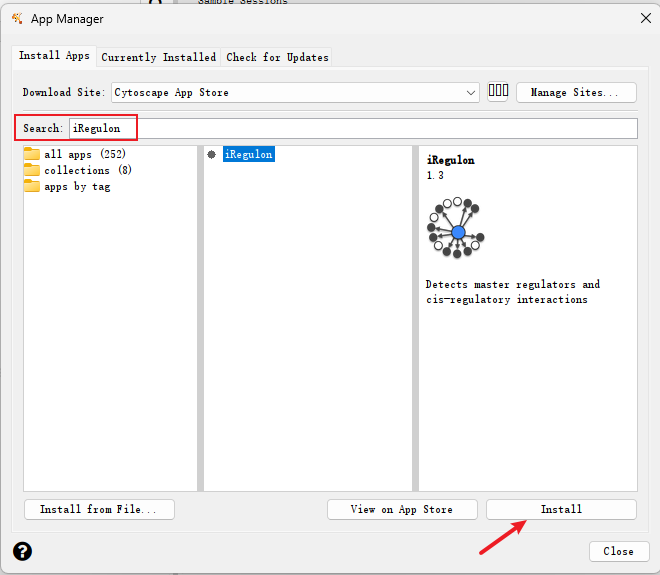
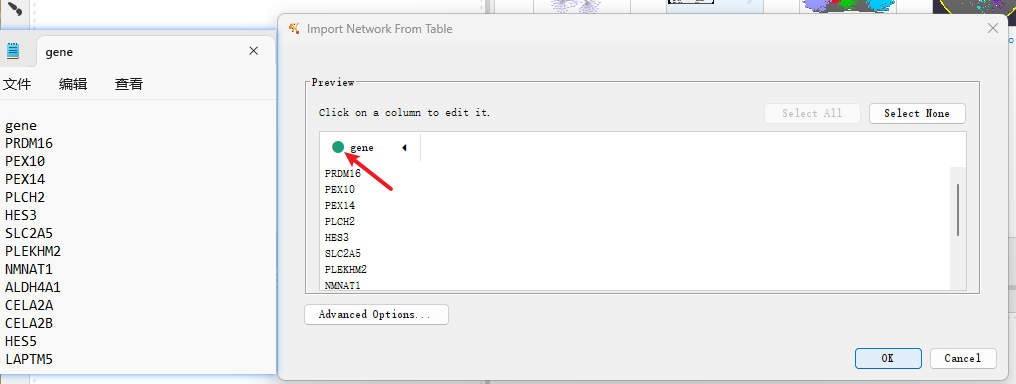
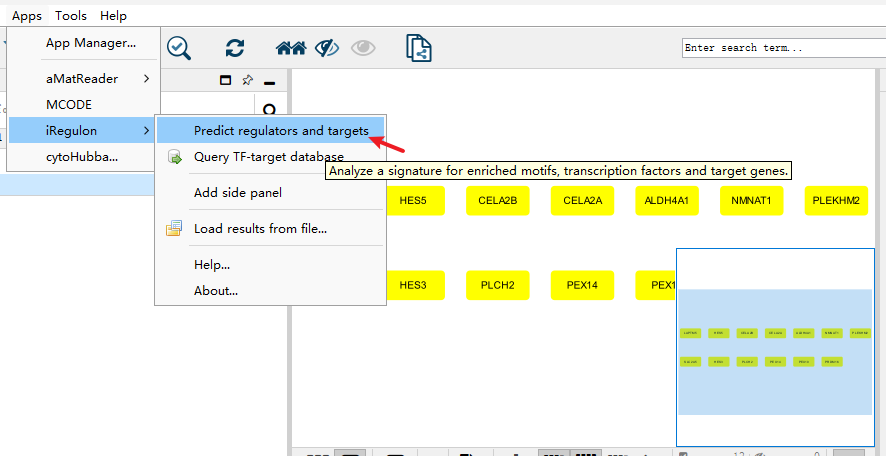
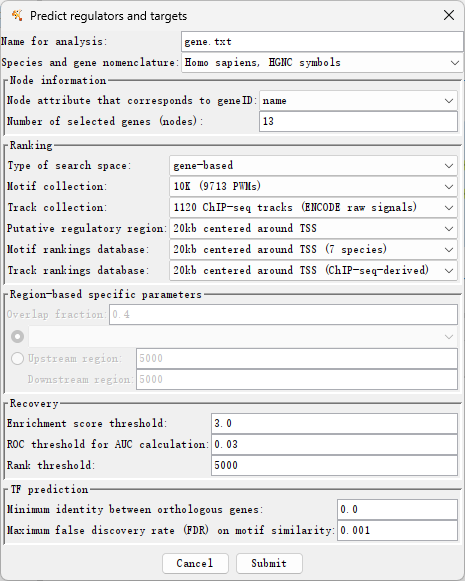
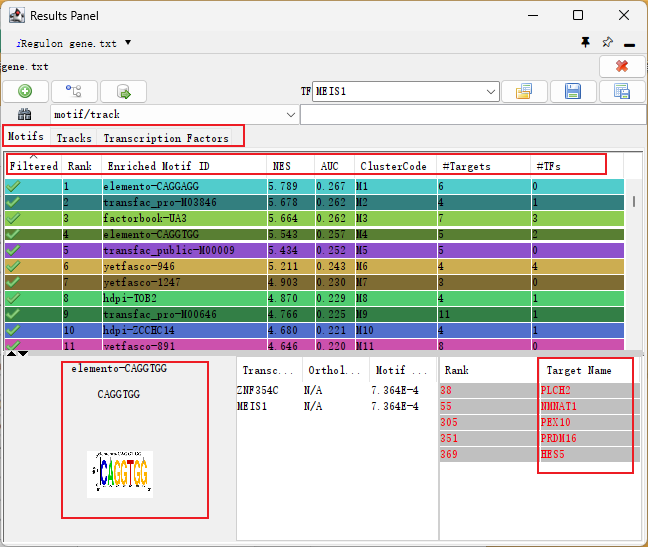
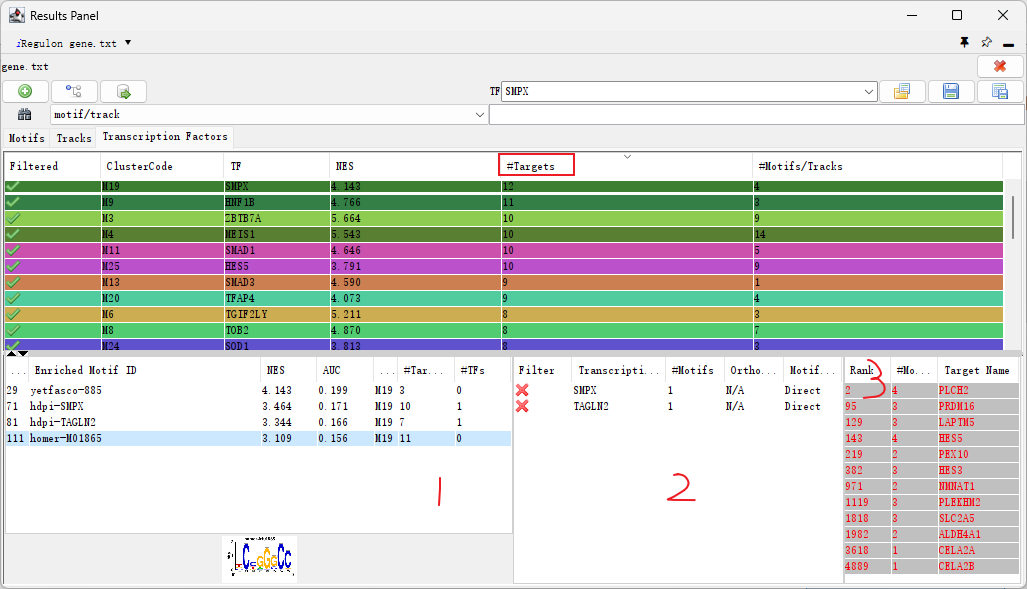
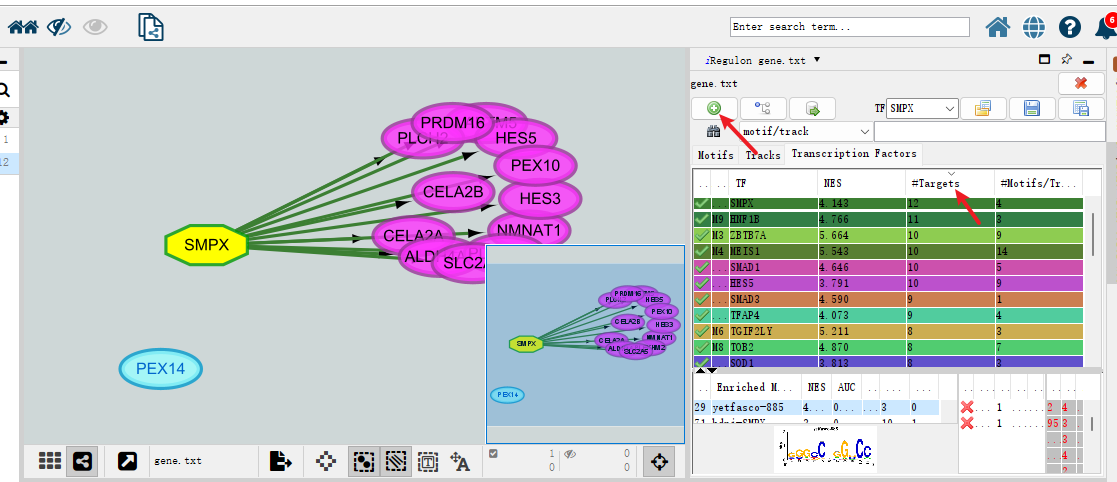
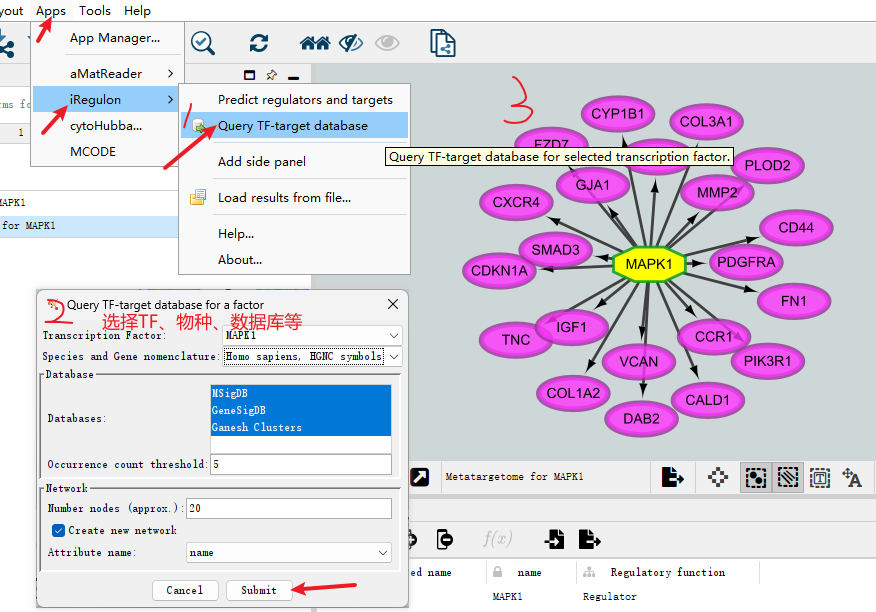
六、iRegulon插件 iRegulon作为一个Cytoscape插件,可以识别目标基因的重要调控因子,支持人类、小鼠和果蝇基因。该插件主要使用近10000个TF motifs数据库和1000个ChIP-seq数据集或“tracks”来检测富集的TF motifs或ChIPseq峰。接下来,它将富集的TF motifs和“tracks”与靶点基因联系起来。 首先在Apps里的App Manager...里搜索并安装iRegulon插件。 首先需要准备用于分析的靶基因列表,并导入Cytoscape(选为source node即可)。 选中需要分析的基因(黄色为已选中),然后打开Apps→iRegulon→Predict regulators and targets。 打开后出现以下参数窗口: Name for analysis:分析命名; Species and gene nomenclature:物种和基因命名法(人、小鼠和果蝇); Node information: Number of selected genes:所预测的基因数目; Ranking: Type of search space:基于基因; Motif collection:10k / 6k; Track collection:1120 ChIP-seq/ 750; Putative regulatory region:起始位点上下游端; Motif / Track rankings database:排序所依据的数据库; Recovery: Enrichment score threshold:富集分数; ROC threshold for AUC calculation:AUC值; Rank threshold:排序阈值; TF prediction: Maximim false discovery rate (FDR) on motif similarity:发现错误率。 插件需要调整的参数较多,一般都是在原理涉及到的数据库,若需要详细了解可查看对应插件文献。一般使用默认参数即可。 结果主要分三个部分:Motifs,Tracks,Transcription Factors。 Motifs: Enriched Motif ID:特征序列ID; NES:根据PWM库富集出来计算得分,类似匹配程度; AUC:基因里包含多少比例的共表达基因来表示; ClusterCode:聚类(根据相似的Motifs); #Target:目标基因数目; #TF:转录因子数目; 左边:logo展示图:用于描述序列特征(字母的相对大小表示它们在序列中的频率,每个字母的高度与该位置的相应碱基的出现频率成正比,常以bits为单位); 中间: Transcription Factor:转录因子; orthologous identification:同源性; Motif Simialrity : Motif 间相似性; 右边:靶基因的名称。 Motif和Tracks应该是两个类似的求TF的数据库,出来的结果列名也类似,一般选择Motifs的结果。 选择Transcription Factors后,列表主要以TF呈现结果。可以按照靶基因排序,看和目标基因里靶向最多的TF有哪些。例如选中第一个SMPX转录因子后,1里显示这个TF对应的4个Motifs,2里显示Motif对应的TF,3里该TF对应的12个靶基因。 绘制网络图时,可以选择感兴趣的或对应靶基因最多的TF,直接选中再点击上方+,就可出现对应网络。 添加完感兴趣的TF后,可以通过Layout和Style对网络进行调整后导出保存。 此外,iRegulon插件还可以通过TF预测靶基因。 本期cytoscape的使用就介绍到这里,更多cytoscape的实用方法分享,敬请期待!