2023-11-06
(一)De novo基本概念(是什么)
全基因组De nove测序即基因组从头测序,是指不依赖于已知的基因组序列信息,对某个物种的基因组进行测序,然后应用生物信息学技术对测序数据进行拼接和组装,最终获得该物种基因组序列图谱,然后对组装出的基因组序列进行结构预测、功能基因注释,得到该物种的物理图谱信息。
(二)De novo的应用方向(什么时候做) (1)一个物种未有过参考基因组,需要组装出该物种的基因组。 (2)一个物种有参考基因组,但不是很完整,需要更高级别的基因组。 (3)一个物种单一参考基因组一不能涵盖本物种的所有遗传信息,想要组装一个泛基因组,包含更多的遗传多样性。
(三)De novo测序发展历程 基因组De novo历程与DNA-seq的发展相伴随。1977年,第一代桑格Sanger测序技术发展至今,已经足有40年时间。测序读长从长到短,再从短到长。目前第二代短读长测序技术在全球范围内上占有着绝对的垄断位置,近几年第三代测序技术也得到了飞速发展。测序技术的每一次变革和突破,都对基因组学研究、疾病医疗研究、药物研发、育种等领域产生巨大的推动作用。基因组Denovo也经历了四个主要阶段:
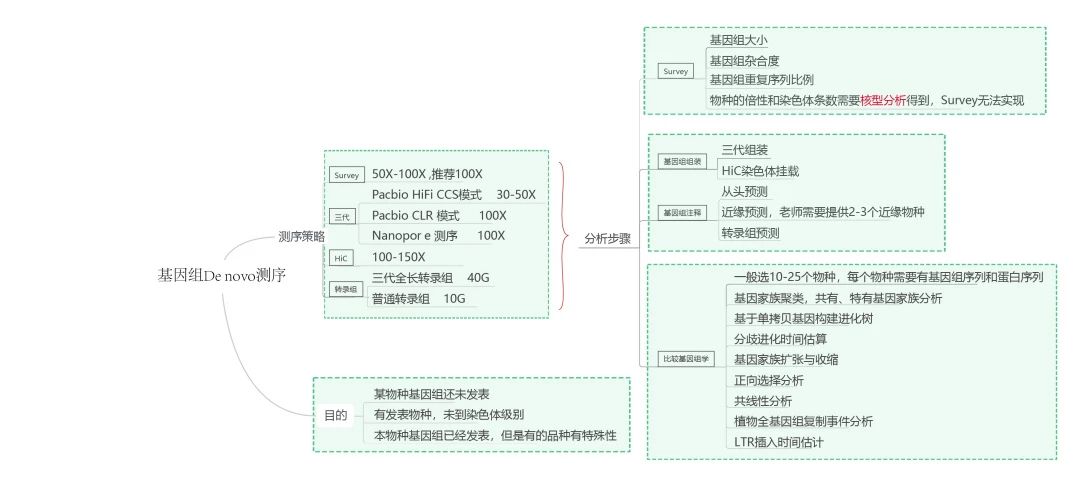
(四)De novo不同测序平台(如何做) 二代测序:基于二代测序平台数据,进行基因组的Survey,得到基因组的大小、杂合度、重复序列情况,了解基因组复杂度情况,一般建议测序数据量100X。 三代测序:基于三代测序平台数据,进行基因组组装,得到基因组的组装碎片段(contig)。三代目前有多种平台,具体推荐如下:Pacbio HiFi数据,建议30X~50X(推荐);Pacbio CLR数据,建议100X;Nanopore数据,建议100X(不推荐)。 三代的优势是读长长,举个例子:基因组组装类似拼图,测序读长就类似拼块,我们知道拼图时某个拼块越大(读长长),越好完成拼图(基因组),是不是就很好理解三代长读长对于基因组组装的优势啦~ HiC测序:基于染色质三维空间结构的一种测序方法,将三代测序数据组装的基因组碎片段(contig)挂载到染色体上,实现chr级别的基因组组装。一般建议测序数据量100X。 ONT ultra-long(超长ONT):读长更长,用于补基因组gap,实现若干条染色体达到端粒-端粒的0gap级别的基因组。相继有西瓜、拟南芥、水稻、玉米等物种的T2T基因组被构建。预示着T2T基因组(4.0级别)的时代即将来临。一般建议测序数据量50X。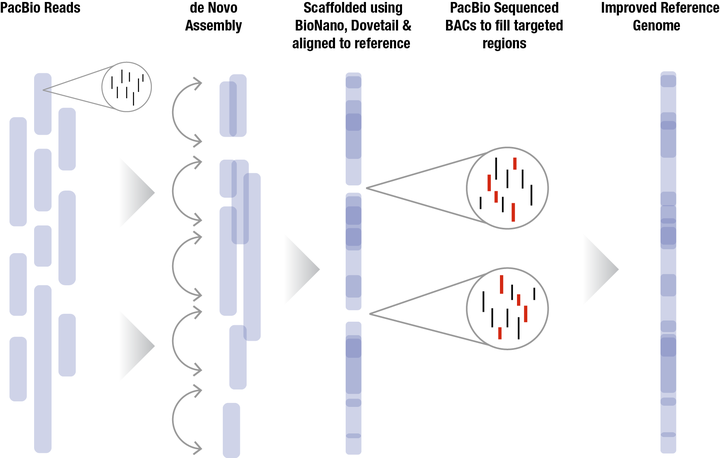
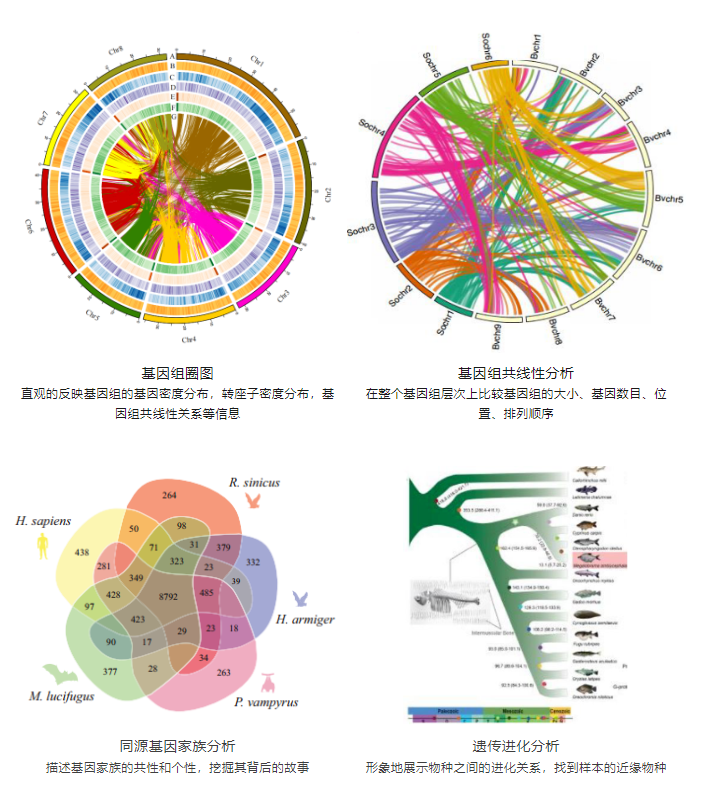
(五)Denovo常见结果展示
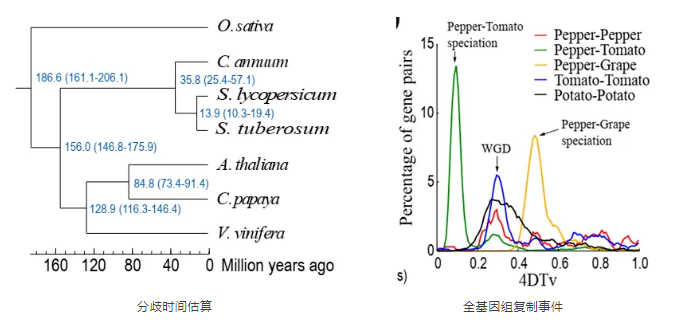
(六)Denovo案例解读
【派森诺项目文章】染色体水平的基因组组装揭示了雉鸡的免疫基因组织和进化特征 一、研究内容 采用二代测序、三代Pabio测序和Hi-C辅助基因组组装技术,对雉鸡进行了de novo测序、组装和注释,获得了染色体级别的高质量雉鸡参考基因组。 二、研究结果 1、核型和系统发育分析表明,雉鸡是火鸡的姊妹种,在24.7~34.5 Ma之间出现分化; 2、基因组中发现在一系列钙相关基因中发生了拷贝数改变和特异性正选择,在雉鸡的适应性进化中起着重要作用; 3、组装了雉鸡完整的MHC-B区域,从而扩展了MHC的光谱,为进一步研究禽类MHC基因的进化和家禽免疫提供了有价值的资源; 4、对国内雉鸡品种进行了基因组重测序群体遗传学分析,找到了与家养雉鸡体重相关的人工选择区域,揭示了生产性状的人工选择信号。
基因组学的研究意义深远,它不仅推动了基础科学的进展,揭示了生物的演化和遗传机制,还为人类健康、农业发展、生态保护和科技创新提供了新的思路和工具。随着技术的不断飞跃和数据的积累,基因组学的研究将在更多邻域发挥重要的作用,推动生物科学和应用的不断发展。 上海派森诺生物科技股份有限公司一直致力于动植物基因组学的研究。如需进一步讨论,欢迎发邮件或者致电我们哟(邮箱地址:genome_support@personalbio.cn,联系电话:021-80118168-6611)!
(七)派森诺高分项目文章
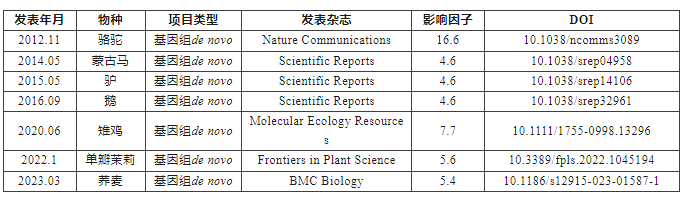
参考文献 He C, Zhao LL, Xiao L, et al. Chromosome level assembly reveals a unique immune gene organization and signatures of evolution in the common pheasant[J].Mol Ecol Resour. 2021 Apr;21(3):897-911.doi: 10.1111/1755-0998.13296


