
2024-01-02
好消息!
Greengenes2数据库现已加入派森诺微生物多样性的流程中!!!
在高通量测序中,使用16S rRNA扩增子方法和宏基因组学方法的研究通常会产生不同的结果,这通常被归因于PCR扩增偏差,目前还没有很好地方法解决这一问题。 2023年7月,Nature Biotechnology(IF: 46.9)发表了Daniel McDonald(通讯作者Rob Knight)等人的文章,引入了Greengenes2。这是一个参考树,通过将序列插入到全基因组系统发育树中,将基因组和16S rRNA数据库统一在一个一致的、集成的资源中。结果表明从相同的样本中生成的16S rRNA和宏基因组数据在主坐标空间、分类学和表型效应大小上是一致的。此外,Greengenes2数据库的系统发育覆盖率远大于SILVA、Greengenes和GTDB等数据库。Greengenes2数据库将会是微生物组研究的一个利器。
鸟枪法宏基因组测序和16S rRNA基因扩增子测序研究在微生物组研究中被广泛使用,但使用这些方法的研究者通常发现它们的结果差异较大。这种跨方法的标准化不足限制了微生物组用于可重复性生物标志物的发现。造成这样结果的一个关键原因是二者依赖于不同的分类和系统发育。例如,Web of Life ( WoL ) 和Genome Taxonomy Database ( GTDB ) 提供的全基因组树只覆盖了一小部分已知的细菌和古细菌,而SILVA和Greengenes则较为全面,但通常无法链接到基因组。 本研究作者利用迭代的方法产生一个单一的大规模参考树,统一这些不同的数据层(基因组和16S rRNA),即为Greengenes2。数据来源及主要处理流程如下: 1、首先从NCBI中均匀采样了15,953个细菌和古细菌基因组的全基因组,并使用新的工作流程 uDance总结 380 个全局标记基因的进化轨迹,重建了准确的系统发育树。这项工作即WoL版本2 ( WoL2 ),是对之前发布的WoL1(10,575个基因组)的重要升级。 2、进一步添加了来自Living Tree Project(LTP)2022 年1月发布的18,356个全长16S rRNA序列、来自Karst等人和地球微生物组计划500(EMP500)的1,725,274个接近完整的16S rRNA基因以及所有全长使用uDance v1.1.0将GTDB r207的 16S rRNA序列定位到基于基因组的主干,生成明确表示的 16S rRNA的基因组支持的系统发育。 3、使用支持深度学习的系统发育放置插入了来自Qiita的23,113,447个短V4 16S rRNA Deblur v1.1.0扩增子序列变体(ASV)(检索于2021年12月14日)以及来自SILVA v138的线粒体和叶绿体16S rRNA。这一步代表来自Qiita超过300,000个公共和私人样本的ASV,包括整个环境微生物组计划EMP和美国肠道计划(American Gut Project/Microsetta,图 1a)。 4、使用uDance确保基于基因组的关系保持固定,并推断全长16S rRNA序列之间的关系。对于短片段,保持基因组和全长关系固定,并彼此独立地插入片段。经过对片段放置进行重复删除和质量控制后,生成了一棵涵盖来自31个不同地球微生物组项目本体论(EMP Ontology 3,EMPO3)环境的21,074,442个序列的树,其中46.5%的物种级叶子被完整的基因组覆盖。使用tax2tree v1.1将分类标签修饰到系统发育上。物种注释使用GTDB r207,并结合2022年1月发布的LTP。分类学以GTDB为优先顺序进行协调,包括保留GTDB的多系标签等。物种注释将使用最新版本的GTDB和LTP每6个月更新一次。 图1 | Greengenes2数据库覆盖信息以及使用Greengenes2实现16S扩增子与宏基因组结果一致性。 a:Greengenes2系统发育,ASV多分支折叠;树枝末端颜色表示在美国肠道计划(AGP)、地球微生物组计划(EMP),两者都有(Both)或两者都没有(Neither)。外圈颜色代表Top 20门; b:进化树同a,使用是否存在于SILVA 138的最佳BLAST比对结果进行着色。外圈颜色与树枝末端颜色相同。 c:EMP样本和通过ASV片段放置添加到树中的新分支长度的量(通过总主干分支长度归一化)。 d:配对的16S V4 rRNA ASV和全基因组鸟枪样本的Bray–Curtis PCoA展示(数据来自The Microsetta Initiative的THDMI子集); e:数据同d,但用属水平数据计算Bray-Curtis; f:数据同d、e,但在ASV和基因组标识符水平上计算加权UniFrac。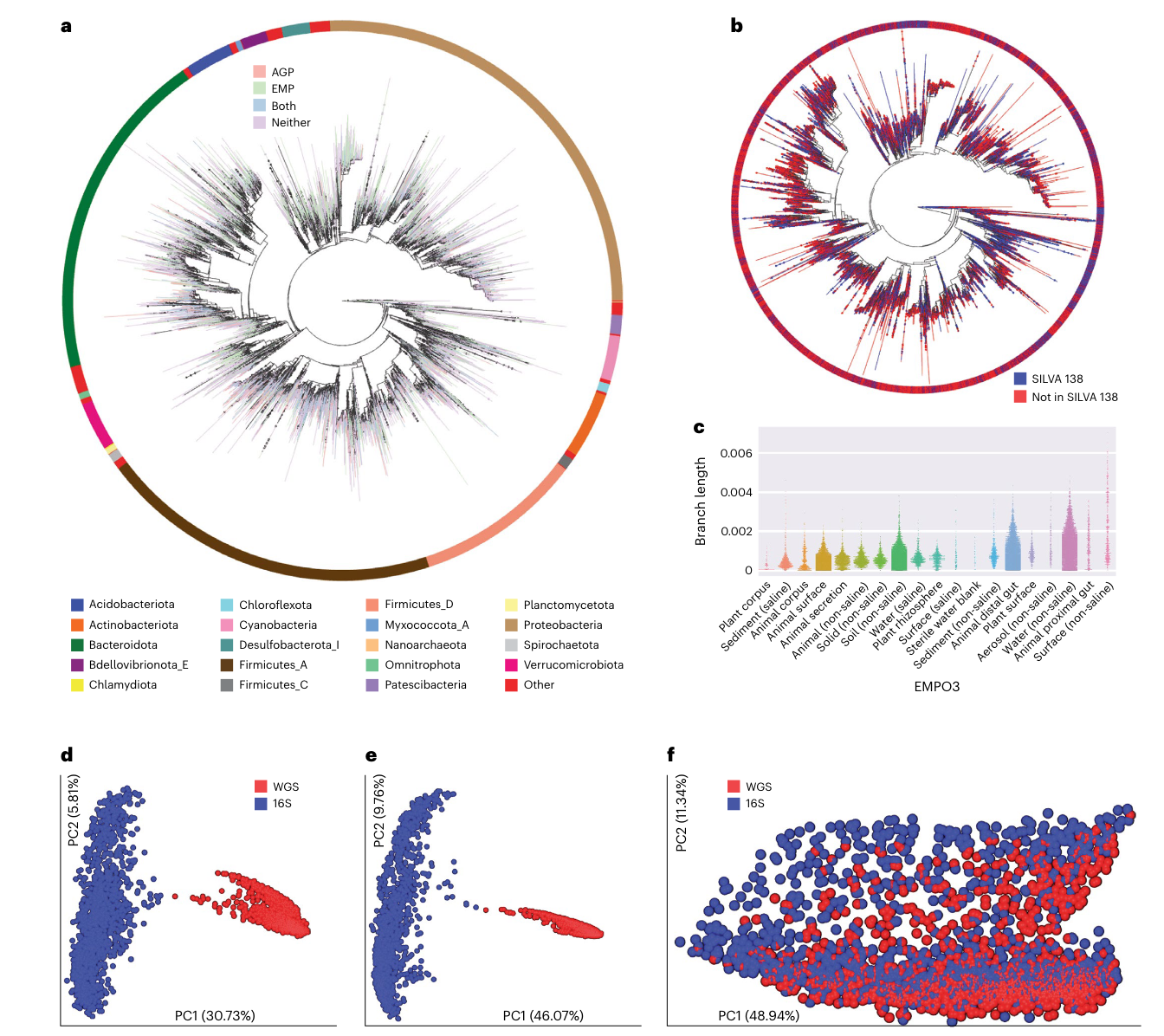
Greengenes2的系统发育覆盖率远大于SILVA、Greengenes和GTDB等。过去比对16S和宏基因组数据集的各种努力仍然导致分布不重叠,并且只有普氏分析(Procrustes)等技术才能显示结果之间的关系。在两个大型人类粪便队列中,16S和宏基因组数据都是在同一样本上生成的,Bray-Curtis(非系统发育)排序在特征水平上无法一致(图1d),并且在特征属水平合并仍表现不佳(图1e)。而UniFrac(一种系统发育方法)与Greengenes2树一起使用提供了更好的一致性(图1f)。对非人类环境的适用性,在16S和来自EMP的宏基因组数据的特征级别上计算了Bray-Curtis和加权UniFrac。本研究也测试了环境类型样本,与人源数据一样,Greengenes2系统发育使用后结果具有更好的一致性。 作者发现,每个样本的宏基因组和16S分类相对丰度分布甚至与物种水平一致。首先使用Woltka流程计算了宏基因组数据的分类配置文件。使用来自q2-feature-classifier(v2022.2)的朴素贝叶斯分类器将每个级别的GTDB r207分类结果与SILVA v138(图2a)或下至物种水平与Greengenes v13_8(图2b)进行比较(无法进行物种水平的一致性比较)。相比之下,Greengenes2在属水平提供了极好的一致性(Pearson r = 0.85),在物种水平上提供了良好的一致性(Pearson r = 0.65)(图2c)。有趣的是,这棵树现在已经足够完整,以至于精确匹配16S ASV然后从树上读取物种注释甚至比朴素贝叶斯分类器表现得更好(属水平 r = 0.54和物种水平 r = 0.84)。 总之,这些结果表明,使用一致的、综合的分类资源显著提高了使用不同数据类型的微生物组研究的可重复性。 图2 | 16S rRNA ASVs和宏基因组数据之间的分类学和效应大小一致性。 a-c:饮食微生物组计划的16S和全基因组宏基因组图谱之间的样本分类学比较。实线表示中位数,虚线表示第25和第75个百分位数。 d-e:用Evident对来自饮食微生物组计划的成对16S和全基因组鸟枪样本进行的效应大小计算。使用16S的ASV和鸟枪样本的基因组标识符以最大分辨率进行计算。这里显示的数据是人类肠道微生物组样本。星号表示特定的变量。
参考文献 McDonald D, Jiang Y, Balaban M, et al. Greengenes2 unifies microbial data in a single reference tree[J]. Nature Biotechnology, 2023.