
2024-01-08
组学测序的脚步已迈入单细胞时代,在科学探究的过程中,众多科研人员正在准备或已经将单细胞测序技术纳入自己的科研方法和项目亮点当中,对于单细胞转录组数据的处理,可能会遇到大大小小的疑问。本期就针对数据处理过程中的常见疑问、出现原因及解决方案做一个汇总,希望能对各位老师在数据挖掘上提供些帮助,同时也祝愿各位老师新的一年里基金申请顺利,成果产出丰硕,科研再上巅峰!
1、单细胞项目中,最终每个样本的细胞捕获数目是怎么判定的?
答:
10x单细胞平台的初步细胞数目判定是由Cellranger软件完成的。Cellranger是10x公司发布的针对10x单细胞数据进行质控分析的软件,它对于细胞数的判定是根据一定的算法规则来的。单细胞数据中,我们认为一个Barcode(或者说一个液滴)就是一个细胞。通常认为含有细胞的液滴应该含有更多的mRNA,因此其在 UMI 总量上应该与空液滴(背景噪音)存在明显的区分(也就是我们常说的 Barcodes 排序图上的拐点,见图1右图)。然而实际上有时候无法仅通过 UMI 总数很好地区分空液滴和非空液滴。尤其当样本中混杂了不同大小的细胞,小细胞由于其转录丰度较低的特点,可能会难以与空液滴区分。
因此,CellRanger的算法采用了两步法来识别细胞( Lun et al., 2019)。
▶第一步,把每个Barcode所含UMI进行统计,所有大于某一UMI阈值的Barcodes被识别为细胞。这一步保证了高RNA含量的Barcodes被保留。
▶第二步,把剩余未通过阈值的Barcodes与空液滴RNA表达谱进行比较来回收可能的低RNA含量细胞。如果样本中本身所含细胞类型的转录丰度差异大,那么低表达细胞很容易被误判为背景,这种情况下解决方案可参见后续问答3。

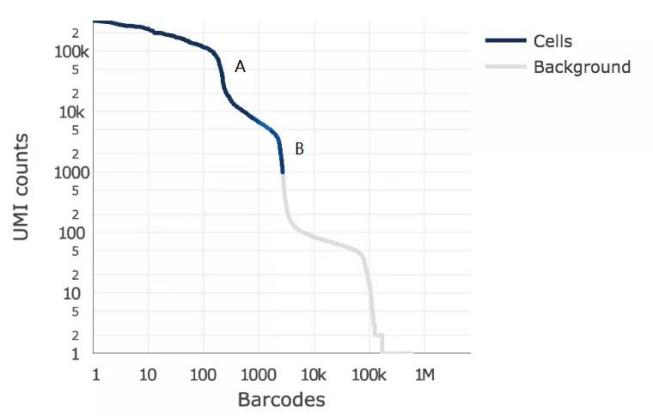
图1 细胞判定算法与Barcode-UMI分布图
2、为什么Cellranger质控显示细胞数目与预期不符,可能原因有哪些?
答:
首先我们要知道,单细胞实验由于其技术原理限制,无法准确保证最终细胞捕获数目一定与预期细胞数分毫不差,小范围内的上下波动都是正常现象。影响细胞数目判定的因素有很多。那么具体有哪些因素会影响最终的细胞捕获数目呢?
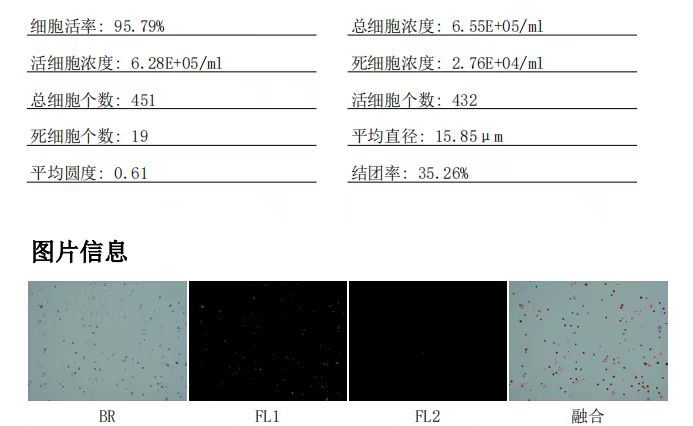
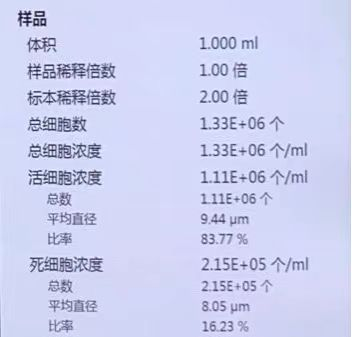
① 样本方面因素,比如:细胞状态(活性和内部状态)、细胞悬液背景杂质、细胞浓度、细胞大小是否均一、细胞形状是否规则、细胞结团率等都有关系。在此额外提一句,什么是细胞内部状态,就是有一些细胞,它虽然检测活性的时候可能是合格的,但是它的内部状态可能已经处在凋亡阶段,那这时候我们是没办法去判断的,那后续上了单细胞的仪器之后,它可能在形成油包水之前或过程中就凋亡了,在这个过程中我们没有办法去判断和检测,因此最后可能会出现质控显示细胞数目与预期不符的情况。
经过我们多年实验经验发现,不同的细胞悬液情况可能导致最终数据呈现也不同:
如果悬液中大细胞与小细胞都比较多,那么由于高丰度RNA与低丰度RNA细胞区分太明显而导致小细胞被误判定为背景噪音区,从而造成计算得到的细胞数目与预期捕获数目相比偏少;
如果悬液中小细胞整体偏多,那么有可能会使细胞与背景噪音无法很好的区分开,从而造成计算得到的细胞数目与预期捕获数目相比偏多;
如果悬液中部分细胞内部状态不好,状态差的细胞在形成液滴的过程中可能会进一步降解,那么也会导致计算得到的细胞数目与预期捕获数目相比偏少;
如果悬液中背景杂质过多,杂质也会进入液滴中,此时容易发现最终判定得到的细胞数目与预期相比偏多。
当然除以上之外也会有其他情况,比如细胞结团过高可能会导致双细胞率偏高等······如果老师有关于细胞悬液制备的任何问题都可随时咨询我们,我们有丰富的样本制备经验~
小鼠主动脉血管解离展示:
人前列腺癌腰椎转移样本解离展示:

非人鼠样本解离展示:

图2 不同类型样本解离结果展示
② 外界因素,比如仪器细胞计数准确度、细胞活性准确度以及人为实验操作等等。但实际上这种情况基本不会发生,因为我们在做之前都会对仪器进行质检,从而避免这种批量性问题。
3、如果cellranger质控显示细胞数目与预期不符,这种情况会影响数据发表吗?
答:
① 单细胞测序发展至今,大部分都会做多个生物学重复,因此个别样本的细胞数捕获数目偏低或偏高对整体数据分析基本无影响,最终分析也不会看单个样本细胞数目绝对值的差异,更多的是看不同组间的细胞类型和占比。
② Cellranger结果里除细胞数目外还有2个指标(中位基因和测序深度)也非常重要。因为总的测序数据量是固定的,当捕获细胞数目变少,意味着平摊到每个细胞的测序数据量增加了(即测序深度增加),测序深度增加会提高中位基因数和测序饱和度指标,也就意味着每个细胞捕获到的基因数目多了,这样可能会提高一些中低丰度基因的检出率。
③ 细胞数目不是决定数据质量的最重要因素,很多高分文章实际上单个样本捕获的细胞数目也并没有那么多,建议可以将重点放在下游的数据挖掘上。比如这篇2023年的Cell Discovery上的小鼠肠道文章(DOI:10.1038/s41421-023-00578-4,IF:33.5),12个小鼠共获得3万多个细胞,平均每个样本细胞数不足3000;2023年的Signal Transduction and Targeted Therapy上的人类胎儿BM基质的细胞的文章(DOI:10.1038/s41392-023-01338-2,IF:39.3),9个胚胎样本最后得到不足9000细胞,平均每个样本细胞数不足1000;2022年的Nature Genetics上的人结直肠癌文章(DOI:10.1038/s41588-022-01088-x,IF:30.8),质控后获得了来自70个样本的总计20w个细胞,平均每个样本细胞数不足3000;
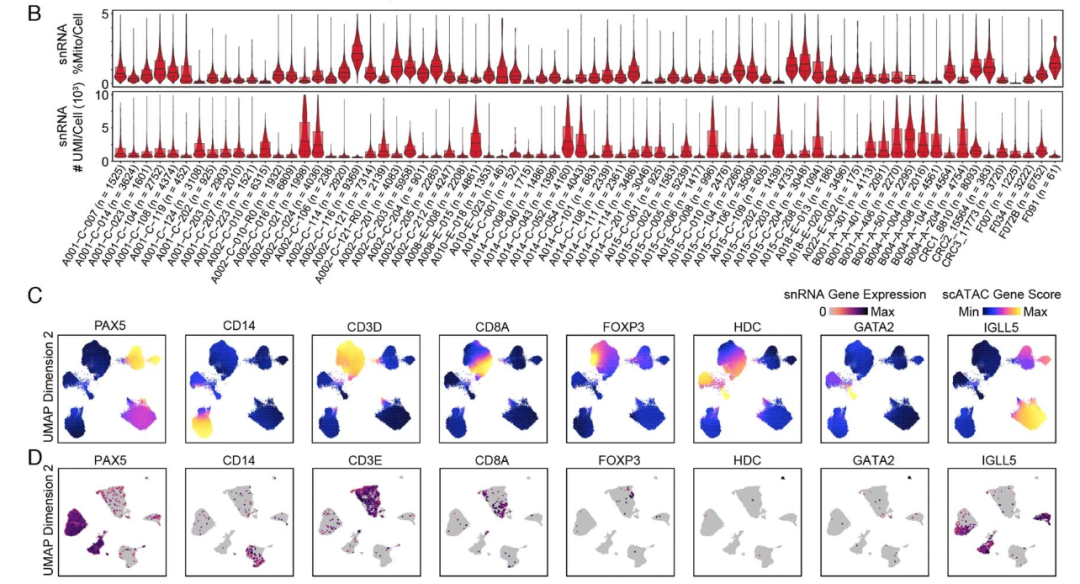
图3 人结直肠癌项目样本数目和总细胞分群结果展示
④ 我们在后续数据挖掘时也可以根据具体情况进行人工调整,比如使用Cellranger自带的参数--force-cells进行尝试调整(该参数是10x官方推荐使用的)。或者是通过细胞过滤参数再进一步把低质量的细胞和异常高表达的细胞过滤掉。

图4 10x官方对于细胞判定不准情况的补充说明
4、什么是低质量细胞,低质量细胞可以去除吗?
答:
低质量细胞一般有两种,①是被误判定为细胞的背景噪音(具体原因见问题1);②是状态较差的细胞。这类细胞一般中值基因/UMI还行(因此不会在细胞过滤的时候被过滤掉),但是会发现这群细胞会泛表达各种细胞的marker,或者是表达的基因大部分都是线粒体基因或核糖体基因(特殊样本除外),这种细胞分析价值不大,并且因为其表达谱异常,通常会被分到一个群里,在注释的时候可以区分出来并直接删除低质量细胞,避免影响后续分析。
参考文献:
[1]https://www.10xgenomics.com/support/software/cell-ranger/algorithms-overview/cr-gex-algorithm
[2]Lun, A., Riesenfeld, S., Andrews, T. et al. EmptyDrops: distinguishing cells from empty droplets in droplet-based single-cell RNA sequencing data. Genome Biol 20, 63 (2019). https://doi.org/10.1186/s13059-019-1662-y
[3]Becker, W.R., Nevins, S.A., Chen, D.C. et al. Single-cell analyses define a continuum of cell state and composition changes in the malignant transformation of polyps to colorectal cancer. Nat Genet 54, 985–995 (2022). https://doi.org/10.1038/s41588-022-01088-x