2024-01-11
细菌核糖体核糖核酸(rRNA)是存在于细菌细胞中的一类RNA分子,约占总RNA 80% - 98%的比例,主要由三个不同长度的部分组成:5S rRNA(120nt)、16S rRNA(1542nt)、23S rRNA(2906nt),rRNA作为核糖体中起主要作用的结构成分,其主要作用是:1、具有肽酰转移酶的活性;2、为tRNA提供结合点;3、为多种蛋白质合成因子提供结合位点;4、在蛋白质合成起始时,参与同mRNA选择性的结合以及在肽链的延伸中与mRNA结合。rRNA在蛋白质合成过程中扮演着关键的角色,高含量的rRNA可以确保蛋白质合成过程的高效进行。
虽然rRNA在蛋白质合成过程中起到关键作用,但是在研究遗传调控机制或NGS测序技术等方面,还是要看信使核糖核酸(mRNA)。而在微生物单细胞转录组测序中,由于原核生物mRNA缺乏PolyA尾,无法效仿真核生物进行mRNA的捕获,从而选择进行rRNA的消减。目前应用于微生物单细胞转录组测序的rRNA去除方法主要有:基于探针的RNase H介导的rRNA去除方法;基于CRISPR-Cas9的rRNA去除方法。
基于探针的RNase H介导的rRNA去除方法
RNase H是一种核糖核酸内切酶,可特异性切割DNA、RNA杂交双链中的RNA分子,同时保证DNA序列的完整性。针对细菌rRNA序列设计的高特异性DNA探针,通过DNA探针与细菌rRNA序列杂交结合,进而使用RNase H可特异性降解DNA/RNA杂合链上的RNA链以及使用DNase I去除寡核苷酸探针,来达到去除细菌rRNA的目的(图1)。该方法主要针对rRNA,在微生物单细胞测序技术中需要在胞内进行rRNA的去除,而在对微生物细胞悬液进行微流控上机前应该尽量减少实验操作步骤,减少对微生物细胞样品的影响。其次该方法非常依赖细胞透化效果的好坏,透化效果直接关系到寡核苷酸探针与rRNA序列的结合程度,间接决定rRNA的去除效果,因此实验数据存在不确定性。
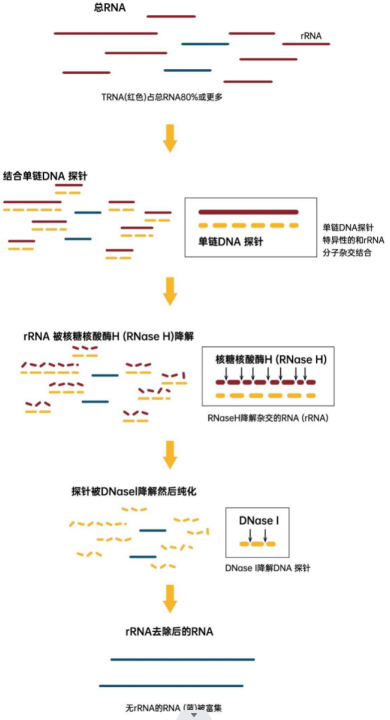
图1 使用特定探针进行RNase H介导的rRNA去除
基于CRISPR-Cas9的rRNA去除方法
天然的CRISPR-Cas系统在细菌适应性免疫中发挥作用,可在不损害细菌基因组的情况下去除侵入的噬菌体DNA。该机制涉及Cas核酸酶对外源DNA的特异性切割。该系统可以通过提供特定的靶向rRNA序列或其他丰富序列的引导RNA一起使用,在文库制备完成后可以对rRNA或其他高丰度转录本的文库片段进行降解。在微生物单细胞测序技术中,将准备好的测序文库与Cas9核酸酶一起孵育,该核酸酶已与特定的引导rRNA预复合。然后在文库中切割包含任何目标序列的所有文库片段。孵育之后是进行磁珠纯化,以去除短的文库片段和被切割的文库片段。由于此步骤去除了大部分文库片段,剩余的文库片段将通过另一轮PCR重新扩增(图2)。该方法很适合在文库制备前不宜进行rRNA去除的样品,但是其引导RNA设计相当复杂,需要很强的专业背景,此外测序文库需要进行两轮PCR扩增,可能会增大序列扩增的偏倚。
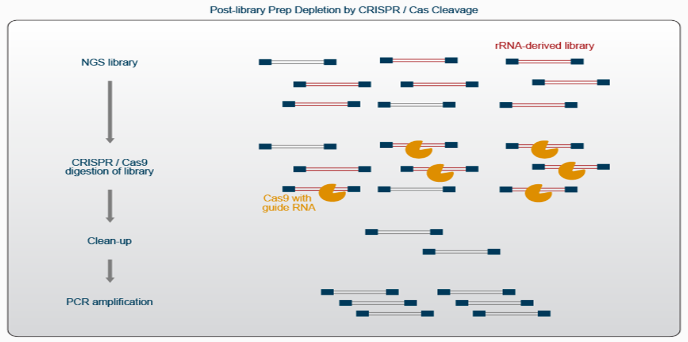
图2 CRISPR-Cas9指导文库制备后rRNA的去除
-

以上就是目前在微生物单细胞测序领域出现的rRNA的去除方法。目前本公司推出的新产品smRNAPSN-seq采用的rRNA去除方法,经多种微生物细胞样品测试,rRNA占比最低可达5.82%,后期我们会进一步优化rRNA去除方法,加强数据稳定性,进一步降低rRNA占比(图3)。
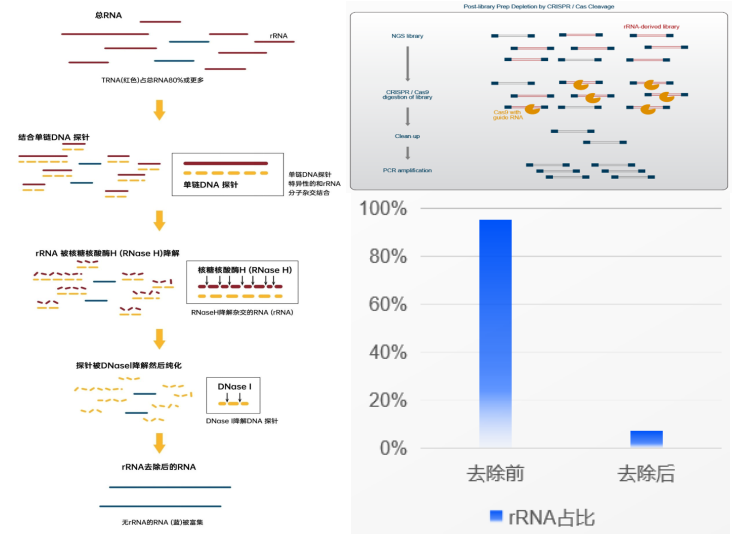
图3 smRNAPSN-seq的rRNA占比


