2024-01-22
好消息!派森诺宏基因组Binning分析全新流程现已研发完成!!!
Binning分析是宏基因组分析的高级分析,基于Binning分析可以对宏基因组的结果进行延深和拓展。相信大家和小派一样对于宏基因组Binning分析有很多问题,那么和小派一起来看看吧!
01、Binning分析是什么? 宏基因组分箱(Binning)是一个基于宏基因组测序得到的reads或contig 基因序列进行聚类分箱的过程。类似于将我们的不同颜色的衣服整理分类到不同的衣柜中。重要的是,binning分箱可以精细到单个纯菌的基因组水平。同时,我们也可以基于得到的单菌基因组进行更加深入的分析。 宏基因组Binning的原理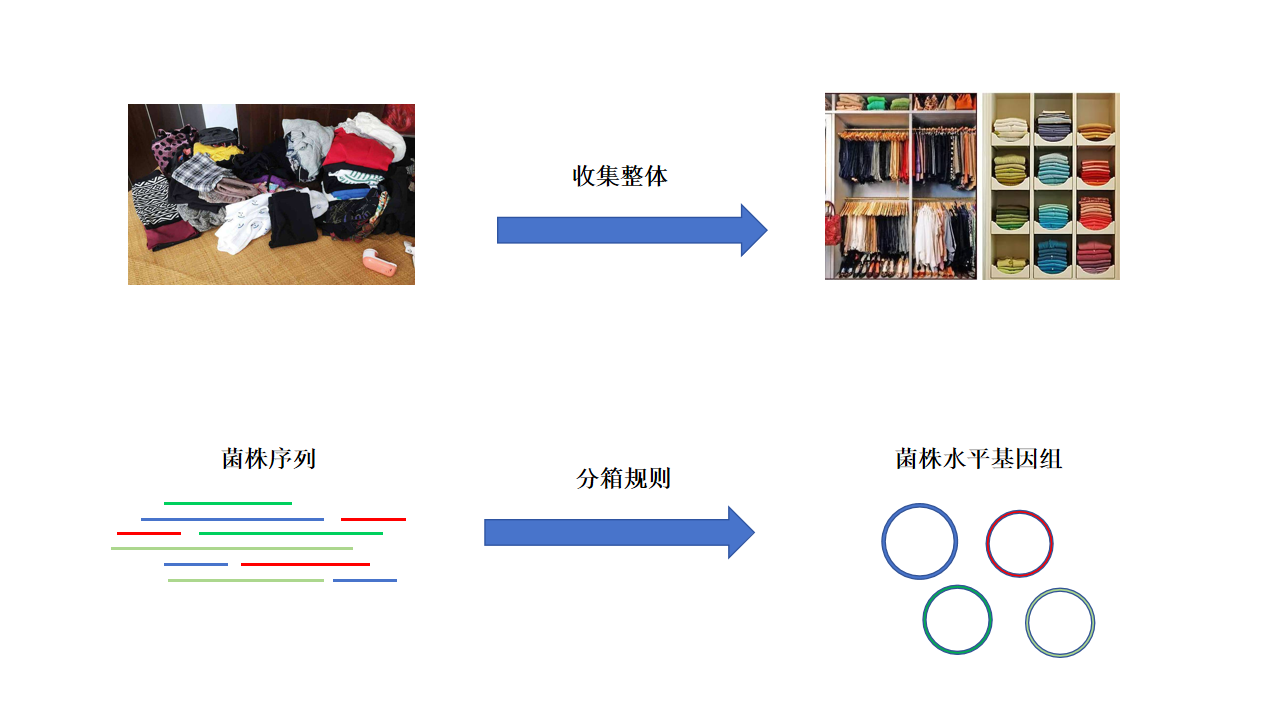
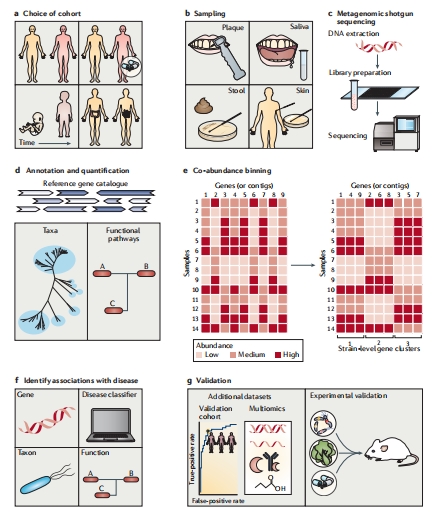
02、Binning 有什么用? (1)获得单菌基因组草图 通过对Binning得到的bins进行后续组装,可以得到许多无法培养和未知的细菌、古菌、病毒的基因组草图。接着就可以进行单一菌株的基因和功能注释、比较基因组分析、进化分析等,使我们得以洞察这些无法在实验室培养获得的菌株的微进化机制,宿主的互作机制和营养代谢途径等。 基于宏基因组Binning分析单菌草图构建的系统发育树 (2)联合分析 得到样本中的keystone的单菌、进行系统发育分析、MWAS整合分析。从地球上最深的马里亚纳海沟到探索月球生物资源,从植物内生菌到人体病原菌,Binning可以挖掘极端环境中的新物种和暗物质、植物和微生物的共同进化机制、回答临床微生物与宿主的互作机制。当然,Binning也可以与其他组学、metadata进行整合分析,从而深入研究细菌、古菌、病毒、真菌与宿主的功能机制。 基于宏基因组Binning进行的关联分析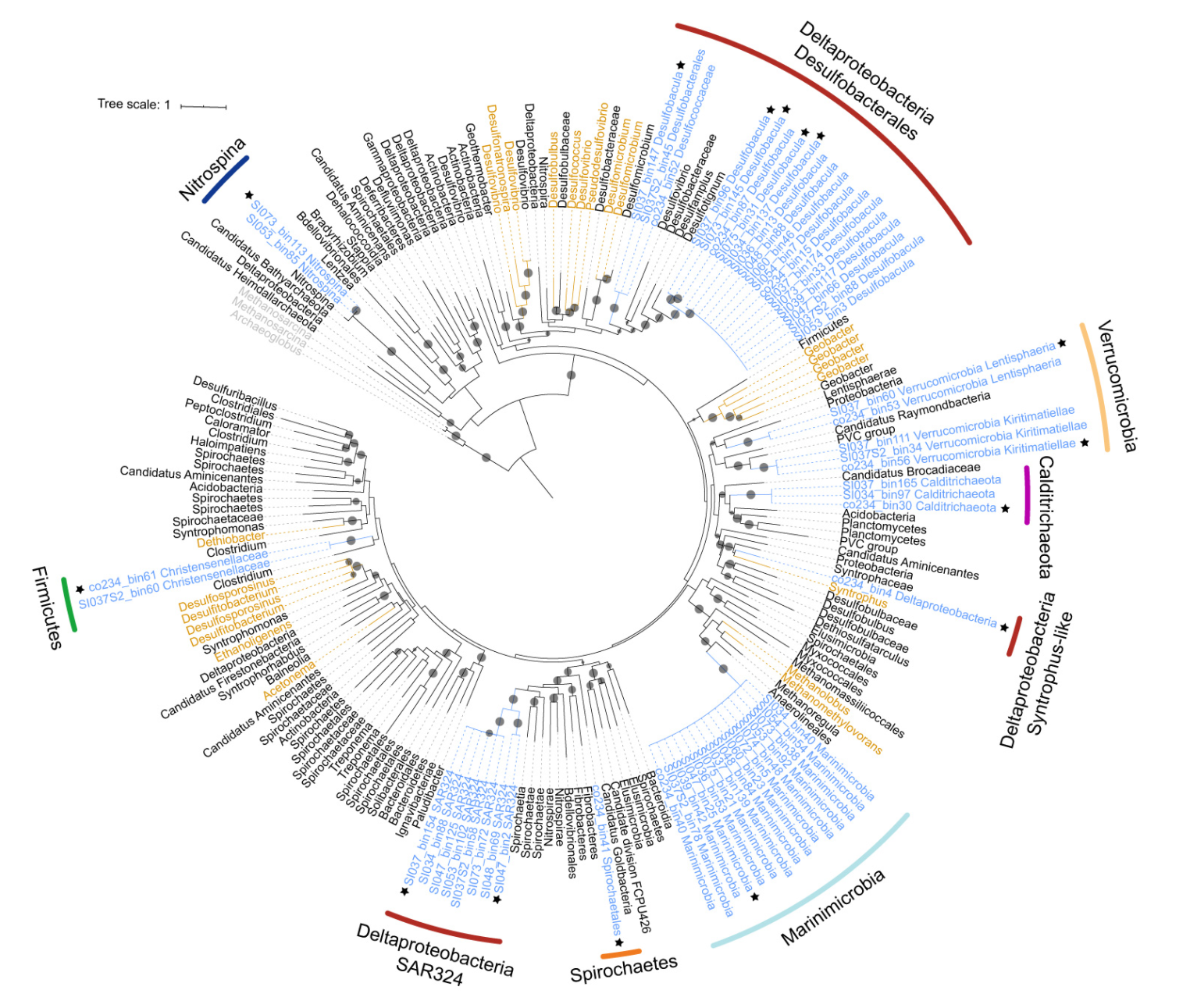
03、我做细菌基因组完成图和框架图就可以获得单菌的基因组,为什么还要做Binning分析? 细菌基因组完成图和框架图是获得细菌基因组最好的一种方法。但是细菌基因组完成图和框架图需要我们从样品中分离细菌的纯菌,获得单菌纯度比较高的DNA,而我们知道自然环境中大约99%的微生物是无法培养和找到合适的分离条件的,这时候就要借助Binning分析获得样品中无法培养的微生物基因组
04、我做Binning分析就一定能获得目标微生物的基因组吗? Binning分析是获得无法培养的微生物基因组最好的一种方法。Binning分析的结果与测序的数据量,测序的数据的数据质量,序列的拼接效果和目标微生物基因组的复杂程度都有关系。所以不一定能100%获得目标微生物的基因组。但是派森诺宏基因组Binning全新升级,使用全新新流程,更加严格的参数,更加多样性化的分析,提高您获得目标微生物基因组的可能。 派森诺宏基因组Binning分析流程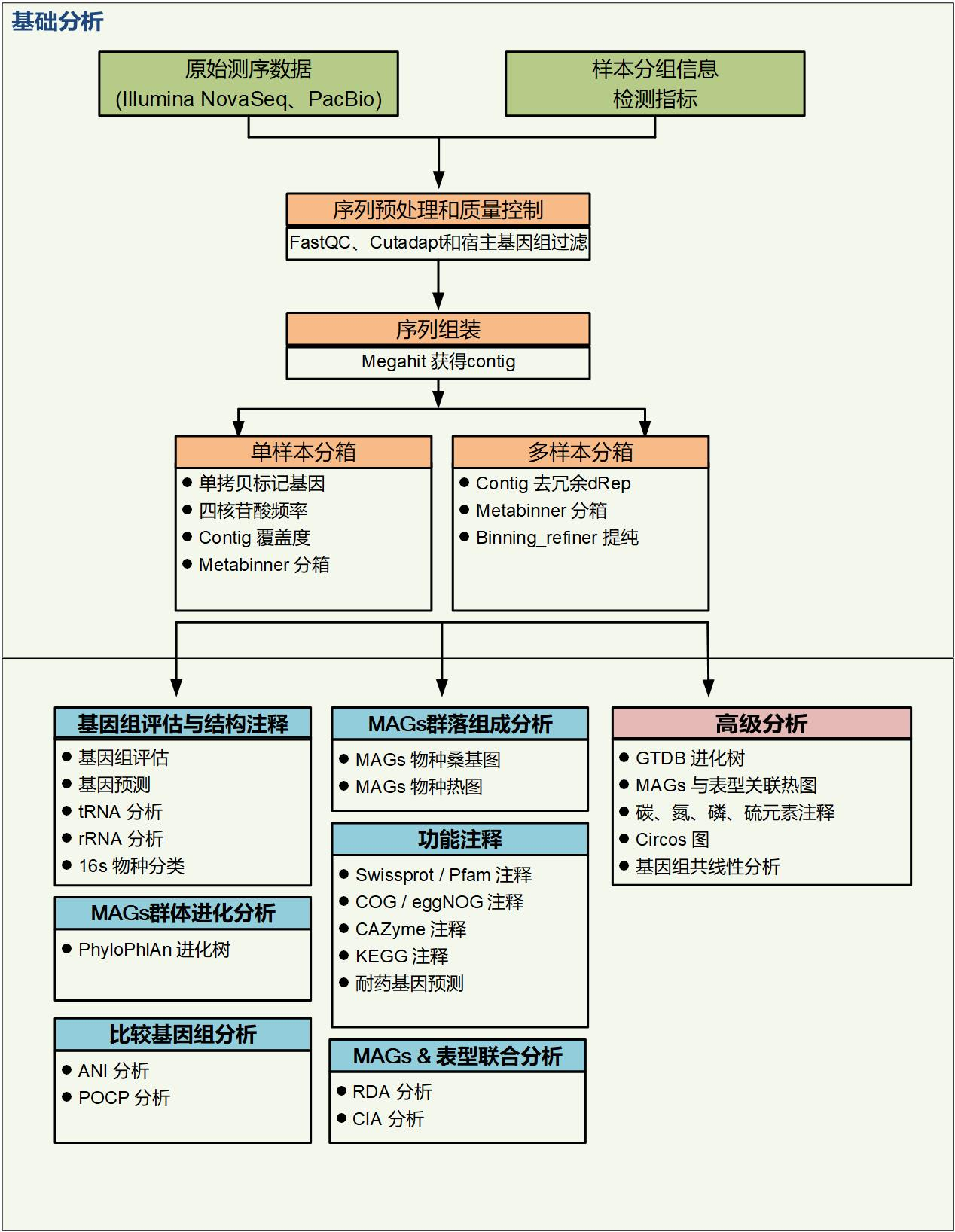
05、派森诺的宏基因组binning分析结果有哪些? 我们升级的分析结果十分丰富,其中包含了基础的bin的统计查找、物种的功能注释等内容。更重要的是,老师们可以自由选择系统发育分析的方法,比如Phyloplan 、GTDB等。此外,我们也可以将其他的元数据和bins进行高级关联分析,从而解决各研究领域的科学问题。 派森诺宏基因组Binning分析结果预览,更多结果请查看派森诺宏基因组Binning分析报告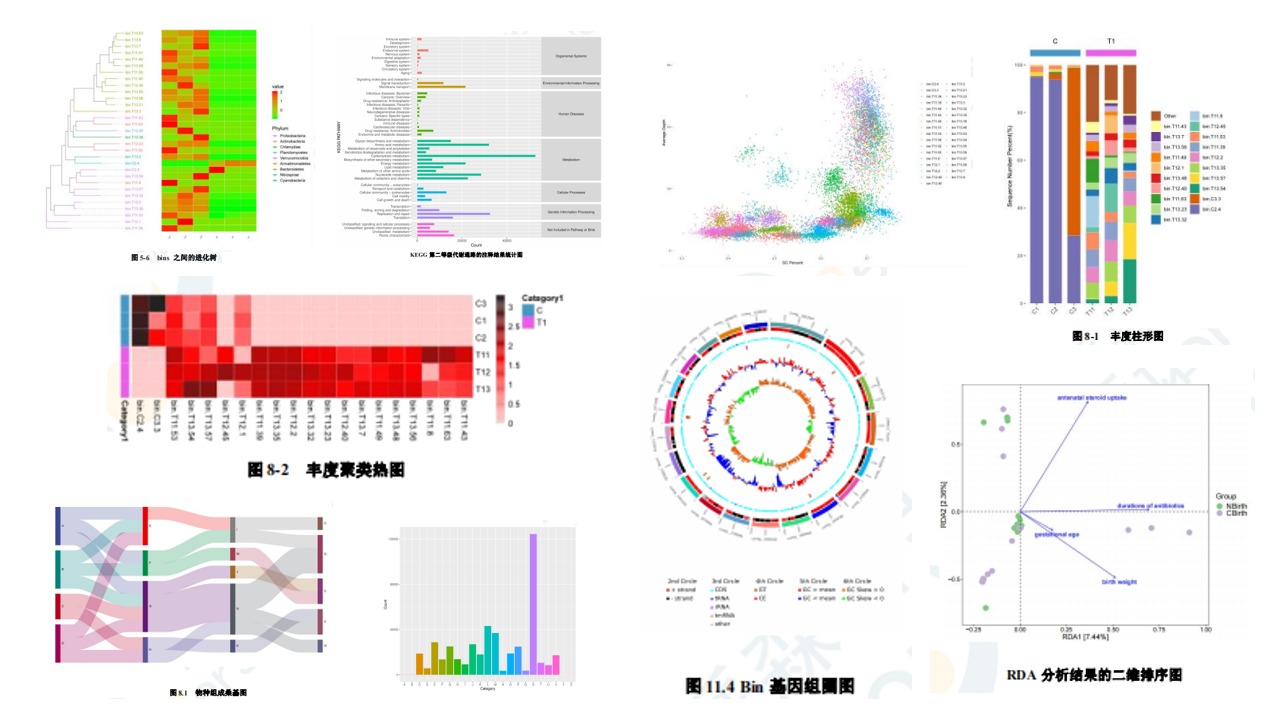
06、派森诺的宏基因组binning分析有哪些项目经验? 派森诺宏基因组一直是派森诺的王牌产品,宏基因组binning分析作为宏基因组的高级分析也是积累了大量经验。 派森诺宏基因组Binning发表的文章 点击标题查看原文 【派森诺项目文章】16S扩增子和宏基因组测序揭示依赖重砷的生物固氮代谢机制! 【派森诺项目文章】 宏基因组学和机器学习共同辅助识别水稻根部微生物中镉积累特征的生物标志物! 【派森诺项目文章】荣登10分ISME!新研究发现锑金属污染下,微生物修复的新机制! 【派森诺项目文章】Microbiome:固氮内生菌可提高中国芒草生态适应性
最后的最后,Binning的强大之处不仅在于发表高分文章,如果想要获得更多可能,快来探索派森诺宏基因组Binning分析吧! 参考文献 Lin H, Ascher DB, Myung Y, Lamborg CH, Hallam SJ, Gionfriddo CM, Holt KE, Moreau JW. Mercury methylation by metabolically versatile and cosmopolitan marine bacteria. ISME J. 2021 Jun;15(6):1810-1825. doi: 10.1038/s41396-020-00889-4. Epub 2021 Jan 27. PMID: 33504941; PMCID: PMC8163782 Wang J, Jia H. Metagenome-wide association studies: fine-mining the microbiome. Nat Rev Microbiol. 2016 Aug;14(8):508-22. doi: 10.1038/nrmicro.2016.83. Epub 2016 Jul 11. PMID: 27396567.


