
2024-02-23
1、空间转录组学的发展历程
自20世纪90年代以来,随着高通量测序技术的迅猛发展,转录组学作为研究细胞内所有转录RNA的学科逐渐崭露头角。研究者们开始全面揭示基因表达模式和调控网络。然而,传统转录组学技术无法研究细胞内不同亚细胞结构和环境对基因表达的影响。为了更全面地理解基因表达的调控过程,空间转录组学迅速兴起,致力于揭示基因在细胞内的空间分布、亚细胞位置和相互作用,为理解基因表达的空间调控机制提供了新的视角。
转录组技术的发展经历了三个重要阶段:第一个阶段是对大量混合细胞进行转录组测序(Bulk RNA),获得的是一个大细胞群体中单个基因的平均表达水平,但无法展示单个细胞的转录表达水平。第二个阶段是单细胞转录组测序(Single cell RNA sequencing,scRNA-seq),能够在单个细胞水平上构建每个细胞的表达谱,反映细胞间的异质性。但是在单细胞悬液制备过程中会丢失空间位置信息,而这是器官功能的关键特征之一。在对这一需求的驱动下,Joakim Lundeberg课题组于2016年提出了空间转录组学的概念,并发表了第一个基于原位捕获RNA的空间转录组学技术。空间转录组测序的出现使细胞的空间位置信息和基因表达数据相结合,可以同时获得细胞的空间位置信息和基因表达数据。这为组织细胞功能、微环境互作、发育过程谱系追踪以及疾病病理学等多个领域提供了重要的研究手段。随后,一系列能够进行高通量原位RNA检测分析的技术都被归为空间转录组学技术的范畴。尽管这些技术的原理各不相同,但它们有一个共同点,即记录所检测RNA分子的空间位置信息。

图1 Bulk,Single Cell和Spatial分析的维度比较
20世纪90年代后期,一些关于空间转录组学的文章从激光捕获显微解剖(LCM)、微阵列或RNA-seq和单分子荧光原位杂交(smFISH)等技术的使用开启了空间转录组学的发展历程。20世纪80年代的一些技术,虽然现在并不称为“空间转录组学”,但它也是在空间中获得转录信息。加州理工(Caltech)大学Lambda Moses和Lior Pachter老师在其在线电子书Museum of Spatial Transcriptomics中将这部分空间转录组技术称为“前传时代”[1]。前传时代的技术为我们刻画了空间转录组的一般技术特点:成像,定位,表达量。同时,能够看到“前传时代”的空间转录组一直在努力的一个方向是成像和单细胞(分辨率),而表达量往往不是高通量获得的。尽管由于受限于分辨率,易操作性,可用的软件,丰富的数据库等原因,尤其是基于NGS的高通量表达量的获取在前传时代中是没有的,使得“前传时代”很多技术已经式微,但“前传时代”还是进行许多有益的尝试,甚至可以说是为当下时代的空间转录组技术提供了参考。
相比之下,在“前传”之后的技术Lambda Moses和Lior Pachter将其称之为“当前时代”。许多“当前时代”的技术的基础都是在20世纪70年代到21世纪初的几十年里建立起来的。例如,微阵列技术于1995年首次报道[2],最初用于量化与打印在载玻片上的cDNA杂交的转录本,但很快在1999年被用于量化LCM样本的转录组[3]。当前流行的技术,如空间转录组学(ST)和10X Visium均依赖于这种微阵列技术,可以从安装在微阵列载玻片上的组织中捕获转录本。
近年来,空间转录组学技术经过了迅速发展,在分辨率和通量方面取得了显著提升,空间转录组也于2020年被《Nature Methods》评为年度技术方法,同时在2022年被《Nature》杂志评选为值得关注的7大年度技术之一。2023年空间组学再获殊荣,成为《World Economic Forum》评选出的目前最有潜力、对世界产生积极影响的十大技术之一。
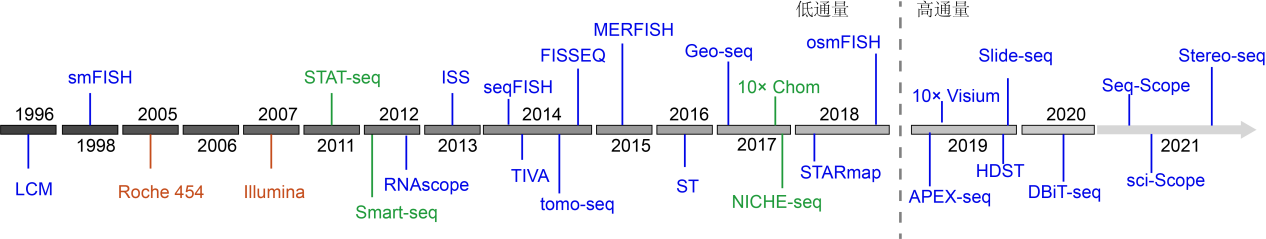
图2 空间转录组研究技术的发展

图3 空间转录组技术近年所获荣誉
2、空间转录组学的技术方法
空间转录组学的概念最初源自FISH(fluorescently-labeled RNA sequences),利用带有荧光的探针与目标结合来实现靶基因的原位检测,从而实现对定性定量和相对位置的分析。根据原理的不同,空间转录组技术可以分为基于成像[4]和基于测序[5]两大类。基于成像的空间转录组技术主要通过显微镜观察显色基团与探针结合后的mRNA进行原位成像。基于成像的空间转录组技术又能够进一步分为原位杂交(in situ hybridization, ISH)和原位测序(in situ sequencing, ISS)。而基于测序的空间转录组技术则无需事先知道基因序列信息,它能够从组织中提取mRNA并同时保留其空间位置信息,随后通过高通量测序技术对mRNA进行检测和分析。
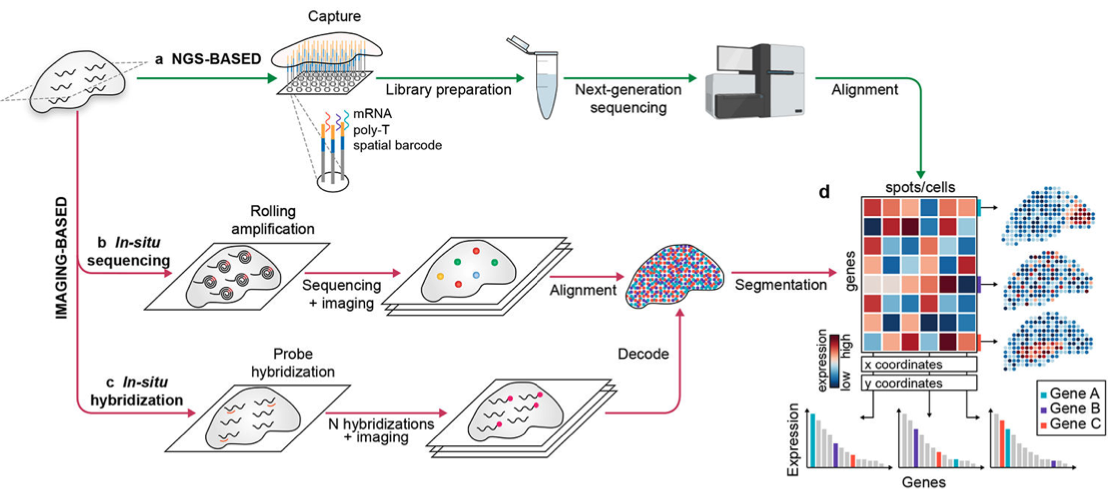
图4 空间转录组技术原理示意图
2.1 基于原位杂交的空间转录组技术
基于原位杂交(ISH)的方法是一种基于成像的技术,其基础是利用互补荧光探针与目标序列进行杂交来进行检测。2014年,加州理工学院的蔡龙教授开发出连续荧光原位杂交(sequential fluorescence in situ hybridization SeqFISH)技术[6],该技术即为顺序杂交的多重smFISH方法。通过4种颜色,8轮杂交(48=65536)足以编码人类或小鼠基因组中的所有基因。另一种比较有代表性的技术是MERFISH[7],该技术的核心原理是将mRNA和其他RNA分子定位于细胞中以便于检测其表达水平,靶向探测的过程分为两个阶段:荧光原位杂交和成像分析。通过设计杂交探针Panel,在设计时输入我们关注的Marker基因,使得每一个RNA都被分配了一个唯一的二进制条形码。大多数其他基于smFISH的技术,如HybISS[8]和splitFISH[9],也都是使用类似seqFISH或MERFISH的条形码[10, 11]。
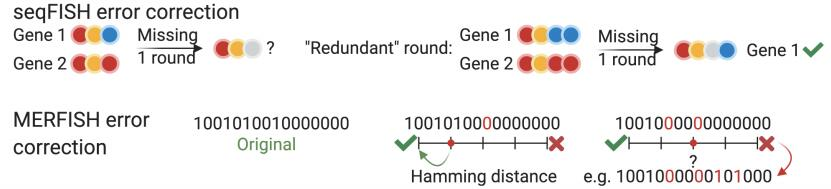
图5 基于原位杂交的空间转录组典型技术原理示意图
2.2 基于原位测序的空间转录组技术
ISS方法通过测序产生空间转录组信息。具体而言,该方法通过逆转录RNA、滚动循环扩增和测序等步骤实现。Ke等人采用基于挂锁探测,滚动环扩增(RCA)和测序连接化学的方法,成功研究了约50个靶向基因在癌症、结核病和大脑发育中的表达情况[12]。STARMap则在此基础上结合了水凝胶化学的进展,改进了挂锁和引物的设计,实现以单细胞分辨率同时定位160到1020个基因[13]。此外,原位测序还具备非靶向分析的潜力。以FISSEQ为例,该方法利用随机六聚体引物在固定细胞中逆转录RNA,然后通过cDNA的环化生成非靶向的测序文库[14]。FISSEQ进一步提高了原位测序技术的检测通量,能够获得包括基因表达、RNA剪接和转录后修饰等在内的全基因组级别的基因表达图谱,并保留了它们的空间位置信息。与单细胞RNA-seq相比,FISSEQ对于功能重要的转录本的富集程度更高,达到10倍以上,但其局限性在于测得的基因数目较少,测序深度较低,从而限制了其灵敏度。
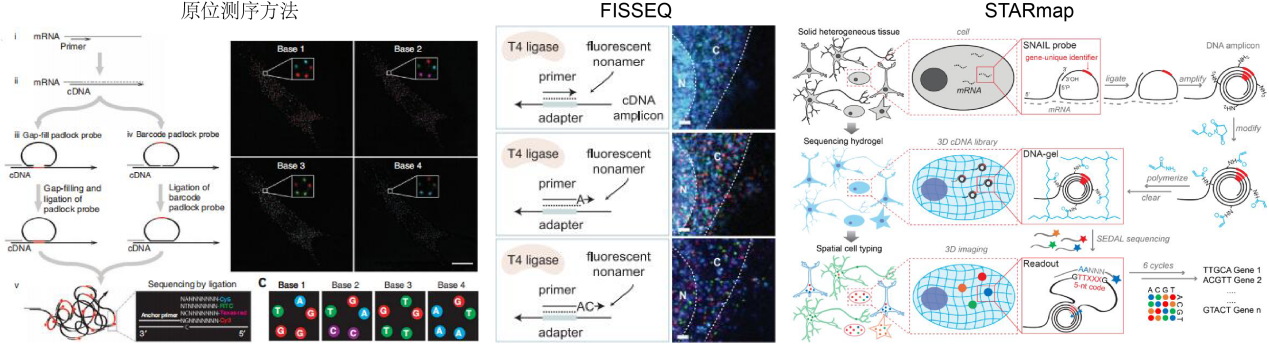
图6 基于原位测序的空间转录组代表性技术原理示意图
2.3 基于高通量测序的的空间转录组技术
基于NGS的方法源于单细胞RNA-Seq方法的概念创新,在文库准备之前添加一个空间条形码[5]。2016年,瑞典皇家理工学院的Joakim Lundeberg利用基因芯片技术开发了首个基于NGS的空间转录组学方法[15]。这项方法的核心创新在于,在逆转录之前,在空间条形码微阵列载玻片上捕获多聚腺苷化RNA,以确保每个转录本可以通过独特的位置分子条形码映射回其原始点[16, 17]。2018年底,10×Genomics宣布收购了这项技术,并于2019年发布了Visium空间基因表达解决方案。Rodriques等人基于NGS技术发明了一种名为Slide-Seq的空间转录组解决方案,该技术使用直径为10 μm的磁珠覆盖的玻片,这些磁珠带有已知的barcode修饰[18]。在这个方法中,每个随机条形码的位置是通过原位索引获得的,实现了更高分辨率(10 μm),提高了灵敏度(每个珠子500个转录物)。Vickovic等人提出了一种名为高分辨空间转录组(HDST)测序方法[19]。该方法通过在载玻片表面雕刻大量直径为2 μm的小孔,并将直径为2 μm的硅胶磁珠分配到这些孔中。通过在磁珠表面连接带有特定barcode序列、多种唯一分子标识符(UMIs)和poly-dT的寡核苷酸链,可以捕获对应位置细胞的mRNA进行反转录,并进行文库构建和转录组测序。DBiT-Seq方法采用了平行微流控通道将DNA条形码传递到组织载玻片表面,将两组条形码A1-50和B1-50进行交叉流动,然后原位连接,得到组织像素的二维马赛克,每组都包含一个独特的完整条形码[20]。Stereo-seq则使用以阵列模式沉积的随机条形码DNA纳米球,以实现纳米级分辨率[21]。Seq-Scope使用基于Illumina合成测序平台对随机条形码单分子寡核苷酸进行固相扩增,实现了亚细胞分辨率的空间条形码,可用于可视化核和细胞质转录本[22]。Pixel-Seq采用了一种创新的方法,通过在定制的凝胶表面上排列“连续”的polony寡聚物,改进了空间条形码方法,与现有方法相比,分辨率提高了200倍[23]。利用这些技术,可以研究正常和疾病组织的细胞组成及其与基因表达的关系,也可用于发现新的组织生物标志物。
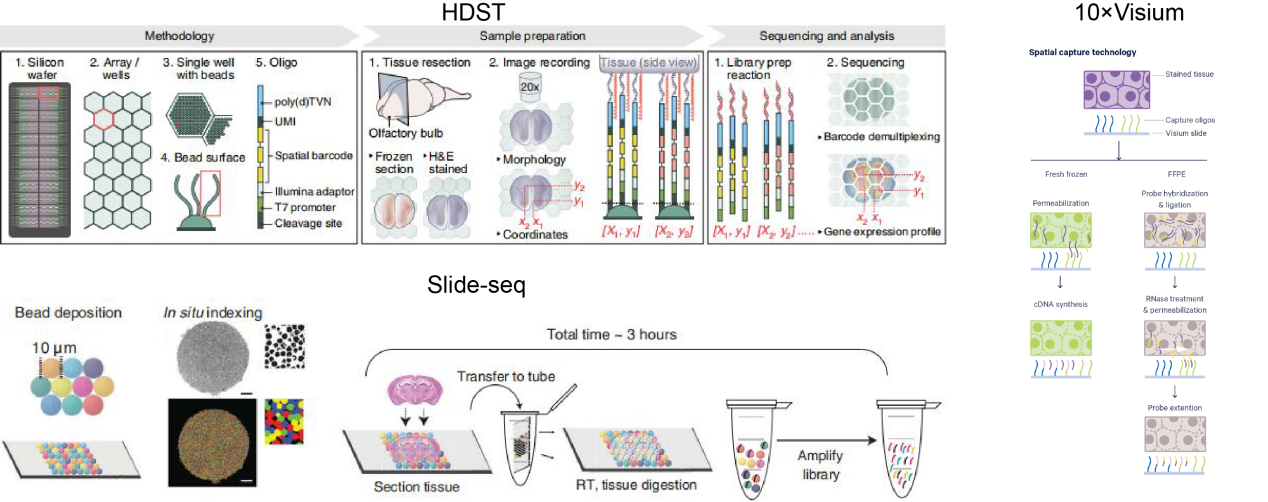
图7 基于高通量测序的空间转录组代表性技术原理示意图
2.4 基于活细胞标记的空间转录组技术
上述几类空间转录组技术,需要对组织进行切片,而组织一旦切片,就无法获得完整单细胞的基因表达信息,当前的捕获方法难以从单个细胞中分离出mRNA而不损害相邻组织。2014年,Lovatt等开发了一种新的空间转录组方法——TIVA[24],第一次非破坏性地从活细胞中捕获mRNA。该方法设计了一种光激活的标签(TIVA-tag),TIVA-tag包括一段poly(U)序列和两个poly(A)片段,该标签通过二硫键连接的细胞穿透肽穿透细胞膜,以使TIVA-tag进入细胞,之后在细胞中进行光裂解,使得poly(U)序列暴露出来,并与细胞中mRNA的poly(A)尾退火连接。组织裂解后,用链霉素亲和素和磁珠纯化得到TIVA-tag-mRNA结合产物。之后,将mRNA洗脱下来,进一步用于转录组分析。
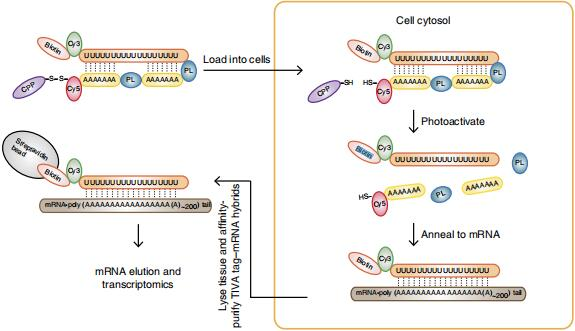
图8 TIVA技术原理图
3、空间转录组学的应用前景
空间转录组学在生命科学研究中具有广阔的应用前景和应用领域。由于空间转录组技术提供了一个无偏的空间组成图像,已被用于生成组织图谱,作为参考提供了有价值的资源。它可以帮助我们了解组织发育和器官形成过程中基因表达的时空动态变化,揭示细胞类型和基因表达之间的关系,以及相邻细胞之间的相互作用。此外,空间转录组学还可应用于疾病研究,通过分析正常组织和疾病组织的空间转录组数据,深入理解疾病的发病机制,寻找新的治疗靶点。
在神经生物学方面:空间转录组学可以将脑细胞类型与形态学、生理学和连接性相关的功能学联系起来,从而全面了解脑神经回路,系统地了解脑细胞类型及其在组织中的定位,并解析其工作机制。
在发育生物学方面:空间转录组学图谱可用于阐明心脏发育、精子发生和肠道发育的空间动力学,解析细胞的分化轨迹和发育谱系及其在组织中的空间定位研究组织器官的正常发育。
除了正常的发育和生理之外,空间转录组学很适合研究疾病中的组织结构紊乱。例如,空间转录组学能够识别在癌症中起作用的机制,即正常生理功能的组织结构发生改变。空间转录组学还为神经退行性疾病、感染和炎症过程以及风湿病中组织失调机制提供了见解。
总而言之,空间转录组学为我们提供了一种全新的视角来研究基因表达和细胞组成在空间维度上的分布,对于理解生物系统的复杂性和疾病的发生机制具有重要意义,未来将在更多的领域发挥作用,并为生命科学研究带来更多突破。
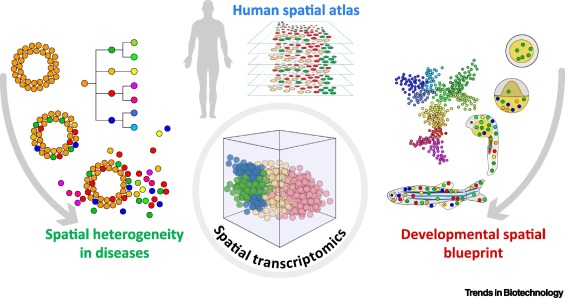
图9 空间转录组学的应用
4、主流空间转录组技术的解析
空间背景对推断更深层次的生物学意义的重要性是毋庸置疑的,但技术掣肘,例如敏感性、组织类型依赖性以及获取详细单细胞信息的能力有限等使得不同类型的空间转录组技术其应用场景也各不相同。下表对几种代表性空间转录组技术进行了归纳,并从样本类型、空间分辨率、灵敏度、细胞通量等角度将各种主流空间转录组技术进行了对比。
表1 主流空间转录组技术对比
应用方向 | 发现性工具 | 应用性工具 | |
原理 | 基于NGS | 基于ISH | 基于ISS |
代表技术 | 10×Visium/HD、HDST、Slide-seq、Stereo-seq | smFISH、seqFISH、MERFISH、RNA-Scope | HybISS、STARmap、Mip-Seq,10×Xenium |
相关公司 | MGI、10×Genomics | Vizgen、nanoString | 鲲羽生物、10×Genomics |
样本类型 | 新鲜组织、石蜡组织 | 所有适用于原位杂交的样本 | 所有适用于原位杂交的样本 |
空间分辨率 | 0.5-55 μm | 单细胞水平 | 单细胞水平 |
灵敏度 | 仍然低于scRNA-seq | ~100% | ~96% |
细胞通量 | 高 | 较高 | 低 |
检测目标 | mRNA/全转录组 | 靶向 | 靶向 |
自动化 | 自动化 | 半自动化 | 半自动化 |
检测周期 | 长 | 中 | 短 |
5、空间转录组学面临的挑战
目前,空间转录组学面临着几个挑战和不足。
第一,是在技术上的限制。当前的空间转录组学技术在精度、灵敏度和重复性等方面仍需要提高。这也使得仍然需要在数量和质量之间进行权衡。ST和Visium的分辨率有限,检测效率低,但更容易应用于大面积的组织和全转录组。相比之下,基于smFISH的技术具有亚细胞分辨率,拥有超过80%的检测效率,但当应用于10000个基因时效率会降低,而且更难应用于更大的组织区域。由于仍然存在挑战,收集数据的新技术也在不断发展。
第二,与过去相比当下时代空转技术更加精细化。虽然商业化的LCM、ST和Visium已经传播得非常广泛,但基于smFISH的各种高质量技术大多未能走出研发实验室。这可能是由于构建自定义细胞的困难以及针对不同组织定制统一操作规程的挑战,组织中的不同细胞类型、状态和位置的差异,可能导致不同的基因表达模式。如何处理这种细胞异质性并精确地识别不同细胞类型仍然是一个难题。需要开发更高级的算法和技术来解决这个问题,以获得更准确的基因表达图谱,也有部分是因为缺乏统一的、开源的和文档化的软件平台来处理数据。
总而言之,虽然空间转录组学在生命科学研究中具有巨大潜力,但仍需要进一步改进技术和方法,并解决当前的挑战和不足。随着技术的不断发展和方法的改进,相信空间转录组学将为我们提供更多有关基因表达和细胞组成的宝贵信息,为生命科学研究带来更多突破。
参考文献
[1] MOSES L, PACHTER L. Museum of Spatial Transcriptomics[J]. 2020.
[2] MARK SCHENA, DARI SHALON, RONALD W. DAVIS, et al. Quantitative Monitoring of Gene Expression Patterns with a Complementary DNA Microarray[J]. SCIENCE, 1995, 270: 467-470.
[3] LIN LUO, RANELLE C. SALUNGA1, HONGQING GUO, et al. Gene expression profiles of laser-captured adjacent neuronal subtypes[J]. Nature Methods, 1999, 5(1): 117-122.
[4] ZHUANG X. Spatially resolved single-cell genomics and transcriptomics by imaging[J]. Nature Methods, 2021, 18(1): 18-22.
[5] LARSSON L, FRISéN J, LUNDEBERG J. Spatially resolved transcriptomics adds a new dimension to genomics[J]. Nature Methods, 2021, 18(1): 15-18.
[6] LUBECK E, COSKUN A F, ZHIYENTAYEV T, et al. Single-cell in situ RNA profiling by sequential hybridization[J]. Nature Methods, 2014, 11(4): 360-361.
[7] CHEN K, BOETTIGER A, MOFFITT J, et al. Spatially resolved, highly multiplexed RNA profiling in single cells[J]. SCIENCE, 2015, 348(6233).
[8] GYLLBORG D, LANGSETH C M, QIAN X, et al. Hybridization-based in situ sequencing (HybISS) for spatially resolved transcriptomics in human and mouse brain tissue[J]. Nucleic Acids Research, 2020, 48(19): e112-e112.
[9] GOH J J L, CHOU N, SEOW W Y, et al. Highly specific multiplexed RNA imaging in tissues with split-FISH[J]. Nature Methods, 2020, 17(7): 689-693.
[10] MOFFITT J R, HAO J, WANG G, et al. High-throughput single-cell gene-expression profiling with multiplexed error-robust fluorescence in situ hybridization[J]. Proceedings of the National Academy of Sciences, 2016, 113(39): 11046-11051.
[11] MOFFITT J, ; B-M, EICHHORN S, et al. Molecular, spatial, and functional single-cell profiling of the hypothalamic preoptic region[J]. SCIENCE, 2018, 362(6416).
[12] KE R, MIGNARDI M, PACUREANU A, et al. In situ sequencing for RNA analysis in preserved tissue and cells[J]. Nature Methods, 2013, 10(9): 857-860.
[13] XIAO WANG, WILLIAM E. ALLEN, MATTHEW A. WRIGHT, et al. Three-dimensional intact-tissue sequencing of single-cell transcriptional states[J]. SCIENCE, 2018, 361(6400).
[14] LEE J H, DAUGHARTHY E R, SCHEIMAN J, et al. Highly Multiplexed Subcellular RNA Sequencing in Situ[J]. Science, 2014, 343(6177): 1360-1363.
[15] LINNARSSON, STEN, CODELUPPI, et al. Visualization and analysis of gene expression in tissue sections by spatial transcriptomics[J]. Science, 2016, 353(6294): 78-82.
[16] JEMT A, SALMéN F, LUNDMARK A, et al. An automated approach to prepare tissue-derived spatially barcoded RNA-sequencing libraries[J]. Scientific Reports, 2016, 6(1).
[17] SALMéN F, STåHL P L, MOLLBRINK A, et al. Barcoded solid-phase RNA capture for Spatial Transcriptomics profiling in mammalian tissue sections[J]. Nature Protocols, 2018, 13(11): 2501-2534.
[18] SAMUEL G. RODRIQUES, ROBERT R. STICKELS, ALEKSANDRINA GOEVA, et al. Slide-seq: A scalable technology for measuring genome-wide expression at high spatial resolution[J]. SCIENCE, 2019, 363(6434): 1463.
[19] VICKOVIC S, ERASLAN G, SALMéN F, et al. High-definition spatial transcriptomics for in situ tissue profiling[J]. Nature Methods, 2019, 16(10): 987-990.
[20] LIU Y, YANG M, DENG Y, et al. High-Spatial-Resolution Multi-Omics Sequencing via Deterministic Barcoding in Tissue[J]. Cell, 2020, 183(6): 1665-1681.e1618.
[21] FRIEDRICH S, SONNHAMMER E L L. Fusion transcript detection using spatial transcriptomics[J]. BMC Medical Genomics, 2020, 13(1).
[22] CHO C S, XI J, PARK S R, et al. Seq-Scope: Submicrometer-resolution spatial transcriptomics for single cell and subcellular studies[J]. Cold Spring Harbor Laboratory, 2021.
[23] FU X, SUN L, CHEN J Y, et al. Continuous Polony Gels for Tissue Mapping with High Resolution and RNA Capture Efficiency[J]. Cold Spring Harbor Laboratory, 2021.
[24] LOVATT D, RUBLE B K, LEE J, et al. Transcriptome in vivo analysis (TIVA) of spatially defined single cells in live tissue[J]. Nature Methods, 2014, 11(2): 190-196.