2018-08-30

正文:
近期,派森诺生物与上海市儿童医院合作,在《Infection and Drug Resistance》(影响因子3.443)发表论文,通过对儿童中分离获得的25种多重耐药性肺炎链球菌进行重测序分析,研究了基因变异与多重抗药性肺炎链球菌之间的关系。
研究背景:
肺炎链球菌能够引起一系列严重的感染性疾病,在全世界范围内,肺炎链球菌是引起每年582000至926000个幼儿死亡的罪魁祸首。抗菌剂则是治疗肺炎链球菌的药物。但道高一尺,魔高一丈,随之出现了多重耐药性(MDR)菌株,大大降低了抗生素的效力。
多重耐性肺炎链球菌作为可变菌株,其作用机制涉及多种抗生素相关的基因。如肺炎链球菌对β-内酰胺类抗生素的耐药性主要是由青霉素结合蛋白(PBPs、PBP1A、PB2X和PB2B)的突变引起的。但肺炎链球菌复杂的耐药机制仍需要进一步研究。而测序技术,全基因组重测序(WGRS),在流行病学分析、耐药性检测和系统发育研究中非常重要。它通过对基因组测序并与先前测序的参考菌株基因组进行比较,识别两个序列之间的序列多态性,如单核苷酸多态性(SNP)、基因获取、重组、丢失以及插入和缺失(Indels)。本研究对25株肺炎链球菌进行WGRS分析,测定其SNP和Indels等遗传多样性。
研究目的:
遗传变异与耐药性的关系
研究方法:
测序技术:Illumina HiSeq 2000
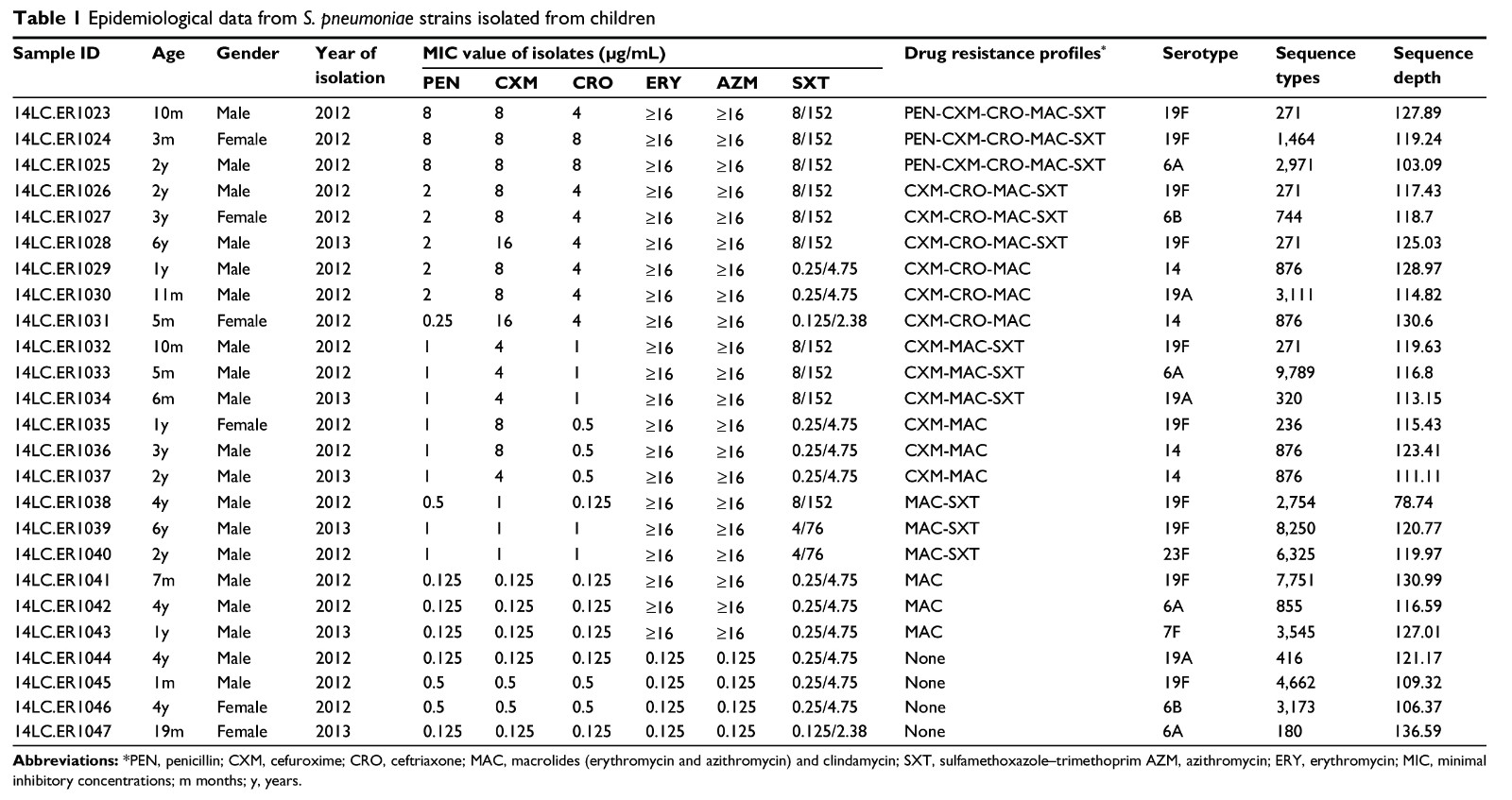
实验对象:25株从儿童体内分离的肺炎链球菌,其中21株为一种或多种耐药性菌株,4株为抗生素敏感性菌株。
实验设计:
对25株分离菌进行重测序,基于SNP进行进化树重构以及主成分分析,并比较敏感菌株和抗菌耐药菌株之间的Indels和SNPs。
研究结果
1. 肺炎链球菌基因组分析
分离的25株肺炎链球菌的序列的大小为2.1 Mbp,占基因组总估计大小的90%。总GC%含量为39.5%,基因组含有2200~2400个开放阅读框。在不同耐药性的分离株中存在相似数量的缺失(范围131~171)。编码区中非同义/同义突变的SNP数目和非编码区的非同义/同义突变也均在16103~28128之间。
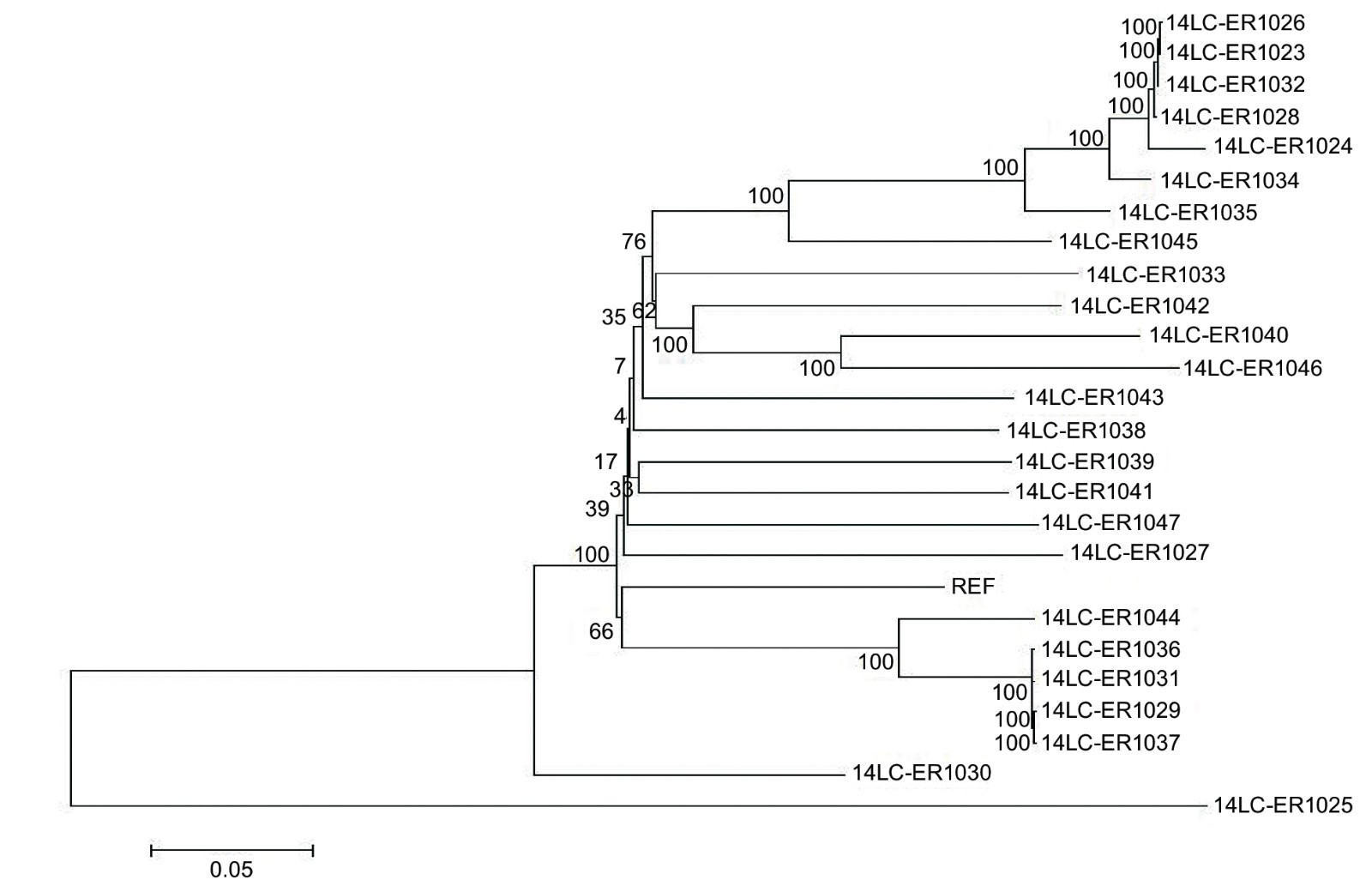
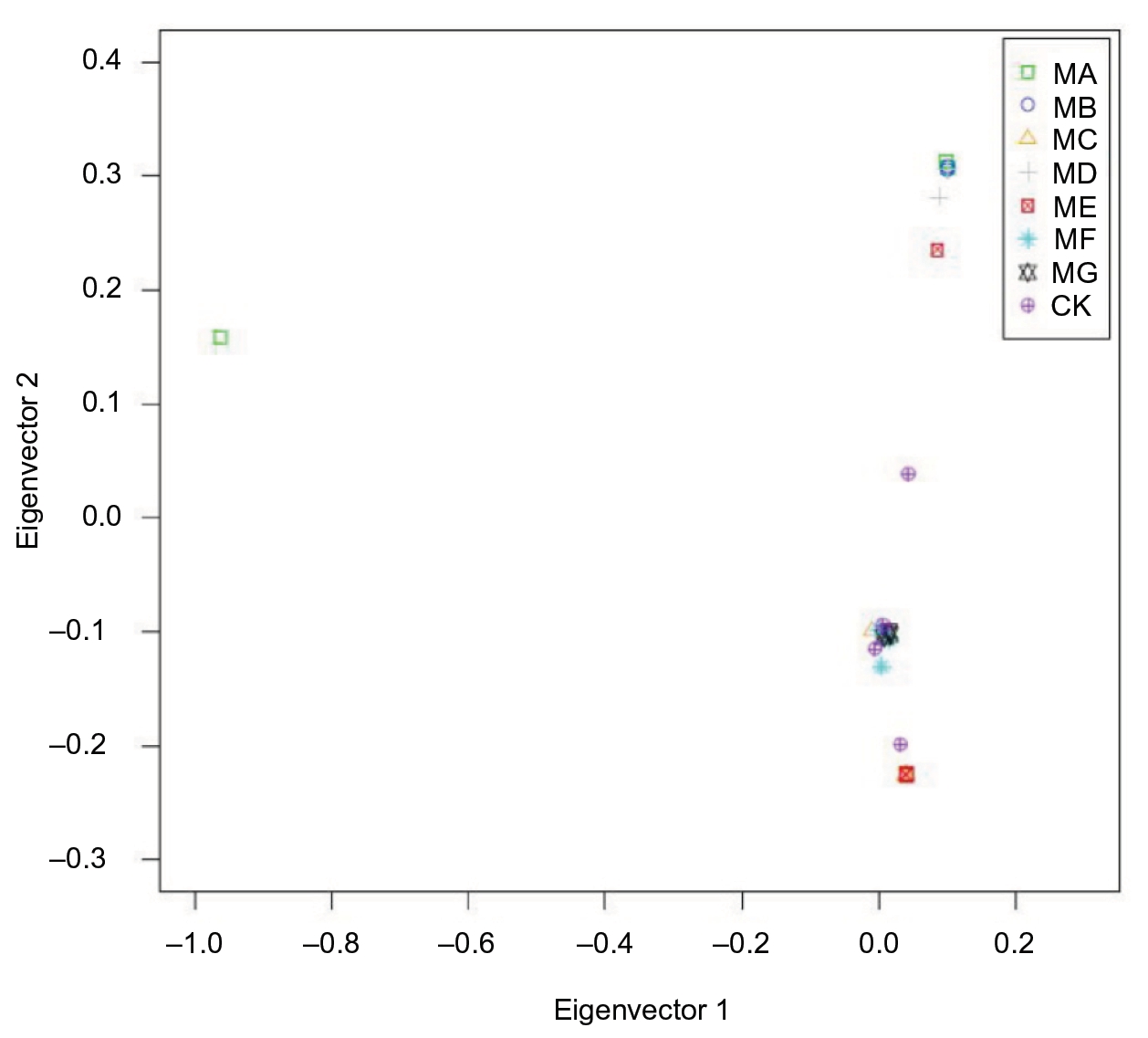
2. 肺炎链球菌分离株基因组水平分析
从SNP进化树图以及PCA分析图可以发现,携带不同抗生素抗性的分离株具有相似的遗传背景。这一结果也符合表1中的不同分离株的血清型。其中14-LC.ER1025分离菌表现出与其他分离菌不同的遗传关系,可能是基因组变异的结果。接下来,依靠SwissProt数据库,对这些特定区域的功能进行了注释。发现它们位于与毒力、蛋白质代谢和抗菌性相关的76个基因中。这表明这些序列具有相似性,并且水平遗传转移可能在它们的进化过程中发生。
3. 遗传变异与耐药性的关系
为了发现基因变异与耐药性的潜在联系,对敏感菌株和抗菌耐药菌株之间的Indels和SNP做了比较。其中,与PEN、CXM和CRO b-内酰胺相关的Indel数分别为77、237和139。此外,21个基因(例如,dpr、dagA、zmpB、dfr、axe1、dctA、pspC、sulA等)中的27个突变显示出高的突变率,并与CXM抗性密切相关。同时,鉴定出465个可能与大环内酯和林可酰胺抗性有关的突变,并且5个基因(lacE、ugd、pspC、pepB等)中的16个突变与该抗性有关。SXT耐药菌株有253个突变,其中14个突变与6个基因相关(dpr、pspC、dfr、dctA、sulA等)具有较高的突变率。与PEN(3480)、CXM(7707)和CRO(5214)β-内酰胺抗性相关的基因的SNP数目分别为1524、1494和1189个。7个基因(Sula、GDHA、HOM、XPT、BGLA、ReGR等)中的35个突变明确显示出高突变率,这些与CXM抗性密切相关。此外,发现14380个突变可能与大环内酯和林可酰胺抗性有关,trkH和PTS-EII基因与该抗性机制有关。与SXT抗性相关的突变数为8012个,涉及1524个基因,其中6个(ugd、dfr、sulB、dpr、dctA和pspC)与SXT抗性密切相关。

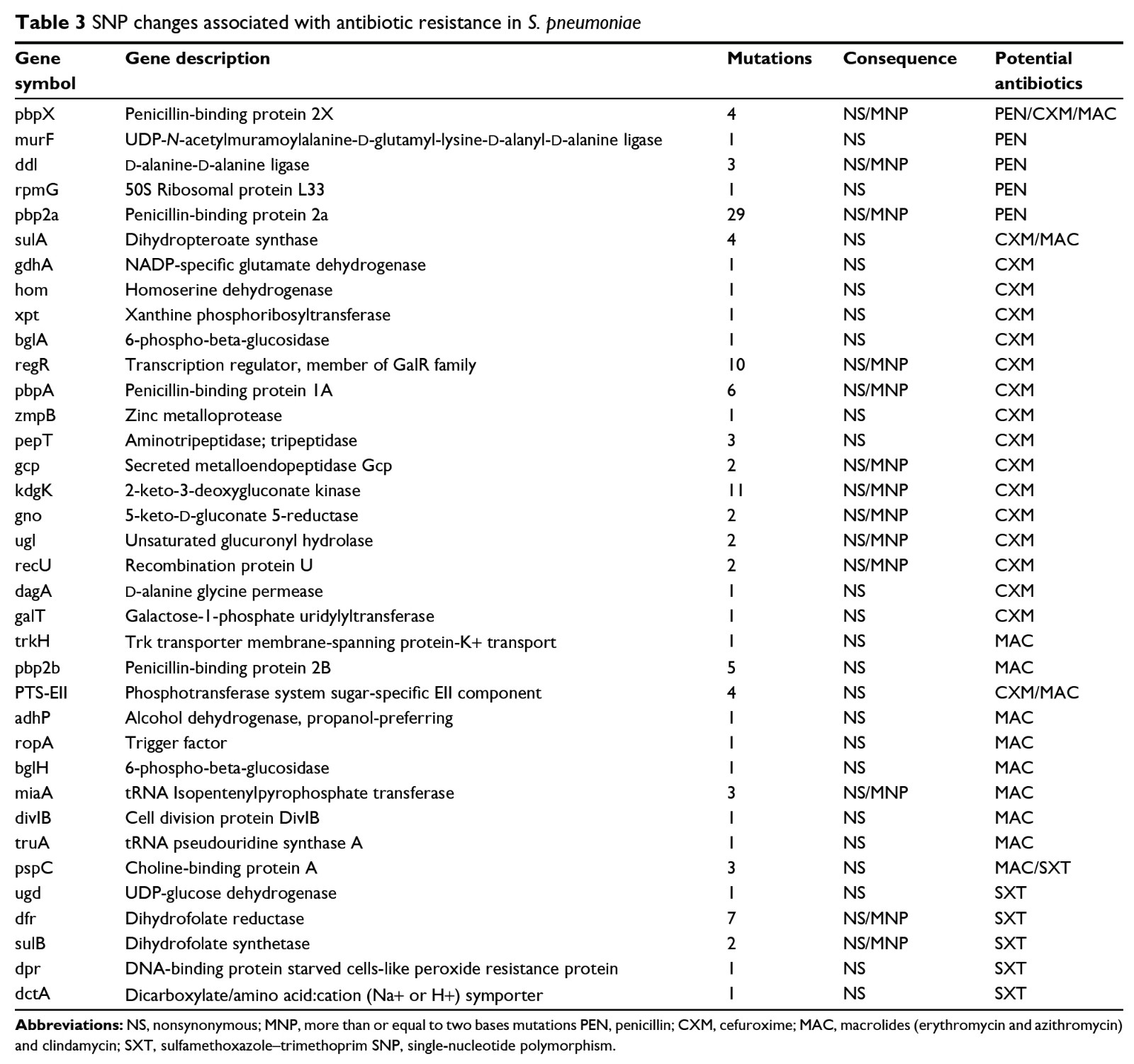
4. 发现与已知抗生素相关的基因
pbp2a、pbp2b、pbpX、murMN和ciaH基因的突变主要与PEN抗性相关,而pbp2a、pbp2b、pbp1b、murMN和ciaH基因的突变与CXM和CRO抗性有关。此外,发现大环内酯抗性菌株携带ermB和mefA基因,它们分别编码核糖体甲基化酶和药物外排蛋白。大多数大环内酯类耐药肺炎链球菌也对四环素耐药,并携带tetM基因。我们发现只有一个基因(rplD)的突变赋予大环内酯抗性,而dfr和sulA基因的突变与SXT抗性密切相关。

总结:
对25株肺炎链球菌的分析表明,具有相同抗生素耐药性的菌株具有不同的遗传背景,反之亦然。即我们观察到的遗传变化可能不依赖于耐药表型,选择、突变和重组的复合效应可以迫使生物体走向细菌进化。水平获取得具有耐药性的转座子可自发的促进生物体适应高耐药性的环境。这些可移动元件有助于肺炎链球菌临床上重要的耐药性转移,另外,不同株肺炎链球菌SNP和indels的突变频率较高,这些变化与一些抗生素耐药基因相关,同时也还需要进一步研究。
本研究的测序和数据分析工作由上海派森诺生物科技股份有限公司完成。
文章索引:
Pan F, Zhang H, Dong X, et al. Comparative genomic analysis of multidrug-resistant Streptococcus pneumoniaeisolates[J]. Infection & Drug Resistance, 2018,11:659-670.
原文链接:


