
2018-11-05

正文
最近,派森诺生物与南京大学合作,在《Bioresource Technology》(影响因子:5.807)发表论文,研究了5°C下添加氯化铵、尿素、L-丙氨酸和D-丙氨酸的SBRs性能,蛋白质、腐殖质、核酸、尿素和游离氨基酸等多种反应器出水特异性DON组成和细胞膜特征、相关基因和微生物群落结构,加深了我们对废水处理中DON起源和潜在威胁的认识。
研究背景
低温(<15°C)可能导致活性污泥(AS)的氨化、硝化和反硝化作用活性下降,这将导致出水DON增加,NH4+和NO2-富集,NO3-积累和一氧化二氮(N2O)流出等问题。许多工作都集中在硝化和反硝化作用,以获得可靠的替代品和在低温下成功运行。作为富营养化源和潜在形成的氮源消毒副产物(N-DBPs),出水DON问题已成为低温下污水处理厂(WWTP)关注的问题。氮源类型对AS群落结构,微生物组成和代谢产物有很大影响,导致出水可溶性微生物产物(SMPs)、游离氨基酸、NO3-浓度的变化。有趣的是,大多数的研究都是关于氯化铵或者室温下的真实废水的,只有少数研究关注到了添加成分。此外,目前尚缺少对于形成DON的氮源的研究。
研究方法
测序技术:Illumina MiSeq、Illumina Hiseq高通量测序平台
测序模式:微生物组细菌16S rRNA基因V1-V2区测序、宏基因组测序
生物反应器设计:5°C(培养箱:Sanyo Electric)下运行的添加氯化铵、尿素、L-丙氨酸和D-丙氨酸进料的四个SBRs(R1,R2,R3和R4),工作体积为2L。播种污泥(SS)取自中国南京市污水处理厂的曝气池(悬浮固体浓度约4400 mg / L)。根据参考文献选择5°C,由于纬度受限和季节变化(40-50°N),冬季的WWTPs存在大约30天的脱氮问题。SS之前没有在低温下驯化过。SBR的水力停留时间为12 h,进料时间、反应时间、沉降时间和空转时间分别为0.5 h,10 h,0.5 h和1 h。从反应器的中间孔排放出水,容积交换率为50%。反应器一般使用浓度为4000 mg/L(挥发性悬浮固体:2433-2979 mg/L)和氧浓度为7.6-8.0 mg/L的固定混合液体悬浮液(MLSS)运行。每周一次从反应器中抽出剩余的污泥。
图1:反应器设计
研究结果
1、生物反应器的性能
R2和R4的COD去除率较高(85.39%和86.82%),R3的COD去除率(81.56%)在稳定过程中低于其他反应器(COD去除率> 80%)。R1,R2,R3和R4的出水TN浓度在稳定期显示为13.49±0.43 mg / L,15.91±0.42 mg / L,14.79±0.53 mg / L和15.81±0.68 mg / L。值得注意的是,用尿素驯化的反应器在5°C时表现出更高的出水NH4 + -N浓度(8.59±1.36 mg / L)。相比之下,用氯化铵进行SBR进料的NH4+ -N去除率为89.15%-94.74%。该研究在没有特殊缺氧时间的情况下,反硝化效率有限,出水NO3--N仍然是氮的主要存在形式(R1-8.63±0.52 mg / L;R3-6.59±0.65 mg / L和R4-8.31± 1.02 mg / L),除了添加尿素的R2(2.34±0.81 mg / L)外。不同氮源的出水DON与TN去除率没有依赖关系(尿素>氨氯> L-丙氨酸> D-丙氨酸)。
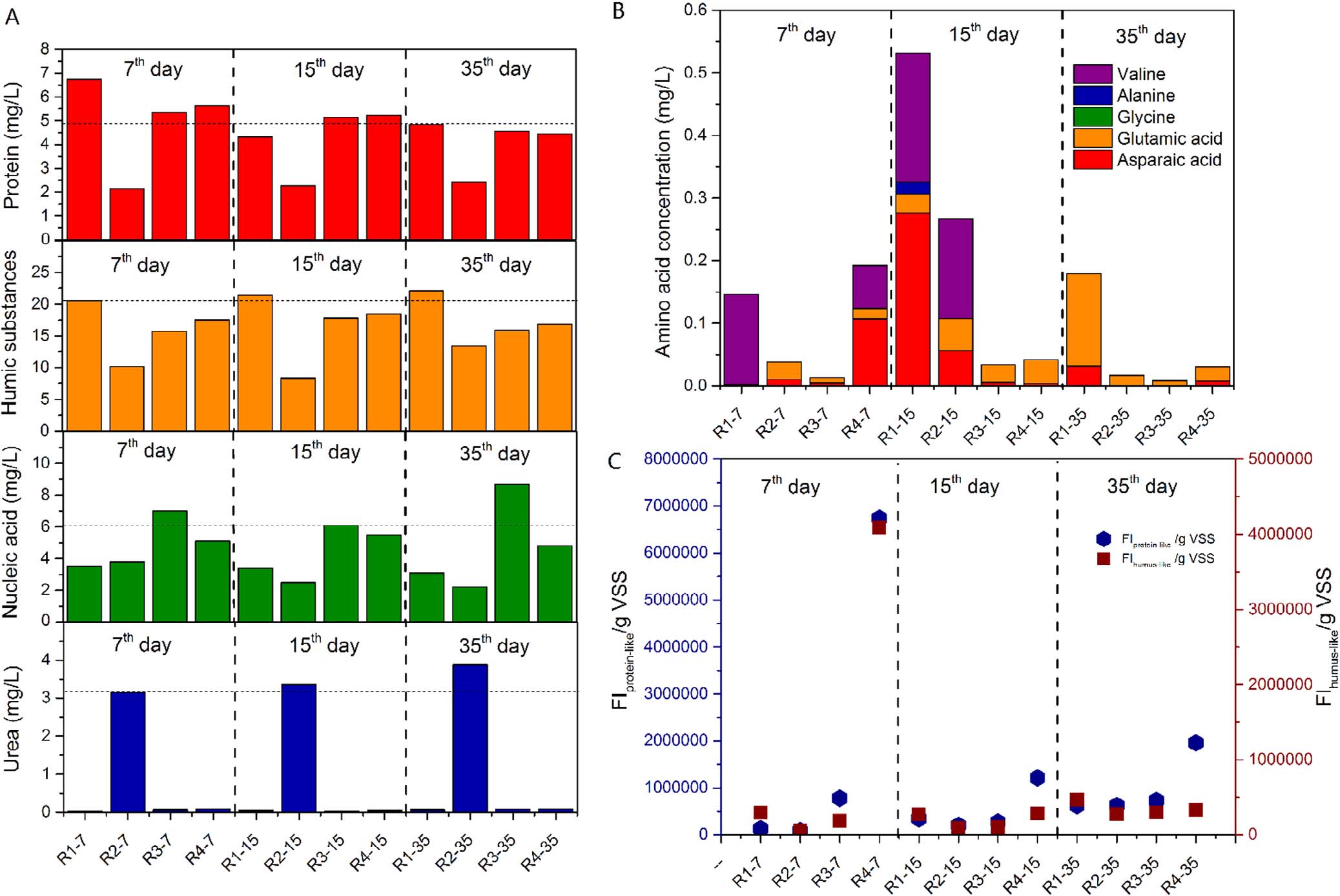
图2:35天内不同氮源的SBR在5°C时COD去除率(A),出水DON(B),TN(C),NH4+ -N(D),NO3- - N(E)和NO2- - N(F)
2、具体的DON组成
蛋白质类似物和腐殖质类似物是AS系统在低温下的主要成分。R4(D-丙氨酸)的流出蛋白质类似物组分的荧光强度(FI)显著高于其他反应器。此外,R1(氯化铵)的流出腐殖质类似物成分的荧光强度与其他成分相比更高。然而,R2的流出蛋白质类似物和腐殖质类似物成分显示出相对较低的大分子物质,尽管它们具有最高的DON。不同的氮源导致5°C时出水DON组分的多样性。R1、R3、R4的出水蛋白浓度(R1-35:4.85 mg/L, R3-35:4.55 mg/L,R4-35:4.44 mg/L)比较接近,而R2最小(R2-7:2.13 mg/L,R2-14:2.26 mg/L,R2-35:2.43 mg/L)。结果表明,氯化铵(R1-7:20.54 mg / L,R1-14:21.44 mg / L,R1-35:22.12 mg / L)有利于出水腐殖质的形成。
核酸、尿素和游离氨基酸也是DON的重要组成部分。L-丙氨酸加料反应器中出水核酸含量较高(R3-7:7.00 mg/L,R3-15:6.10 mg/L,R3-35:8.70 mg/L)。流出尿素仅在尿素加料反应器中出现积累(R2-7:3.14 mg/L,R2-15:3.35 mg/L,R2-35:3.88 mg/L),说明尿素不易降解(< 90%)。当氨化过程受到抑制时,丙氨酸易于在厌氧流出物中残留。然而,作者发现丙氨酸在5°C的好氧过程中很容易降解并消耗。此外,主要氨基酸是天冬氨酸和谷氨酸,它们都存在于所有氮源中。总之,主要的DON是由细胞代谢产物引起,但在5℃时R2中原始底物尿素的残留量不容忽视,占流出物DON的36%。
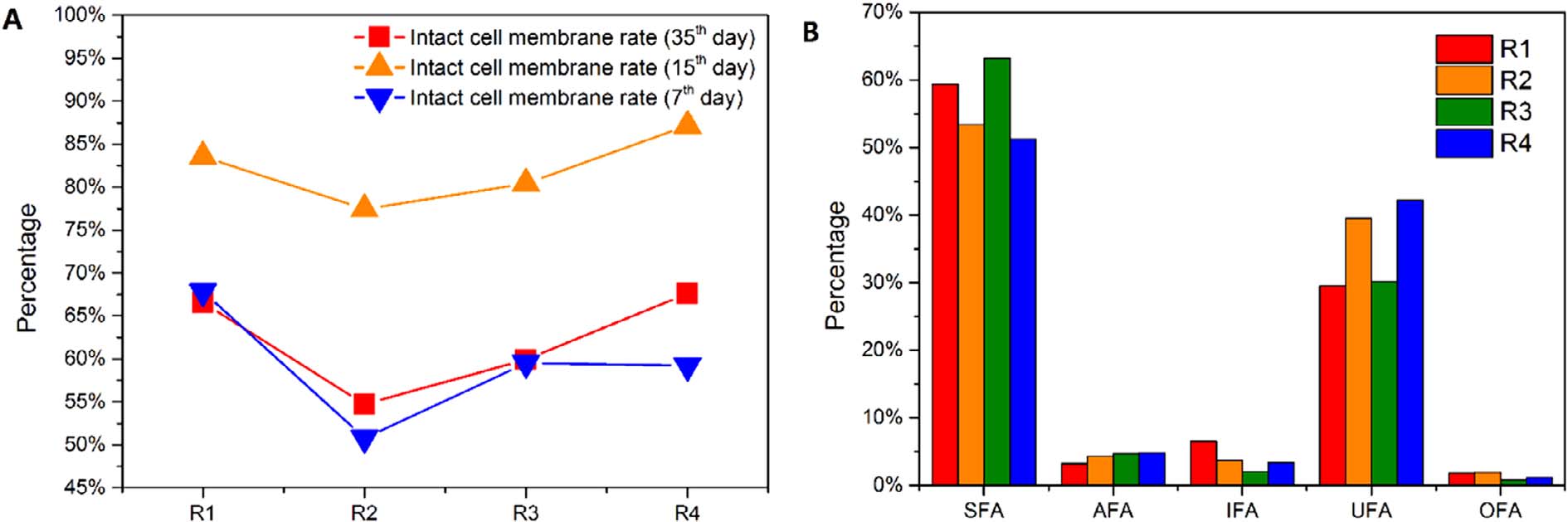
图3:在第7,15和35日采样期间每VSS(C)产生特定的DON组合,包括蛋白质、腐殖质、核酸、尿素(A)和几种氨基酸(B)FI蛋白质类似物和FI腐殖质类似物
3、细胞膜的完整性和组成
通过BacLight染色观察荧光显微镜照片和活/死细胞比显示的细胞膜活性。在相同的采样时间内,用尿素进料的R2,细胞膜完整率在第7天和第35天显示最低(R2-7:50.78%,R2-35:54.74%),这说明尿素对微生物有害。相反,L-丙氨酸或D-丙氨酸作为有机氮源,提供较少的细胞膜完整率,并在第7天(R3-7:59.51%,R4-7:59.20%)、第15天(R3-15: 80.42%, R4-15:87.13%)和第35天(R3-35:59.91%, R4-35:67.61%)差异较小。然而,在实验运行的35天期间,低细胞膜活力表明死细胞可能通过水解导致DON增加。
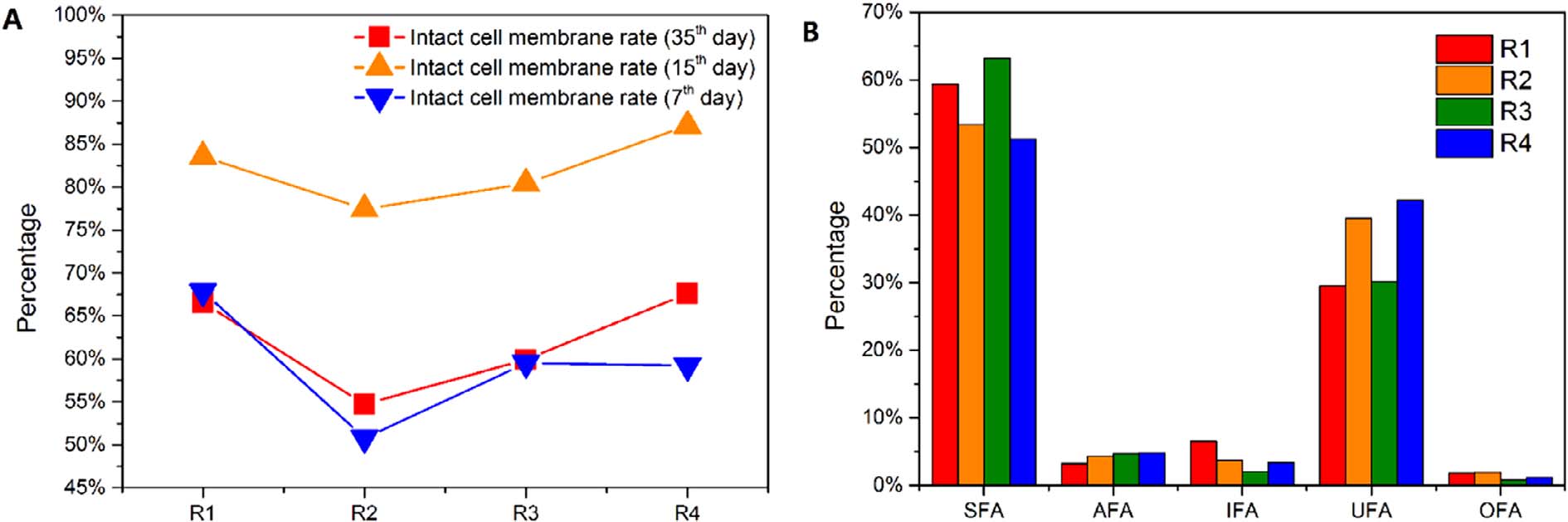
图4 在5℃下具有不同氮源的SBR中细胞膜的完整性(A)和组成(B)
4、宏基因组测序分析相关基因和预测酶的变化
在R1(氯化铵)和R3(L-丙氨酸)中,包括氮代谢在内的能量代谢功能基因丰度较高。与Thaumarchaea相关的氨氧化是硝化过程中的限速步骤,在作为好氧氨氧化标志的氨单加氧酶编码基因R1中富集amoA,有利于提高生物反应器的硝化能力。某些有助于该过程的蛋白质是nxrA(亚硝酸盐氧化还原酶基因),一种周质底物结合蛋白,以及反硝化细菌的narG,nirS,nirK和nosZ基因。氨氧化编码基因(EC 1.7.3.4)在低温下均富集于R1和R3。反硝化酶的编码基因被分配到异养群落,其比AS中的自养群落更具竞争优势。然而,随着硝化过程的发生,在有限的KO数据库中未检测到羟胺加氧酶的编码基因。此外,许多相关基因在低温下也能检测,例如napA,narG,narH。考虑到R1的最高TN去除率,R1中硝化和反硝化基因的丰度似乎促进TN去除,而不是减少DON含量。然而,DON含量可能与氨化中的相关基因和氨氮同化相协调。
与尿素降解酶相关的编码基因,如尿素酶(EC 3.5.1.5)、尿素羧化酶(EC 6.3.4.6)和嘌呤水解酶(EC 3.5.1.54),在尿素进料SBR中富集(R2)。AS中的Nitrospira可以将尿素分解成氨氮,这在5°C的所有反应器中都检测到。此外,天冬氨酸和氨提供氨基,导致尿素循环中尿素的形成,这也解释了出水尿素的产生。L-丙氨酸在低温下比D-丙氨酸更具有稳定性。进料L-丙氨酸的氨化基因数高于进料D-丙氨酸的氨化基因数,说明在L-丙氨酸的利用中需要更多的微生物参与。
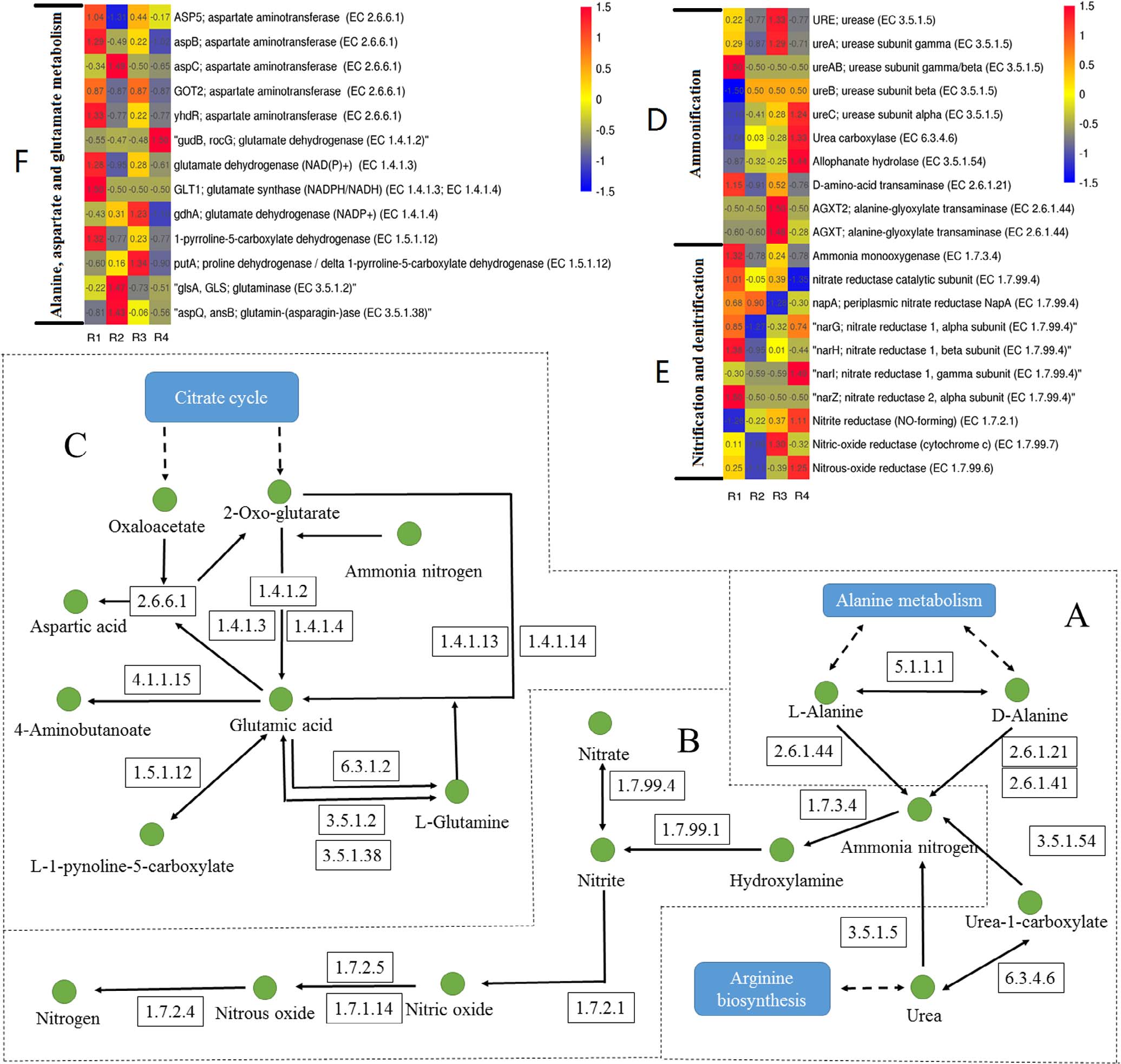
图5 第35天5℃下4个SBRs中,宏基因组KEGG Orthology的氮代谢和丙氨酸、天冬氨酸和谷氨酸代谢的相对基因分布(E值<10-5)
值得注意的是,低温会导致天冬氨酸和谷氨酸的积累,其代谢直接位于氮代谢的上游。在第35天,只有天冬氨酸和谷氨酸都存在于四个反应器的流出物中,这可以在低温下增加蛋白质的稳定性。微生物调节能量代谢和氨基酸代谢,包括谷氨酸,天冬氨酸和丙氨酸,以适应盐度环境。天冬氨酸和谷氨酸积累可能是由低温下的微生物反馈引起的。NH4 +同化(EC 1.4.1.2)是细菌的主要固定化方式。谷氨酸是氨同化作用的主要产物,是各种生物合成反应中的氮供体,可转化为蛋白质合成的其他标准氨基酸、尿素核酸等。在死亡的AS微生物水解后,腐殖质是DON的重要来源。
5、微生物群落组成的变化
宏基因组测序结果中,细菌占99.07%,而Proteobacteria在低温下是主要的门。而16S rRNA基因测序结果则提供了门和属水平上的细菌多样性信息。通过16S rRNA基因测序表明,细菌种类减少到不到一半,这表明超过一半的细菌属不能适应5°C。从聚类分析的结果来看,SS最相似的细菌种群结构是第7天R1(氯化铵)中的样品,这也说明氯化铵在5°C的短时间内保持了微生物的多样性。
在第一阶段(第7天),16S rRNA基因扩增子测序的结果显示Proteobacteria在低温下是主要的门,在R1(氯化铵)中占69.63%,在R2(尿素)中占85.28%,87.94%在R3(L-丙氨酸)中和在R4中的94.84%(D-丙氨酸)。Proteobacteria是5°C时AS中最重要和最主要的细菌门,其他优势门是Chlorobi,Acidobacteria,Actinobacteria,Chloroflexi,Nitrospira和Bacteroidetes,它们也是常见的,低温下在水生生态系统中检测到的门。Pseudomonas占最高丰度(R1-38.13%,R2-37.66%,R3-61.75%和R4-66.06%),其次是Janthinobacterium(R1-11.74%,R2-35.98%,R3-20.69%和R4-24.46%)和Flavobacterium(R1-6.38%,R2-11.88%,R3-1.83%和R4-1.40%),这些常见于寒冷条件下的水生生态系统。
在第二阶段(第15天),Proteobacteria和Bacteroidetes是5°C好氧反硝化中的主要门,其他细菌门占不超过1%。在属水平上,除了R1之外,主要细菌属没有明显变化。R1中的属(> 0.01%)比其他反应器减少的更慢。然而,Pseudomonas、Janthinobacterium和Flavobacterium在所有SBRs中保持较高的丰度,就像在第7天一样。
在第35天的第三阶段,AS中的微生物群落结构与第15天基本保持稳定。在门水平上,Proteobacteria是R1(84.81%)、R2(87.60%)、R3(92.76%)和R4(92.35%)的优势门。Bacteroidetes位居第二(R1-35:12.87%,R2-35:11.86%,R3-35:3.65%,R4-35:4.68%),Actinobacteria居第三。
在以有机氮为进料的SBR中,Proteobacteria和Actinobacteria较为丰富。在属水平上,Pseudomonas(R1-41.37%,R2-39.85%,R3-62.33%,R4-62.33%)、Janthinobacterium (R1-29.87%,R2-36.75%,R3-27.43%,R4-24.62%)、Flavobacterium(R1-12.35%, R2-11.65%, R3-2.90%, R4-4.13%)均丰富(> 3%)。然而,在5℃下,Pseudomonas,Flavobacterium和Arthrobacter在AS中也显示出更高的比例。
采用冗余分析(RDA)来评估第35天出水水质与细菌种群结构(> 1‰)之间的关系。RDA1和RDA2分别解释了总方差的95.6%和3.9%。从4个SBRs的位置看,微生物群落结构明显地根据氮源分为4组。特别是,Simplicispira(R2-0.04%)在用尿素(R2)进料的反应器中占据显著更高的丰度,其与蛋白质呈负相关(Person test,p <0.05)。相反,Nitrospira可有效降解尿素。出水尿素(Person test,p <0.05)与DON含量呈显著正相关。L-丙氨酸和D-丙氨酸的微生物种群存在明显差异。更多的微生物倾向于使用D-丙氨酸,而不是L-丙氨酸,这可能导致R3的出水COD性能较低。此外,L-丙氨酸进料的Dyadobacter (R3-0.05%)丰度更高,Haliscomenobacter(R4-0.18%),Ignavibacterium(R4-0.15%),Thauera(R4-0.05%),Acinetobacter(2.87%),Nitrospira(R4-0.18%),Gp4(R4-0.04%)和Gp17(0.04%)在进料D-丙氨酸的反应器中占更高的百分比。此外,流出物中的腐殖质浓度可能与Caldilinea呈正相关(Person test,p <0.05)。值得注意的是,在氨氮上游的氨基酸和尿素的代谢与DON呈正相关(Person test,p> 0.05),而不是蛋白质,腐殖质和其他大分子有机氮。这意味着通过凝固去除DON可能是有限的。以上结果表明,氮源类型引起了明显的微生物群落差异,从而影响了低温下的DON组分。
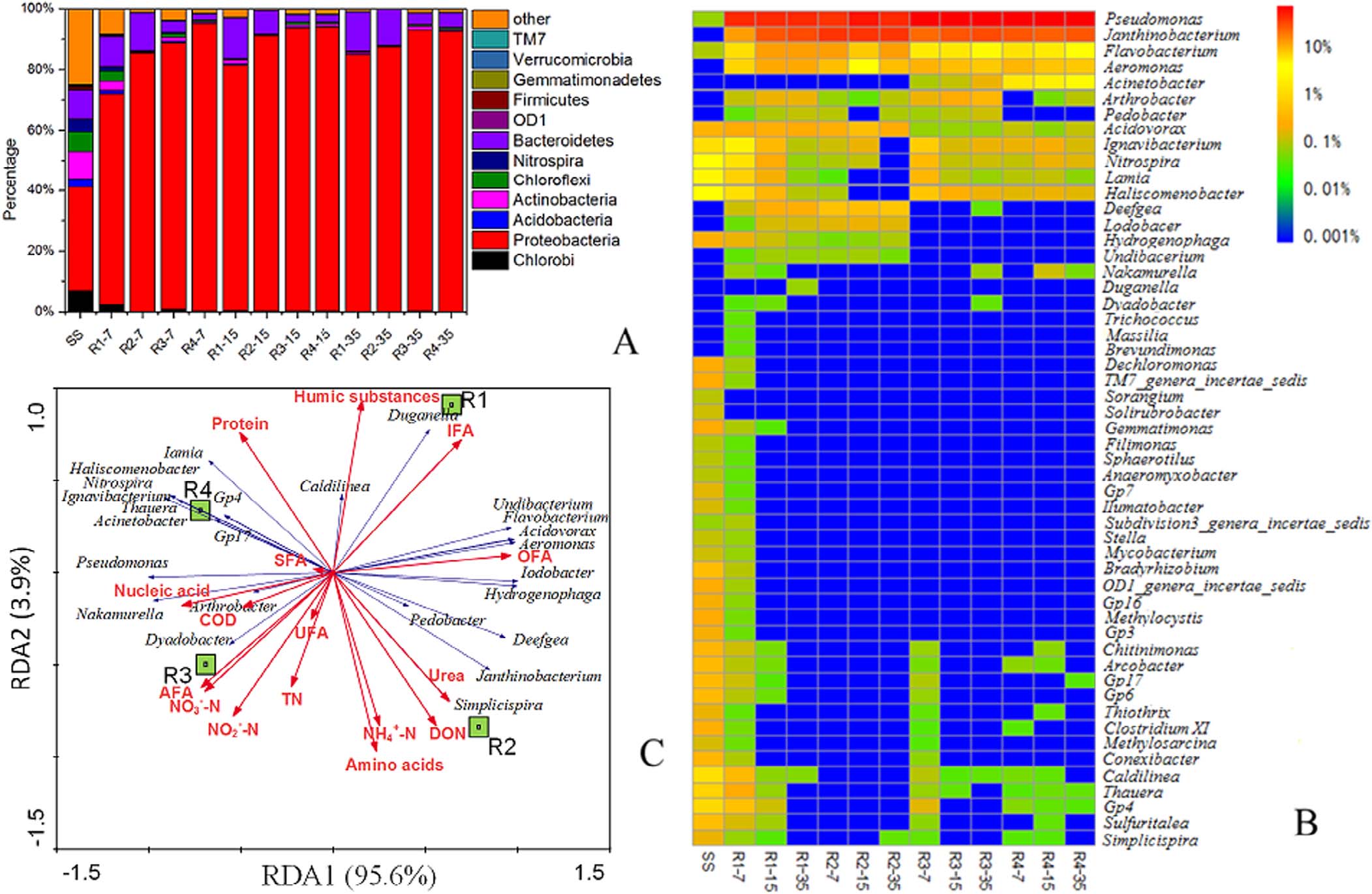
图6 冗余分析(C)和不同氮源类型SBRs 5℃下门和属水平细菌群落
总结
本研究揭示了特定的DON组成取决于氮源,并且DON含量在5°C时是显著的。根据成分分析,DON来自细胞代谢产物,而不是原始底物。所有氮源的出水中都存在天冬氨酸和谷氨酸。推测NH4+同化是DON形成的主要N-固定方式。RDA显示没有单一细菌属可以有效降解DON,而Simplicispira在尿素进料中比例更高。作为氨基酸代谢产物的出水尿素与DON含量具有强烈的正相关性。
文章索引:
Su He, Lili Ding, Kan Li, Haidong Hu, Lin Ye, Hongqiang Ren (2018). Comparative study of activated sludge with different individual nitrogen sources at a low temperature: effluent dissolved organic nitrogen compositions, metagenomic and microbial community. Bioresource Technology 247(2018) 915-923.
原文链接: https://doi.org/10.1016/j.biortech.2017.09.026