2018-11-14

文案 | 转录调控事业部
研究简介
假单胞菌属是一种具有复杂基因组结构的,广泛分布且具有生态重要性的海洋谱系菌。通过研究来自北冰洋的冷适应型假单胞菌BSW20308的基因组到其对温度变化的转录组学反应,发现了该菌两条染色体的进化及其在适应温度变化反应中的不同作用。sRNome (the complete set of small RNAs in the organism)的广泛参与,特别是新型sRNA与tmRNA和6S RNA的共同作用,也被预测在热应激下发挥重要作用。
这项工作证明了多分体基因组的进化以及sRNome在该海洋属的环境适应中的重要性。它更新了人们之前对假单胞菌的理解,并在转录组水平,揭示了环境对海洋微生物的影响。
研究方法
基因组:测序平台PacBio RS II
转录组:将P. fuliginea BSW20308在4°C、15°C和32°C,三个温度下一式三份, 9个样本,培养至早期稳定期,提取RNA,Illumina NextSeq 500测序
研究结果
▶ P. fuliginea BSW20308基因组的一般特征
研究结果显示P.fuliginea BSW20308基因组包括大染色体(chr1,3,924,123bp,GC含量为39.1%)和小染色体(chr2,906,103bp,GC含量为38.7%)。在7个rRNA操纵子中组织了总共23个rRNA基因,其中6个在chr1上,1个在chr2上。 oriC区域(oriC1和oriC2)在序列上显著不同。 在chr1和chr2上分别检测到总共12个和2个基因组岛(GIs)。分别在chr1和chr2上预测了5个和2个推定的次级代谢物生物合成基因簇(SMGCs)。 SMGC可能产生细菌素,芳基多烯,非核糖体肽和其他类型的天然产品。 在chr1上检测到41.1kb的完整原噬菌体和8.1kb的不完整原噬菌体。在chr2上预测了6.5kb的不完整原噬菌体。在chr1和chr2上分别鉴定出四个和一个有规律的间隔短回文重复序列(CRISPR)。
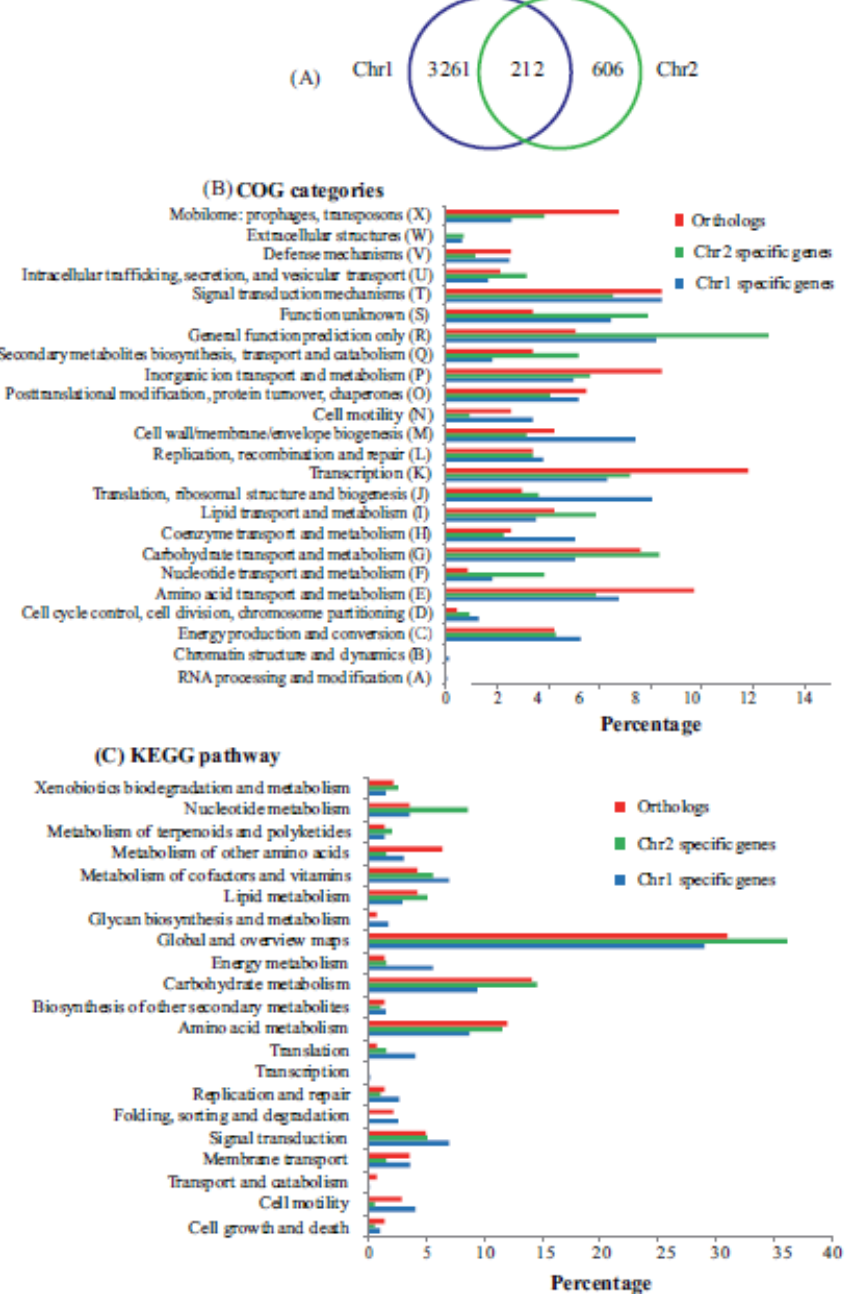
▶ 两条染色体的注释和比较
根据RBH分析,两条染色体共有212个直系同源物,分别占chr1和chr2基因的6%和26%(图A)。直系同源物中最丰富的直系同源簇(COG)是K类(转录),其比例高于染色体特异性基因(图B)。chr1和chr2特异性基因的最丰富的COG类别分别是功能组T(信号转导机制)和R(仅一般功能预测)(图1B)。与chr1相比,chr2特异性基因和直向同源物:R(仅一般功能预测),S(功能未知),Q(次级代谢产物生物合成,转运和分解代谢),U(细胞内运输,分泌和囊泡运输),I(脂质转运和新陈代谢)和F(核苷酸转运和代谢)(图B)。与chr2相比,更高比例的chr1特异性基因属于COG类别M(细胞壁/膜/包膜生物发生),J(翻译,核糖体结构和生物发生)和H(辅酶转运和代谢)(图B)。
KEGG途径分配显示chr1,chr2特异性基因和直系同源物之间存在一些差异。 例如,chr1特异性基因在能量代谢和翻译,以及折叠,分选和降解通路中占最高百分比(图C)。在异生素生物降解和代谢,萜类化合物和聚酮化合物的代谢以及核苷酸代谢的途径中,chr2特异性基因的比例高于chr1(图C)。 chr2特异性基因中不存在一些代谢途径,包括聚糖生物合成和代谢,转录,折叠,分选和降解等(图C)。
▶ 第二条染色体的必要性
P. fuliginea BSW20308的第二条染色体除了rRNA基因外还具有必需基因。 例如,参与细胞分裂的minCDE,编码组氨酰-tRNA合成酶的hisS,类异戊二烯生物合成的脱氧木酮糖途径中的gcpE,以及编码DNA复制末端位点结合蛋白的tus,对于正常生长和存活是必需的,并且仅在chr2上检测到。组氨酸生物合成基因簇含有8个紧密连锁的基因hisIFAHBCDG,仅存在于chr2上。
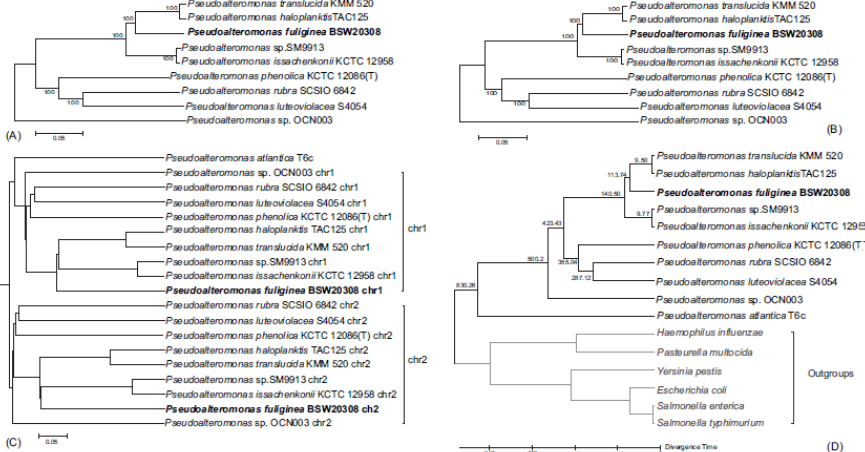
▶ 系统发育和时间尺度分析
本文选择了9个代表性的具有两条染色体的假单胞菌属完整基因组来构建具有P. fuliginea BSW20308的系统发育树。两条染色体树在9个物种中显示出相同的系统发育关系,分枝长度略有不同(图A和B)。基于全基因组的系统发育树也支持这些物种的相同系统发育关系(图C)。它清楚地显示了第一和第二染色体的单独聚类(图C)。根据时间估计(图D),P.atlantica T6c在大约8.3亿年(Ma)之前首先与其他假单胞菌分开。假单胞菌属OCN003是剩余的假单胞菌属的第二种不同物种。
有趣的是,正如估计的那样,北极株P. fuliginea BSW20308和南极株P. haloplanktis TAC125在大约113 Ma之前的分歧之前共享一个假定的祖先。
▶ RNA-seq 数据聚类
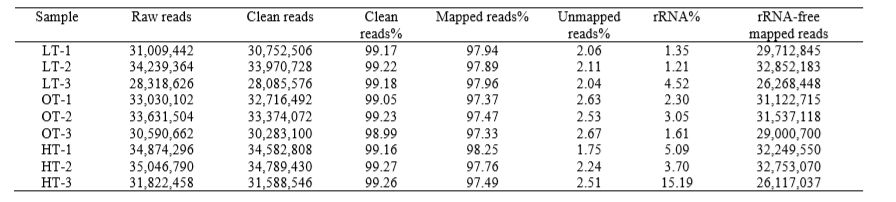
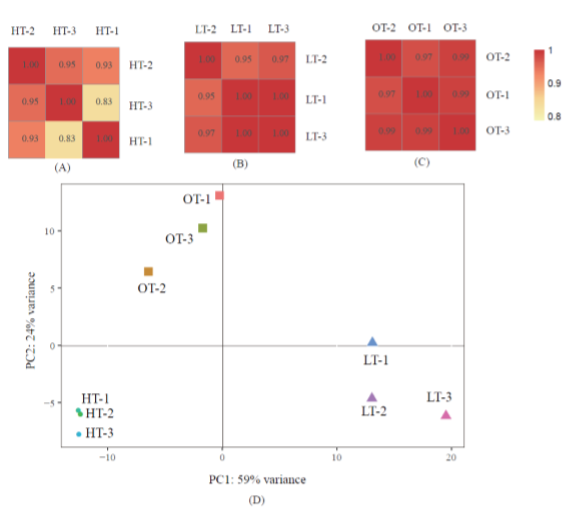
转录组研究了在4°C、15°C和32°C生长的P.Fuligina BSW20308,这三种温度分别代表显示显著增长的最低(LT),最佳(OT)和最大温度(HT)。测序结果每个样本产生超过4G的数据量。此外PCA分析和Pearson相关分析一起表明,相同温度处理中的生物学重复相似性很高,可以组合以代表在相同条件下的总体基因表达。
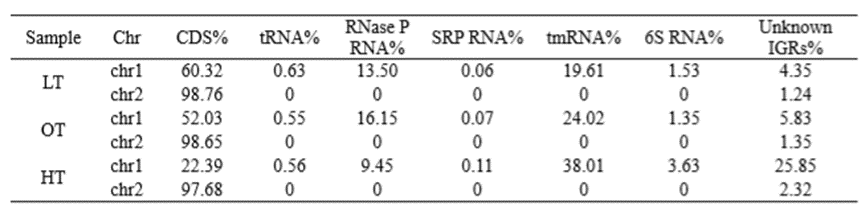
▶ 将RNA-seq数据比对到基因组
超过97%的clean reads被比对到基因组,映射到chr1上CDS的reads的平均百分比随着温度升高而降低,从LT的平均60.32%到HT的22.39%(表1)。除了CDS之外,非编码RNA基因和未知的基因间区域(IGR)在chr1上以不同程度转录(表1)。在HT处检测到高百分比的tmRNA(38.01%)和未知的IGR(25.85%),甚至高于在该条件下转录的CDS(22.39%)(表1)。 RNase P RNA在OT时具有最高的平均百分比,在HT时具有最低的平均百分比。在三种温度处理下检测到相似水平的tRNA基因转录(约0.6%)。相反,在9个转录组中仅在chr2上检测到CDS(超过97%)和未知的IGR(约1-2%)(表1)。
▶ IGRs内新转录本的预测
在高温处理下,未知的IGR在chr1上以相对高的水平转录。映射到未知IGR的最丰富reads与位于chr1上编码GNAT家族乙酰转移酶(D172_RS06075)和编码假设蛋白(D172_RS06080)的基因之间的序列相同,占每个未知IGR中所有未知IGR的99%以上。预计该区域含有316nt的新型sRNA,在此命名为Pf1 sRNA,其在Rfam数据库中没有相似的序列。除了这种新的sRNA之外,还在未知的IGR中预测了另外25种新的转录本,包括另外14种新的sRNA候选物和11种非RNA编码的新转录本。其中,一个新的sRNA(Pf15 sRNA)和两个新的转录本(PfNT1和PfNT2)位于chr2上,而其余的预测转录本都位于chr1上。
由于在HT下,Pf1 sRNA是检测到的最主要的转录物,因此对Pf1 sRNA和基因表达在转录组中的潜在相关性进行研究。使用Pearson r相关性,发现多达644个基因的转录与Pf1 sRNA显著相关(P值<0.01)。其中,12个基因与Pf1 sRNA呈正相关,其余632个基因均与Pf1 sRNA呈负相关。
根据eggNOG分类,大量基因未被分配到COG功能类别中。 632个负相关基因主要分为COG类别S(功能未知),E(氨基酸转运和代谢),C(能量产生和转化),R(仅一般功能预测),J(翻译,核糖体结构和生物发生),U(细胞内运输和分泌)和H(辅酶转运和代谢)。
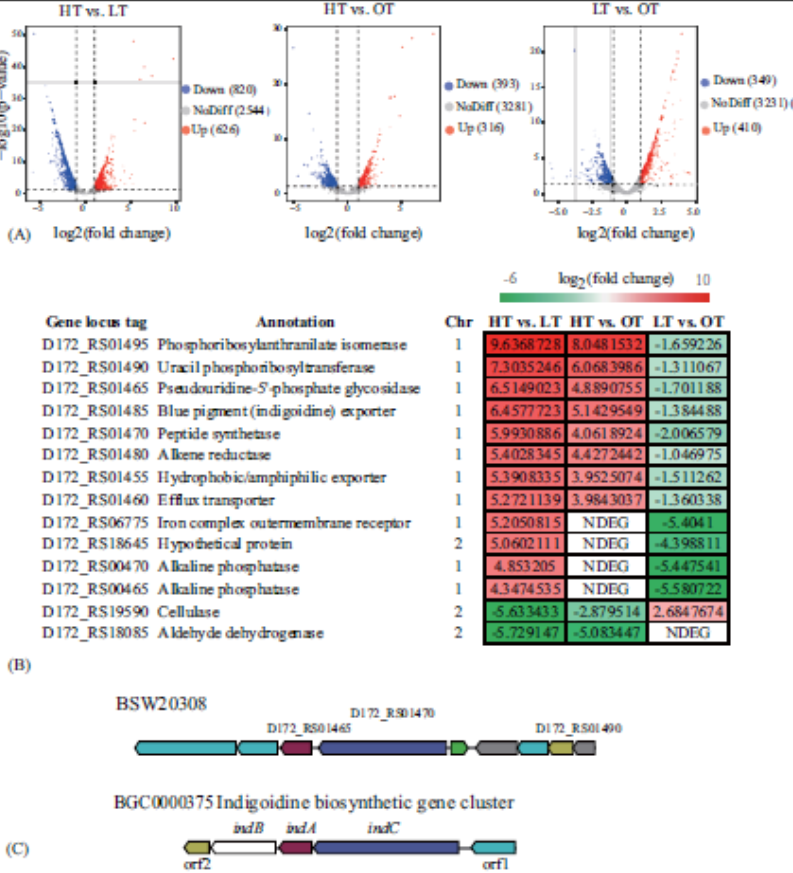
▶ 与温度和染色体相关的差异表达
在HT与LT,HT与OT和LT与OT的成对比较中,在基因组中检测到总共1446,709和759个差异表达的基因(DEG)。 成对比较中DEG的火山图显示在图A中。 在HT处的DEGs比在LT和OT处的表达下调得更多,并且在LT处的DEG上调比在OT时更多。构建热图以显示在所有成对比较中具有log2(倍数变化)超过5的DEGs(图B)。在热图中显示的14个基因中,只有3个基因位于chr2上。在上调基因中,8个位于chr1附近并且在相同方向上定向(图C)。
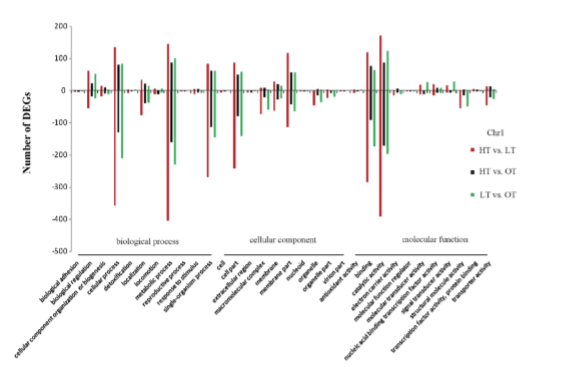
根据eggNOG,GO和KEGG分类对来自两条染色体的DEG进行分析。 在HT与LT的比较中,大多数eggNOG类别被下调,并且在chr1上LT与OT的比较中更多类别被上调。在chr2上,大多数DEG在HT处被上调。 与eggNOG分析类似,大多数GO类别中的更多DEG在chr1处的HT处被下调,并且在chr2处被上调。 KEGG途径表现出与GO和eggNOG分析类似的模式,然而两种染色体都具有在不同KEGG途径中富集的DEG。例如,大多数DEG位于chr1上的翻译途径和chr2上的膜转运途径中。
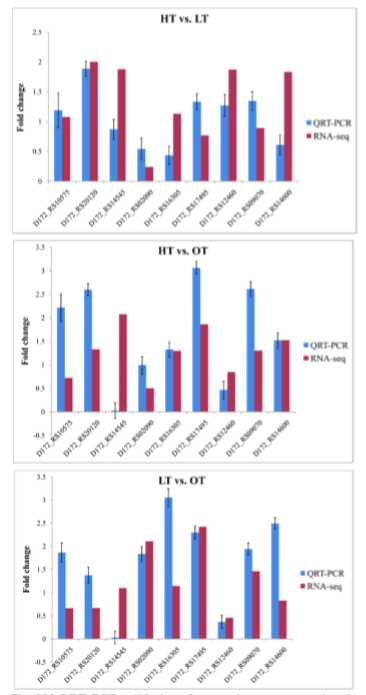
▶ QPCR验证
对8个随机选择的基因成功进行qRT-PCR。对于三个温度成对比较,qRT-PCR结果显示与RNA-seq数据一致,因此来自qRT-PCR的实验数据证实了RNA-seq的转录组分析数据的可靠性。