
2020-08-13

期刊:《Journal of Virology》
影响因子:4.501
最近,派森诺与上海海洋大学合作,在《Journal of Virology》发表论文,利用宏基因组测序,探究了上海滴水湖中新型噬病毒体和藻类大病毒的多样性特点,并发现了以单细胞绿藻为真核宿主的新型Cell-Virus-Virophage(C-V-v)三相互作系统。
大伙可能都听说过噬菌体,但是噬病毒体是什么呢?生命形式总是如此多样而又出乎意料,今天就由小编带领大家,走进噬病毒体的奇妙世界吧!
研究背景
噬病毒体(Virophage)是利用原生生物巨病毒(Giant virus)在真核宿主内形成的病毒工厂(Viral factory)中进行增殖的一类小型双链DNA病毒,它可使巨病毒的正常增殖受到破坏,导致其产生侵染能力下降的畸形病毒颗粒,像是受到了侵染,噬病毒体因此而得名。噬病毒体可伴随巨病毒一同侵染宿主细胞,而由噬病毒体、巨病毒和真核宿主细胞三者所构成的互作系统,称之为C-V-v系统。目前,仅有少数噬病毒体如Sputnik,Mavirus,Zamilon等及其C-V-v系统已被发现和初步探究,而广泛分布于水体环境中的噬病毒体及其C-V-v互作系统仍有待研究。
为进一步探究此类病毒与病毒之间,及病毒与真核宿主之间特殊的互作系统,本研究利用二代高通量测序技术,对滴水湖表层水体中的噬病毒体、藻类大病毒及它们之间的互作关系进行了研究。由于目前发现的噬病毒体都属于DNA病毒,因此,我们既可以用宏病毒组的方法,富集病毒颗粒后进行测序研究;也可以直接采用宏基因组的方法,通过加大测序深度,从海量测序数据中提取病毒基因组的相关信息,进行深入研究。这样,就能帮助我们更进一步地探索和理解神奇的噬病毒体啦~
研究内容
① 噬病毒体基因组挖掘与分析;
② 藻类大病毒基因组挖掘与分析;
③ 噬病毒体与藻类大病毒基因关联分析;
④ 噬病毒体与藻类大病毒基因组特征分析;
⑤ 藻类大病毒中CRISPR-Cas-like系统分析;
研究目的
探究滴水湖中由噬病毒体、藻类大病毒、单细胞真核绿藻所构成的新型C-V-v三相互作系统。
材料与方法
测序技术:Illumina MiSeq高通量测序平台
测序模式:宏基因组测序
实验对象:滴水湖表层湖水
采样方式:通过已灭菌的5 L采水器采集表层水样(<1 m),于20 L采样桶中快速混合润洗后,弃去,再迅速装满。经现场测定环境因子后立刻转移至实验室冷库,留待进一步处理。
实验设计
① 采集滴水湖表层湖水样品;
② 提取水样微生物总基因组;
③ 利用Illumina MiSeq高通量测序平台进行测序和生物信息学分析;
④ 噬病毒体和藻类大病毒的基因组组装与特征分析;
⑤ 噬病毒体的潜在巨/大病毒宿主分析。
结果与讨论
01、滴水湖中新型噬病毒体的发现及其基因组分析
本研究基于负压过滤水体样品至0.22 μm孔径滤膜(已灭菌)的方法,对滴水湖中表层水进行了微生物的富集,总DNA的提取、验证,并采用Illumina MiSeq高通量测序平台完成了双端测序工作(4个run的2×250 bp的Paired-end即PE文库和2个run的2×250 bp的Mate-paired即MP文库)。原始下机数据共计49.68Gb,约1亿条reads。
如图1所示,通过序列拼接,本研究从滴水湖宏基因组数据集中共获得7株新型噬病毒体的全基因组序列,并将其分别命名为Dishui Lake Virophage 2/3/4/5/6/7/8(简称:DSLV2/3/4/5/6/7/8),其序列长度分别为31,238、31,512、30,873、26,593、28,714、29,961和26,605 bp。滴水湖噬病毒体(DSLVs)与已知噬病毒体基因关联分析显示,DSLVs与已知来源于淡水的噬病毒体共享更多的同源基因,而不与已知分离出来的噬病毒体(Sputnik家族、ALM、Mavirus、CpV-PLVs等)共享任何基因,表明DSLVs与来源于淡水的噬病毒体亲缘关系较近(图2A)。如图2B所示,系统发育分析进一步表明,DSLVs与来源于淡水的噬病毒体进化关系较近,而与已知分离出来的噬病毒体进化关系较远,提示它们可能拥有不同的病毒宿主和真核细胞宿主。
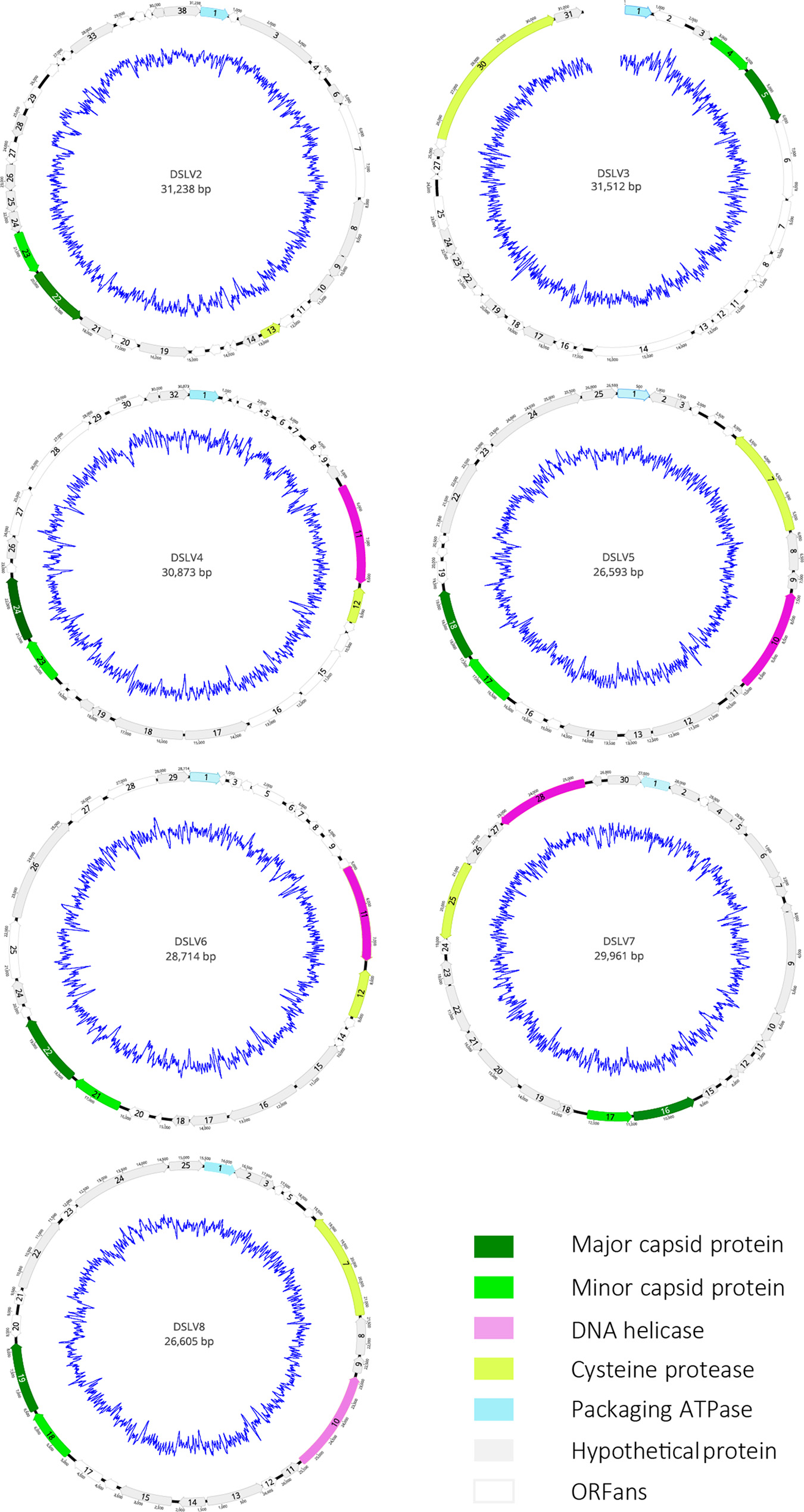
图1 噬病毒体DSLV2/3/4/5/6/7/8基因组图谱
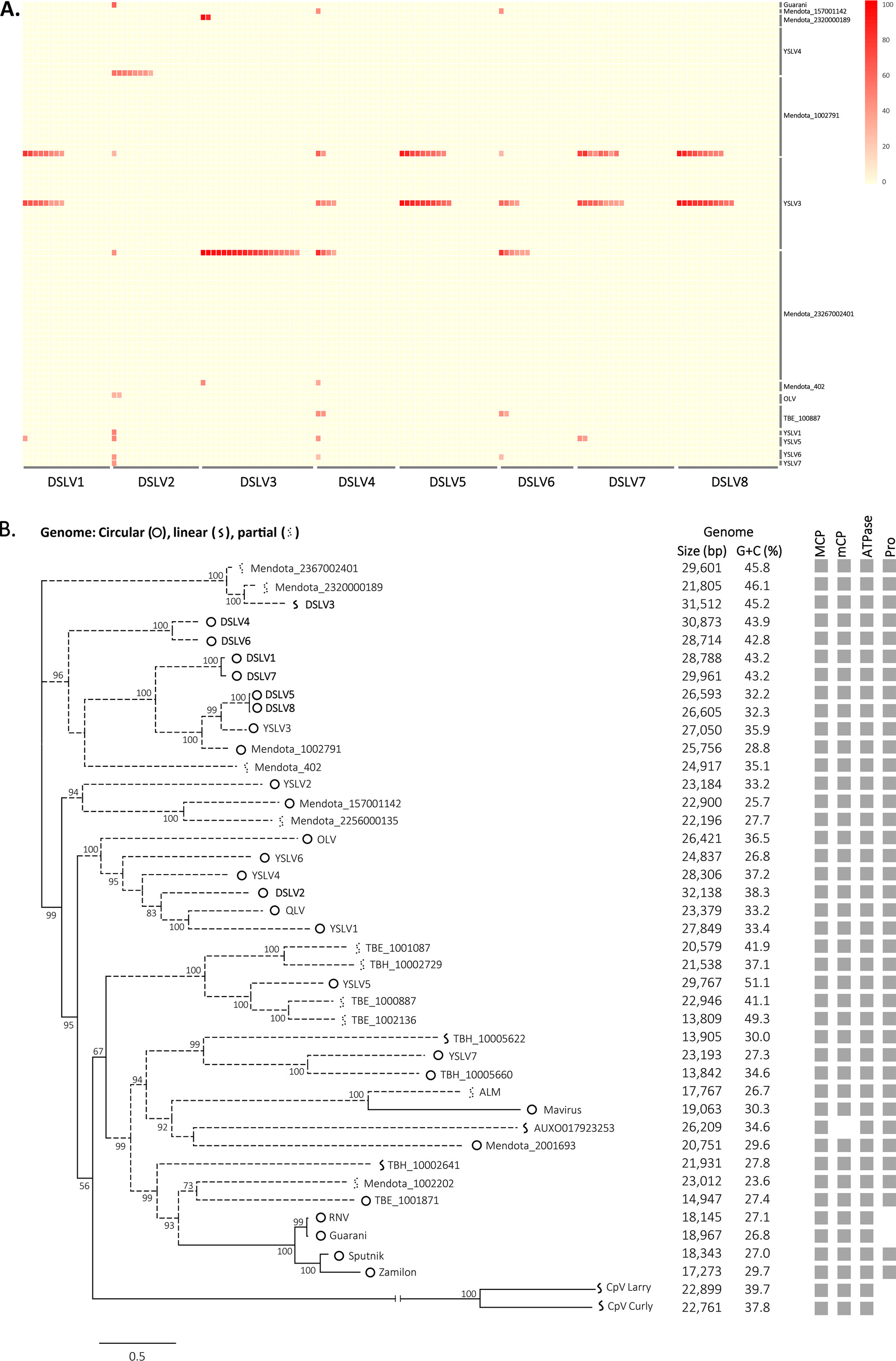
图2 滴水湖噬病毒体与已知噬病毒体基因关联分析(A)及系统发育分析(B)
02、滴水湖中新型藻类大病毒的发现及其基因组分析
通过相关序列的调取和基因组的组装、注释工作,作者从宏基因组数据集中共获得了四条完整的藻类大病毒基因组,大小为190,426,194,169,193,666和392,531 bp,并分别将它们命名为Dishui Lake phycodnavirus 2/3/4(简称:DSLPV2/3/4)以及Dishui Lake large alga virus 1(简称:DSLLAV1),如图3、图4A所示。其中DSLPV2/3为线性基因组,分别编码有247和236个ORFs;而DSLPV4与DSLLAV1为环状基因组,分别编码有220和366个ORFs。通过基因注释发现,DSLPV2/3/4中分别有164、190、149个ORFs与绿藻大病毒科Phycodnaviridae中Prasinavirus所编码的基因具有同源性,表明它们与Prasinavirus具有较密切的亲缘关系,而在DSLLAV1中仅有104个ORFs与病毒编码的基因具有同源性,其中有接近半数(44个)的ORFs与Mimiviridae第三分支(GroupIII)中的藻类大病毒具有同源性,且与其中的绿藻大病毒Tetraselmis virus 1(TetV-1)共享最多的同源基因(16个,图4B),表明DSLLAV1与藻类大病毒中的绿藻大病毒具有较密切的亲缘关系。
系统发育分析表明,在进化地位上,DSLPV2/3/4同样与绿藻大病毒Prasinovirus属距离较近,而DSLLAV1则作为Mimiviridae家族外支与Mimiviridae的距离较近,表明DSLLAV1是一类进化地位较为特殊的绿藻大病毒,并且滴水湖中藻类大病毒具有一定的多样性。
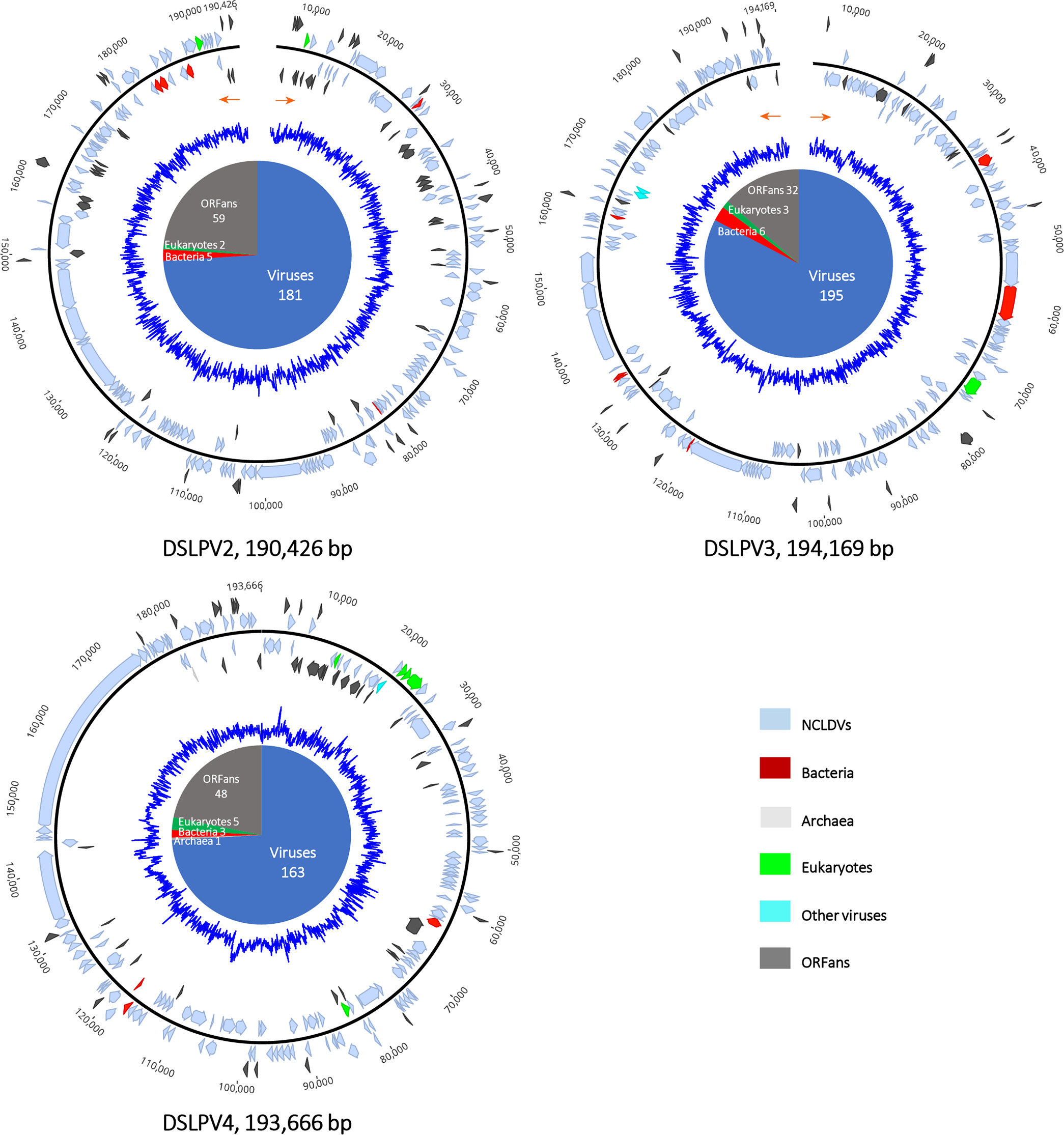
图3 藻类大病毒DSLPVs基因组图谱
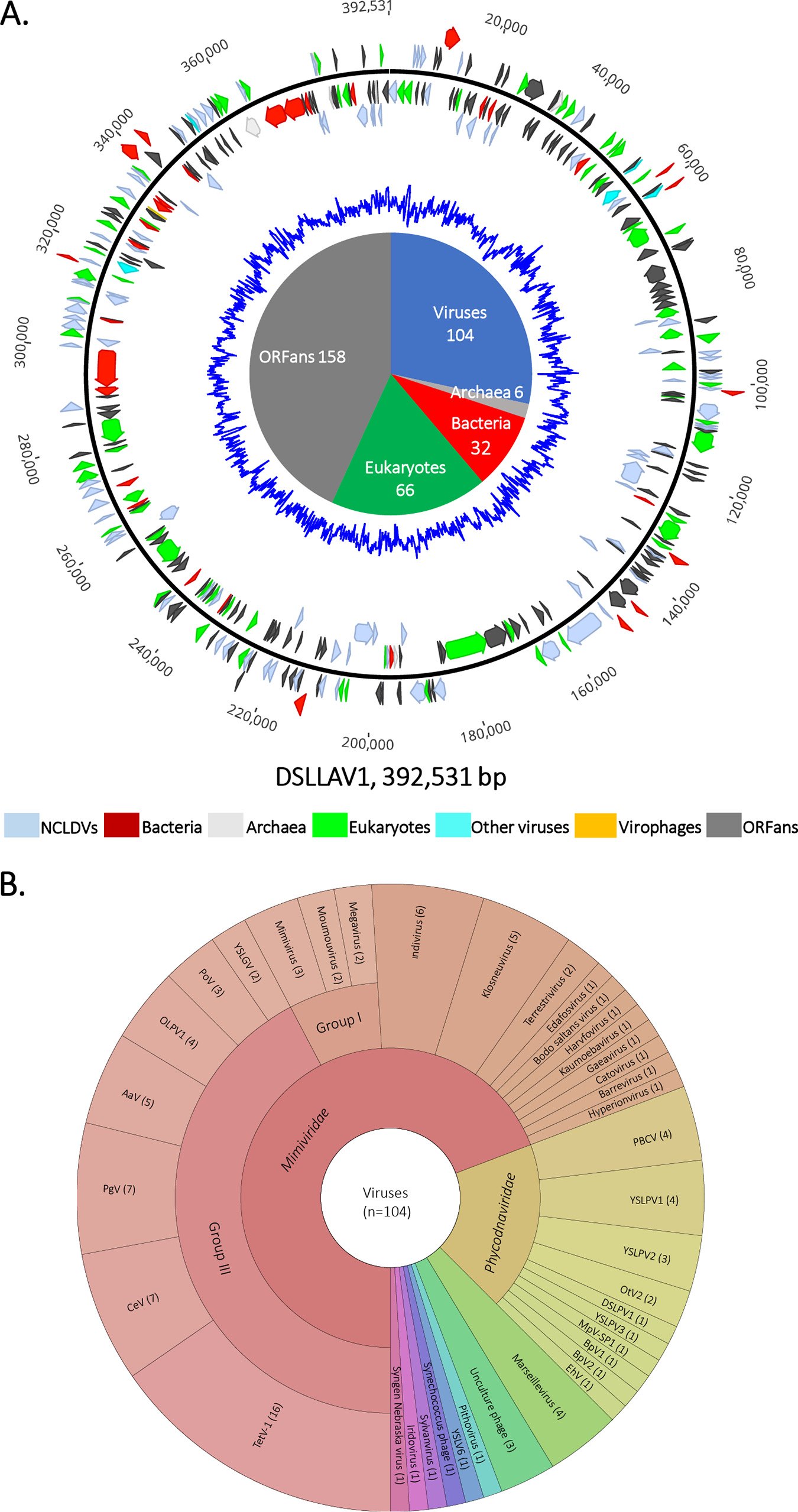
图4 藻类大病毒DSLLAV1基因组图谱(A)和与病毒相关的基因注释信息分类(B)
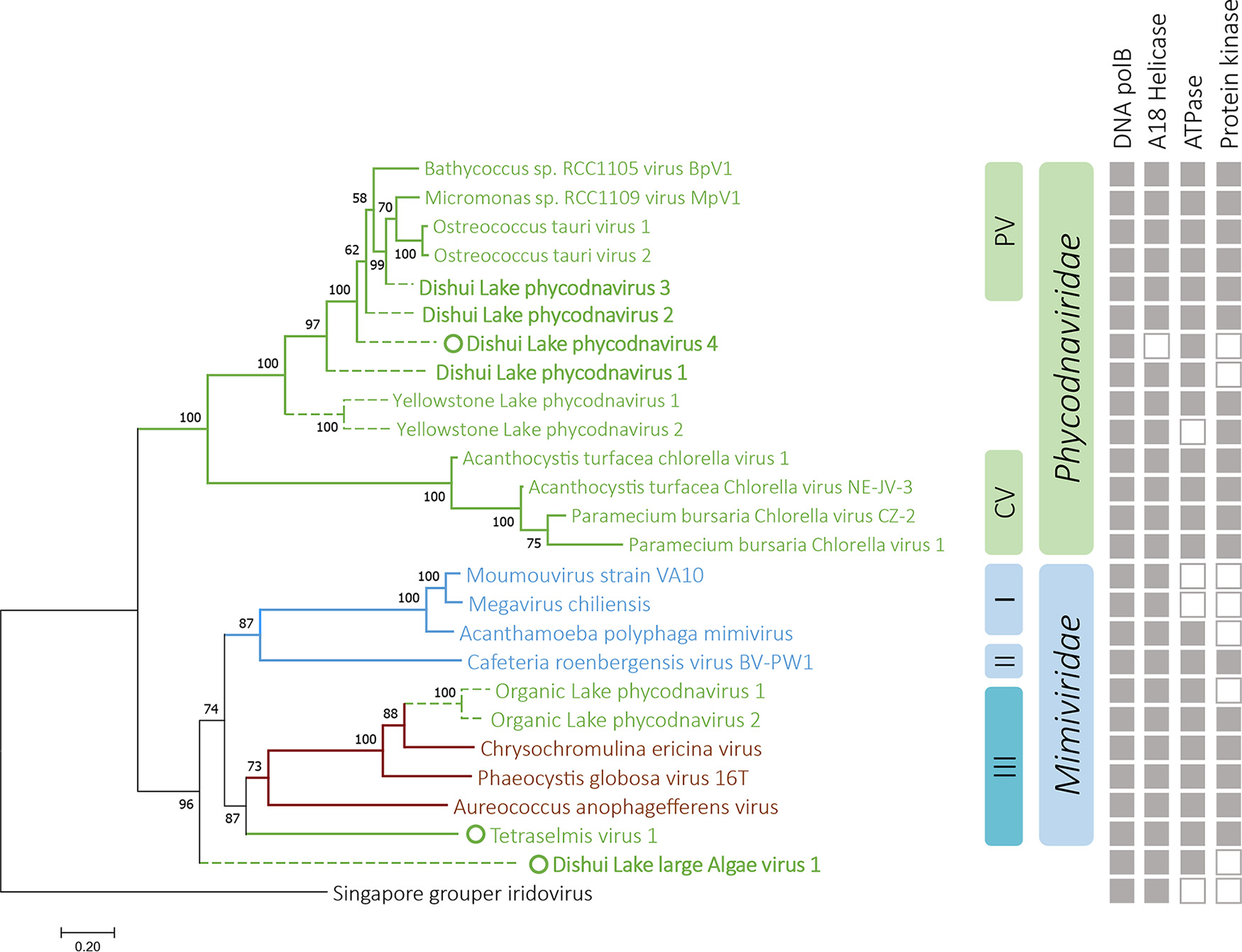
图5 DSLPVs与DSLLAV1系统发育分析
03、噬病毒体与巨/大病毒基因关联性分析
图6中将噬病毒体与巨/大病毒共享的同源基因进行梳理,揭示噬病毒体与其潜在病毒宿主在基因水平上的关联性。以图6B中已建立的C-V-v系统为对照,其中噬病毒体均与其宿主巨病毒具有较好的基因关联,如Sputnik与Mamavirus,Mavirus与Cafeteria roenbergensis virus(CroV)等,且同源基因的进一步系统发育分析同样支持这种关联性(图6D)。通过分析发现,滴水湖中噬病毒体与藻类大病毒尤其是绿藻大病毒具有更好基因关联,如DSLV1/4/6/7与Paramecium bursaria Chlorella virus(PBCV)(图6A),并通过共享同源基因的系统发育分析重现了这种关联性(图6C)。由此表明,滴水湖噬病毒体可能以单细胞绿藻大病毒为病毒宿主并与其一同侵染绿藻细胞。
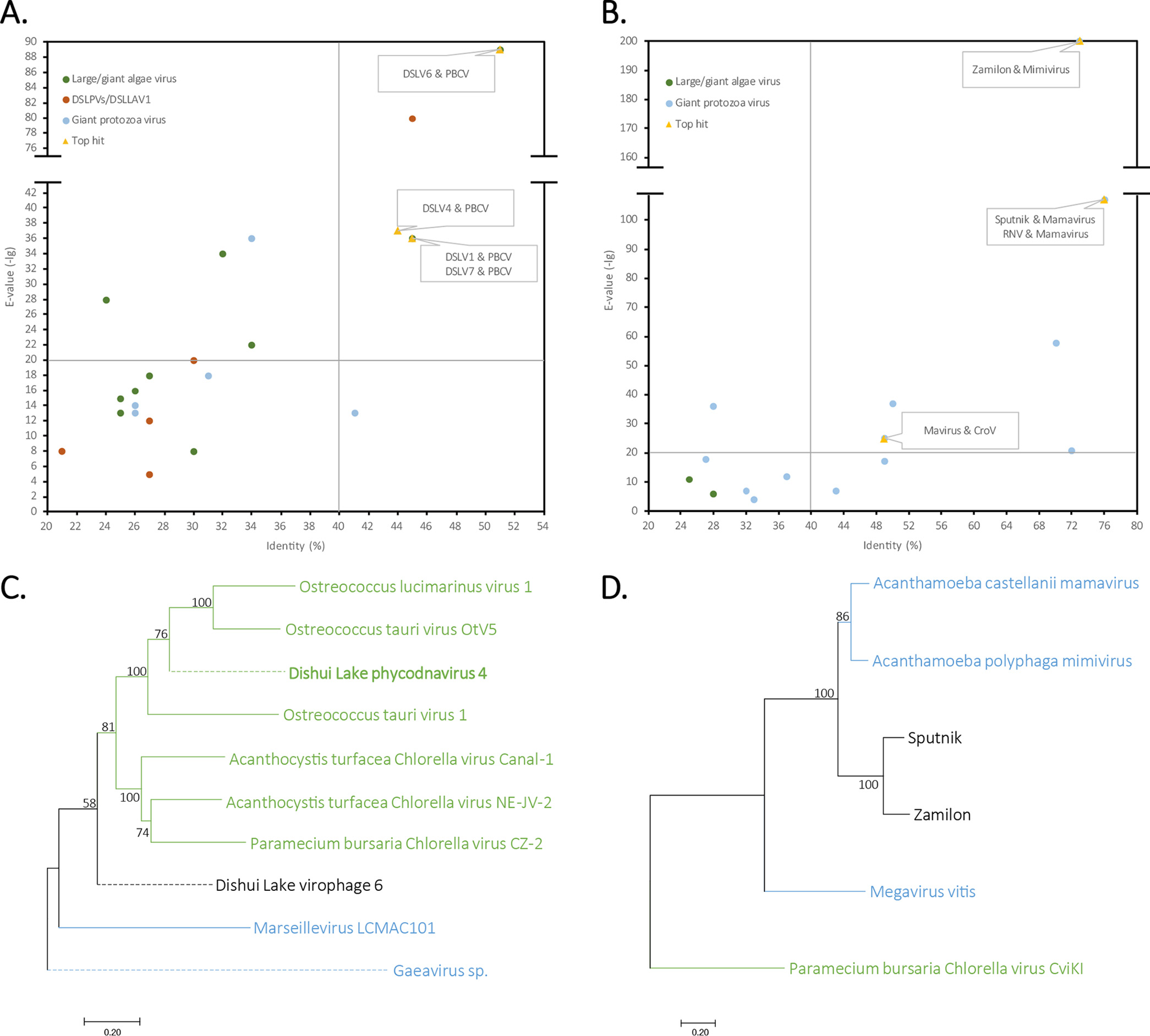
图6 噬病毒体与巨/大病毒的基因关联性
04、噬病毒体与巨/大病毒基因组特征分析
如图7A所示,八个滴水湖噬病毒体共被分为两组。其中,DSLV1-4、4-7的密码子使用模式不仅与侵染绿藻的PBCV-1和TetV-1非常相似,也与Prasinovirus属的其他绿藻病毒、DSLPV4和DSLLAV1非常相似。其中,DSLV5和DSLV8的密码子使用模式则与侵染原生动物的巨病毒(Mimiviridae Group I and II)和侵染藻类的巨/大病毒(Mimiviridae Group III)更为相似。和预期的一样,在对照组中,Sputnik、Zamilon 和Mavirus则分别与它们的宿主病毒Acanthamoeba castellanii mamavirus9(ACMV)和CroV聚在一起,验证了该分析方法的可靠性。综合所有结果表明,侵染绿藻的大病毒包括在滴水湖中发现的绿藻大病毒(如:DSLPV4,DSLLAV1及它们的近亲病毒)更可能是滴水湖噬病毒体的潜在病毒宿主。
与密码子相对使用频率分析结果一致,寡核苷酸相对频率分析结果同样表明,侵染绿藻的大病毒更可能是滴水湖噬病毒体的潜在病毒宿主。如图7B、C所示,DSLVs的二核苷酸相对频率模式与侵染绿藻的大病毒(如PBCV-1、TetV-1、DSLPV4和DSLLAV1)非常相似,但显然不同于对照组中侵染原生动物的巨病毒(如ACMV和CroV)。
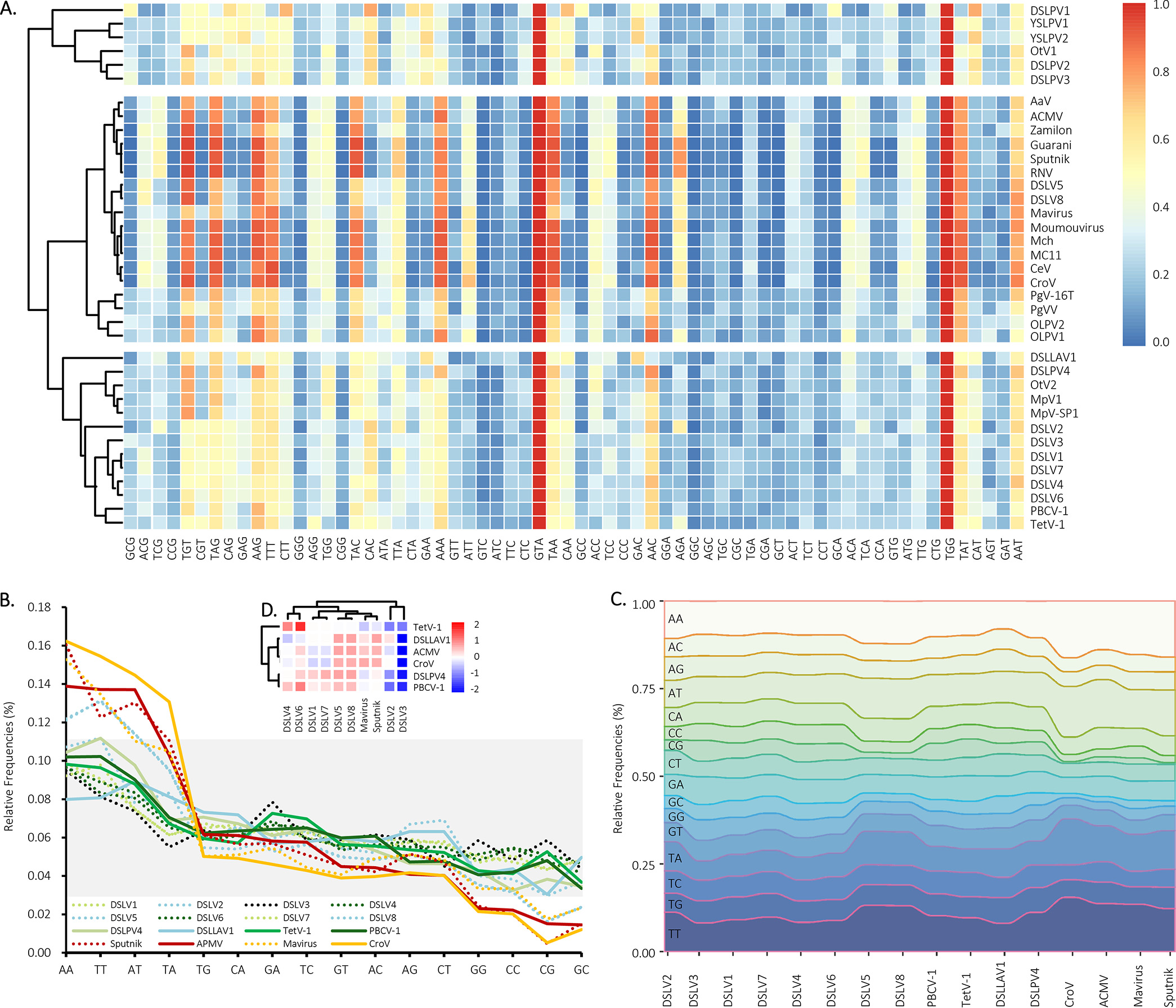
图7 噬病毒体与巨/大病毒之间的基因组特征分析
05、滴水湖大病毒(DSLLAV1)CRISPR-Cas-like系统分析
如图8A所示,分别来自DSLV5 ORF24和DSLV8 ORF24的长度为20 bp的核酸序列片段匹配(DSLV5一个错配,DSLV8无错配)到DSLLAV1基因组上(ORF142)。同时在匹配处的上游和下游存在47 bp的重复序列,且在这个重复序列中,另有8 bp在47 bp中重复了4次,在20bp中重复了2次。此外,在DSLLAV1的ORF142上游分别发现了可编码类Cas4和Cas9蛋白的基因。综上所述,本研究认为,DSLLAV1拥有一个可以防御噬病毒体DSLV5和DSLV8侵染的类CRISPR-Cas的系统,并将其命名为藻类大病毒噬病毒体防御系统LAVVIRE(Large algae virus virophage resistance element)。
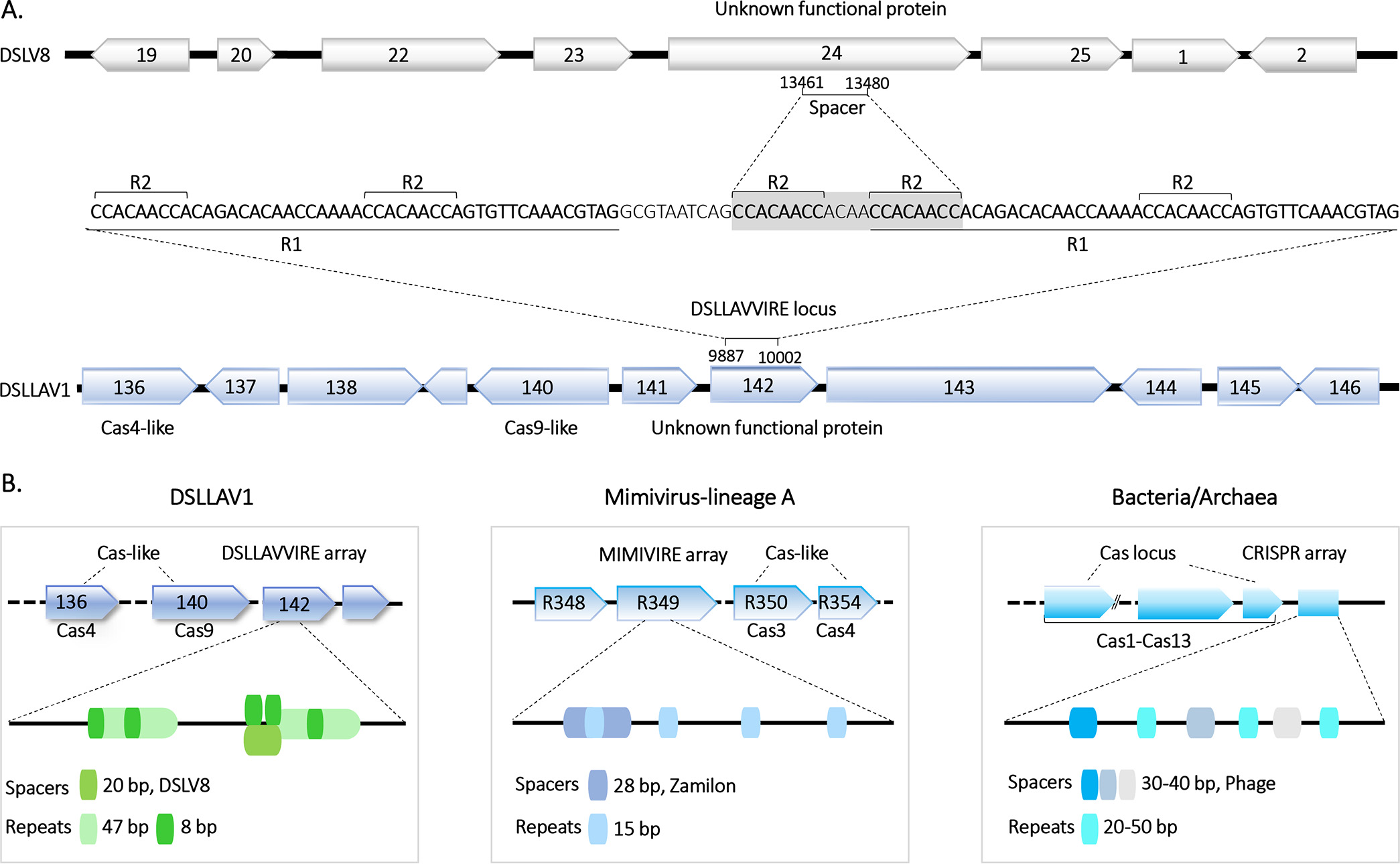
图8 DSLLAV1中的CRISPR-Cas-like系统
06、滴水湖中新型噬病毒体—绿藻大病毒—单细胞真核绿藻三相互作系统
目前已分离的噬病毒体(如Sputnik、Zamilon等)必须依赖宿主巨病毒才能进行正常增殖,并且噬病毒体与其宿主巨病毒共享相似度较高的同源基因。本文通过比较基因组学分析发现,滴水湖中噬病毒体与绿藻大病毒共享相似度较高的同源基因,并进一步通过基因组特征分析揭示了滴水湖噬病毒体与绿藻大病毒之间存在的共同进化关系。在DSLLAV1基因组中发现的抵抗噬病毒DSLV5/8侵染的CRISPR-Cas-like系统再次证实了噬病毒体与绿藻大病毒由来已久的互作关系,所有分析表明滴水湖中噬病毒体与绿藻大病毒、单细胞真核绿藻共同构成了新型Cell-Virus-Virophage三相互作系统。
总结
本研究通过水体宏基因组学的分析方法,探究了上海市滴水湖表层湖水中噬病毒体和藻类大病毒的多样性及其互作关系。通过病毒序列的调取及基因组序列拼接,共获得了多条新型噬病毒体和藻类大病毒的基因组序列,并综合运用基因关联分析(Genetic link)、基因组特征分析(Genomic signature)和CRISPR-Cas系统(CRISPR-Cas system)分析等方法,揭示了滴水湖中新型噬病毒体与绿藻大病毒、单细胞真核绿藻之间的三相互作侵染系统。
本研究的测序工作由上海派森诺生物科技股份有限公司完成。
文章索引:
Xu S, Zhou L, Liang X, et al. Novel Cell-Virus-Virophage Tripartite Infection Systems Discovered in the Freshwater Lake Dishui Lake in Shanghai, China[J]. Journal of Virology, 2020, 94(11).
原文链接:https://jvi.asm.org/content/94/11/e00149-20