
2022-03-28

期刊:《Microorganisms》
影响因子:4.087
派森诺与华中农业大学动物医学院合作,在微生物基因组领域的《Microorganisms》发表新研究成果!本研究检测了九种肠道沙门氏菌亚种,分离出毒性最强的肠炎链球菌211(SE211)。本研究首次获得肠球菌高毒力分离株SE211完整基因组序列,并对其进行比较基因组学和系统发育分析,为进一步研究肠球菌遗传学提供了初步的认识。
研究背景
沙门氏菌是一种广泛存在的人畜共患病病原体,通过受感染的牲畜和家禽引起人类食物中毒,沙门氏菌病例是全球细菌性食源性疾病最常见的原因之一,在世界范围内造成了巨大的经济损失。沙门氏菌是一个高度多样化的属,包括两个物种(邦戈里沙门氏菌和肠道沙门氏菌),其中肠道沙门氏菌被认为是最具致病性的物种。在分类学方面,一方面,肠道沙门氏菌可分为六个亚种,并有2600多个血清型。另一方面,根据沙门氏菌引起的人类疾病,沙门氏菌主要分为两类:伤寒沙门氏菌血清型和非伤寒沙门氏菌(NTS)血清型。
近年来,随着抗生素试剂的广泛应用,沙门氏菌的耐药问题日益突出。特别是沙门氏菌的耐药菌株在一些发展中国家造成了相当大的经济损失,甚至是造成人类死亡。因此研究沙门氏菌潜在的毒力机制,并积极开发有效的治疗方法,对解决其致病性具有重要意义。虽然鼠伤寒沙门氏菌的治病机制已被研究,但许多其他肠道沙门氏菌的致病性仍不清楚。本研究分离得到9株沙门氏菌,通过全基因组测序和比较基因组分析揭示其毒性机制,深入了解该菌基因组中潜在的致病机制。
研究材料与方法
1.实验材料:从鸡肠中分离得到9株肠道沙门氏菌分离物,选择毒性最强的菌株SE211进行高通量测序。
2.测序平台:Illumina NovaSeq、PacBio Sequel
3.分析内容:细菌完成图测序、毒力基因预测、ANI分析、比较基因组圈图、进化树构建、MLST、qPCR实验等。
研究结果
致病性分析及关键毒力基因表达
根据图1A所示实验设计进行急性感染实验,检测九种分离株的毒力表型、细胞入侵和生物被膜,结果表明:分离菌株SE211具有强毒力表型(对鸡的致死率为80%,细胞入侵率高,生物膜形成中等),这与其体内毒力基因spvB、avrA、bcfC和rck高表达一致。因此,本文认为SE211的毒力机制可能高度激活,并与其高致死性和细胞入侵能力(多种毒力特征)相关,因此它可能是阐明潜在致病机制的良好模式菌株。
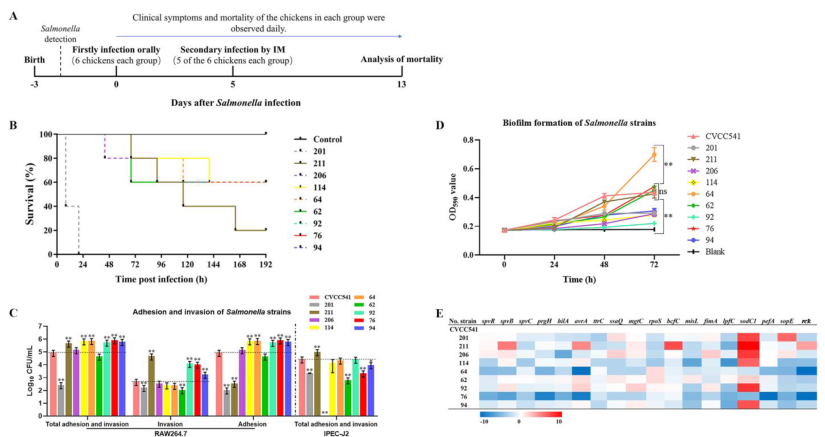
图1九株肠氏沙门分离菌株的基本毒力特征
SE211基因组一般特征分析
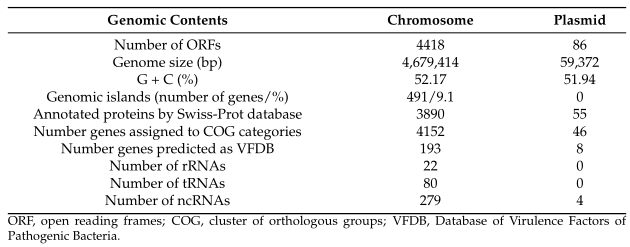
基于上述体外内研究,SE211更倾向于在遗传水平上表现出毒力机制。SE211的基因组由一条染色体和一个含有两个复制子的毒力质粒组成。通过MLST分型法,SE211被鉴定为ST11。SE211染色体的基因组长度为4679414 bp,质粒长度为59372 bp,预测的ORF分别为4418个和86个,GC含量分别为52.17%和51.94%。在染色体基因组中,约9.1%的全基因组长度(491个基因)预测出为基因组岛区域;4418个ORF中的193个基因被预测为致病菌的毒力因子。
表1 SE211基因组基本信息
可移动基因元件分析
对于SE211菌株分析chr区域被称为SPI的毒力相关GIs的结构和主要功能。而对于毒力质粒blast分析表明其与RST的S1:A-:B22的不相容性(Inc)类型F相匹配;该质粒中含有编码关键毒力因子的基因,以及大量已知毒力相关基因和一个未知功能性毒力基因(vsdF),且毒力基因两侧是一些整合移动基因元件(IMGE)。这些存在于细菌基因组中的溶原性噬菌体和移动基因元件(MGE)可能编码CRISPR-Cas系统的抑制基因,称为抗CRISPRs。有趣的是,由于HGT事件可能有助于毒力因子的转移和抗生素耐药性的发展,抗CRISPR可能会影响细菌的发病机制。
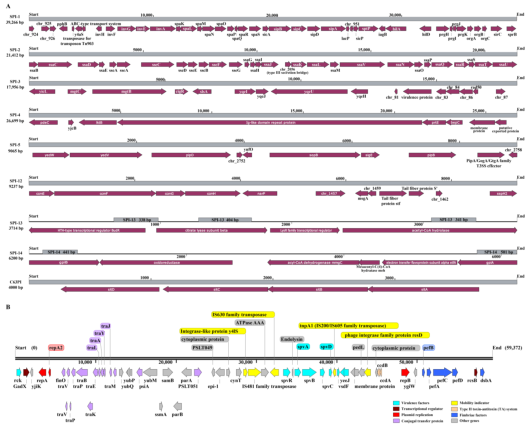
图2 SE211基因组中沙门氏菌致病岛(SPI)和质粒的基因组分析
比较基因组分析
基于ANI分析得到SE211与四种食源性肠炎沙门氏菌株最为相似。系统发育树表明这些菌株的聚类关系与其血清型一致,值得注意的是,SE211聚集在两种食源性菌株(P125109和EC20121179)附近。特别是P125109是一种人类食物中毒肠炎链球菌菌株,最初在英国分离,可追溯到一个家禽养殖场。该树还揭示了肠炎链球菌、鸡链球菌和鼠伤寒链球菌血清型的高度遗传异质性。
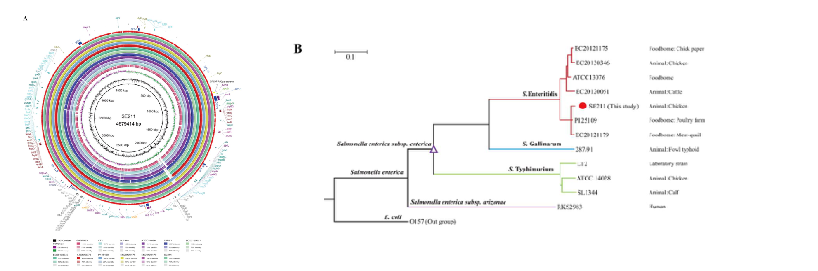
图3 12种沙门氏菌基因组比较和聚类分析
全基因组测序和分析
使用从NCBI下载的12株肠道沙门氏菌菌株(11株肠道沙门氏菌,包括SE211和亚利桑那州沙门氏菌)和243株肠炎沙门氏菌基因组来执行基于cgMLST的MST图。共分成九个MST集群,SE211属于MST集群2。除SE211外,从腹泻患者中分离的两株肠炎沙门氏菌SE104和SE109以及一株从临床分离的菌株也包括在该群中。SE211肠炎沙门氏菌与其他血清型菌株之间的距离很远,而与11个选定菌株的基因组相似,这归因于它们相同的生物学特性和进化环境。
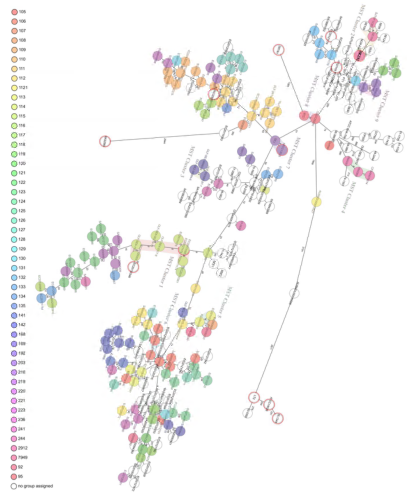
图4 255份肠道沙门氏菌基因组样本的最小生成树(MST)图
研究结论
1.全基因组测序进行的基因型分析可用于了解致病机制,包括与暴发相关的菌株的毒力、耐药性和分子进化;
2.基因组SE211编码许多毒力因子,尤其是位于含有毒力质粒、31个GIs和五个原噬菌体区域的MGE中;重要的是,这些MGE伴随着整合酶和转座酶等IMGE;
3.在SE211的基因组中发现了一个完整的CRISPR-Cas系统,这可能有助于提高其毒力;
4.邻接树和MST在这些基因组中显示出明显的距离;
综上所述,分析肠炎沙门氏菌的遗传和表型特征可以提高对致病机制和系统发育的理解,从而为预防和治疗肠道沙门氏菌感染制定新策略。
本研究的denovo测序和部分数据分析由上海派森诺生物科技有限公司完成。如需进一步讨论,欢迎发邮件或者致电我们哟(邮箱地址:microsupport@personalbio.cn,联系电话:025-58185663-836)!
文章索引:Cui LJ, Wang XR, Zhao Y, et al. (2021). Virulence Comparison of Salmonella enterica Subsp. Enterica Isolates from Chicken and Whole Genome Analysis of the High Virulent Strain S. Enteritidis 211. Microorganisms.doi.org/10.3390/microorganisms9112239.