
2022-04-25

《Agronomy-Basel》
影响因子:3.417
派森诺与中国科学院东北地理与农业生态研究所(大豆分子设计与育种重点实验室)合作,在农学基因组领域《Agronomy-Basel》(影响因子3.417)发表新研究成果!本文利用基于SSR的遗传图谱与BSA定位相结合,定位到与大豆两个农艺性状(总花荚数(TFPN)和单株有效荚数(PNPP))相关的基因。
研究背景
大豆(Glycine max (L.) Merrill)是一种重要的种子作物,种子中含有约40%的可食用油和20%的优质蛋白质。该蛋白质包含完整的氨基酸图谱,包括人类健康所必需的八个氨基酸;广泛的种植和高产使大豆成为一种可持续作物,以满足世界范围内对植物蛋白饮食日益增长的需求。因此,产量一直是大豆改良的主要目标。
产量是一个非常复杂的性状,它由许多不同的组成部分组成,如单株荚果数(PNPP)、单株荚果种子数和百粒重,其中PNPP是决定大豆产量的一个特别重要的因素,因为它与单株籽粒产量有很强的相关性(r = 0.84)。研究表明,花或豆荚的总数量(TFPN)或两者都能大大提高大豆产量。因此,了解TFPN和PNPP的遗传机制将有助于阐明这些性状对产量的遗传贡献,对高产大豆品种的培育具有重要意义。
研究材料与方法
1.实验材料:以大豆品种JY73和地方品种TJSLH为杂交材料,共得到100株RIL分离群体。
2.测序策略:分别选取两个极端性状多花多夹(MFP)与少花少夹(LFP)各30个个体进行混池测序,测序深度为30X。
3.分析内容:基于SSR的遗传图谱构建、BSA精细定位分析、候选基因验证等。
研究结果
1.表型鉴定
亲本TJSLH和JY73在TFPN、PNPP、单株种子数和单株产量4个性状上均存在显著差异。简而言之,4个性状JY73表型值高于TJSLH(图1)。这些特征之间的相关分析(表1)表明,与PNPP 、TFPN显示最强的正相关(r = 0.903, p < 0.01),其次是单株产量和种子数/植物(分别为0.833和0.807,p值< 0.01)。PNPP与单株种子数和单株产量呈显著正相关(r = 0.868和0.903,p值< 0.01)。这些结果表明,TFPN和PNPP都是大豆产量的重要组成部分,可能有助于大豆的产量的提高。
以1个MFP品种 (JY73) 和1个LFP地方品种 (TJSLH) 作为亲本,构建了100个RIL群体,用来对其花荚相关性状进行QTL定位。四个性状的表型值分布如图1所示。在JT种群中,R3期TFPN和R8期PNPP的变化幅度较大,分别为50 ~ 300和10 ~ 160。所有表型值分布范围均较大,表明这4个性状都是由多基因定量控制的。此外,所有4个性状在群体中都观察到强烈的越界分离,这表明对测量性状有积极影响的等位基因分布在亲本之间。
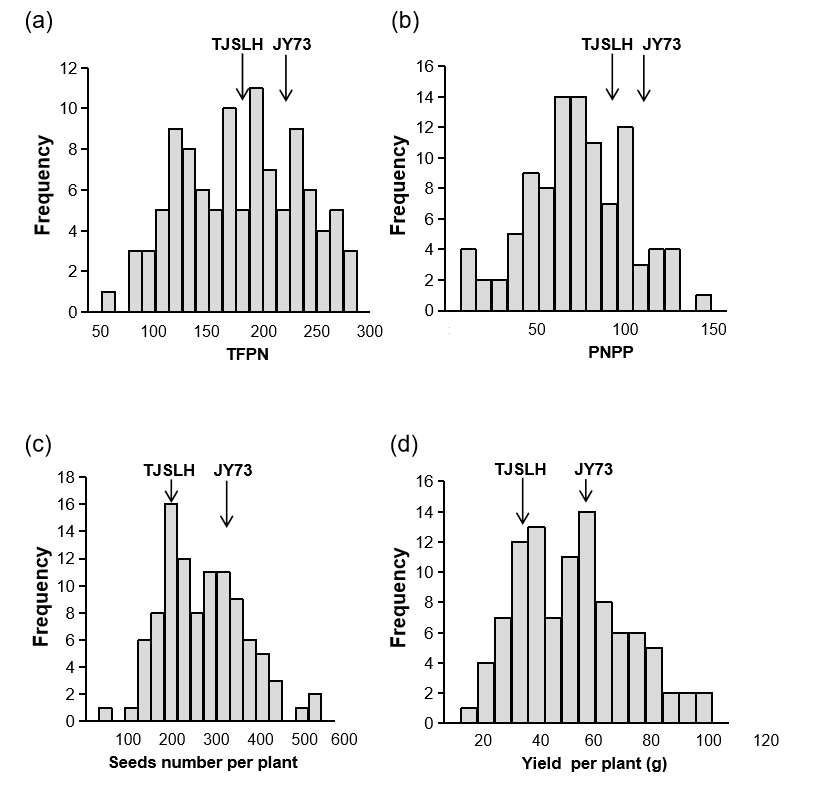
图 1. F2 种群中 TFPN (a)、PNPP (b) 和其他两个农艺性状 (c,d) 的分布。
表 1 大豆 TFPN、PNPP 与产量相关性状的相关系数。

2.TFPN和PNPP两个性状的QTL定位
基于151个标记(138个ssr标记,13个indel标记)进行遗传连锁图谱的构建,一共得到20个连锁群,总图距长度为1351.5 cm,平均图距为11.5 cm。基于该遗传图谱结果进行JT群体花荚数的性状定位,最终在第2、3、4、7和10连锁群上共鉴定出6个控制TFPN的QTL位点,在连锁群2、4、5、7和10上检测到5个控制PNPP 的QTL位点, (表2、图2)。其中,qPN2、qPN7和qPN10在Kuroda et al.(2013)和Ning et al.(2018)中也有发现。在Yang et al.,(2013)中检测到qPN5,这支持了结果。根据不同群体或环境条件下的多个研究,证实了qPN4的稳健表达及其在PNPP中的重要作用。
qPN4对PNPP表型变异的解释率为9.6%,加性效应值为13.89;它也控制TFPN,命名为qFPN4,占TFPN观测变异的9.2%,加性效应值为16.81(表2)。因此,qPN4/qFPN4控制了两个高度相关的性状:PNPP和TFPN。目前,在这一区间内控制花荚相关性状的基因尚未在大豆中被报道。
表 2 TFPN 和 PNPP 的 QTL 信息
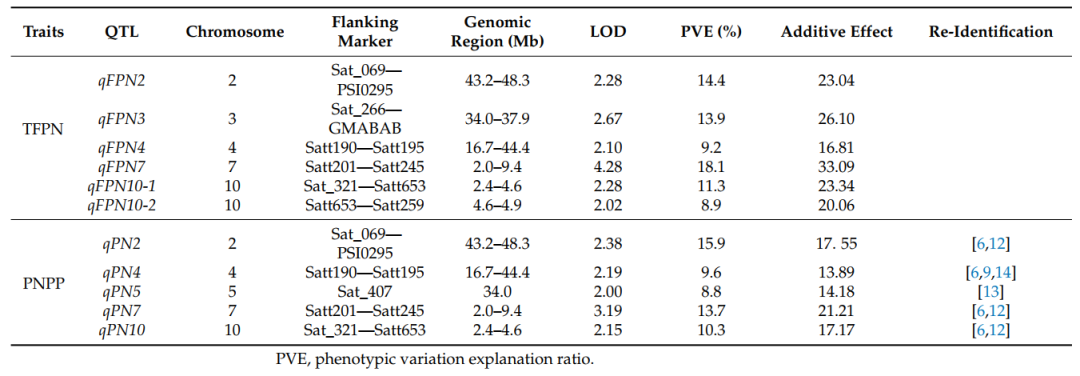
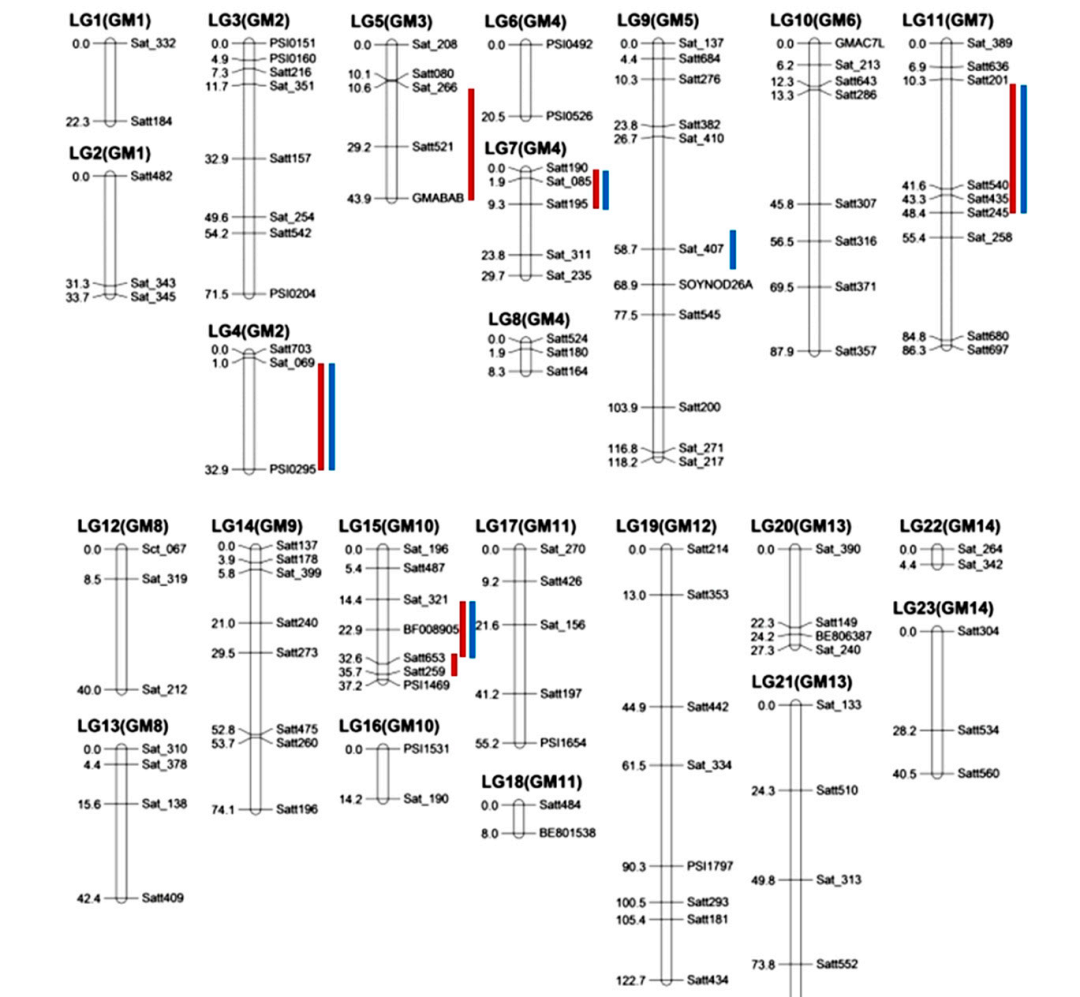
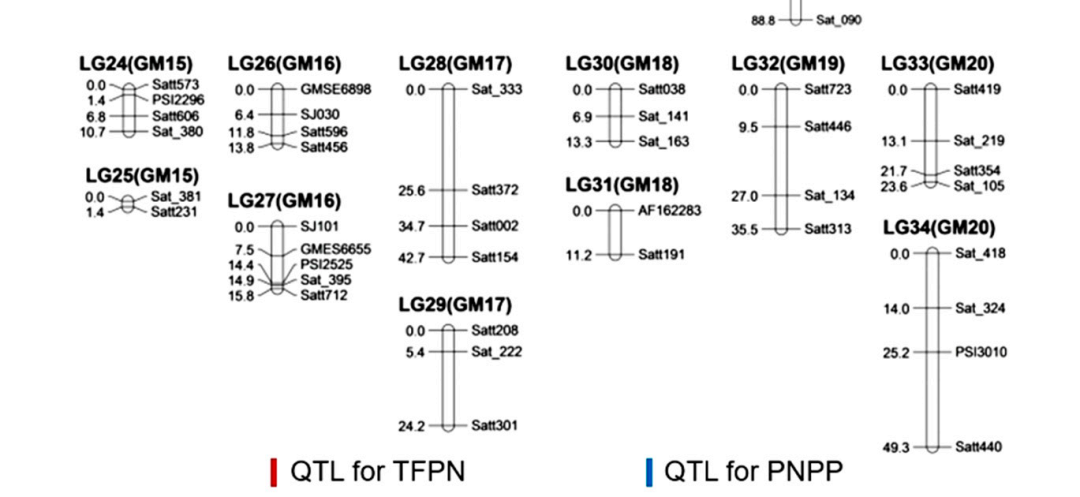
图 2. RIL 群体的遗传图谱和TFPN和PNPP的QTL。RIL群体的遗传图谱和TFPN和PNPP的QTL。
3.基于BSA分析进行候选基因的精细定位
为了探究qFPN4这个区间内是否有关键候选基因,利用具有极端表型的TJSLH (NIL-qFPN4) 、 JY73 (NIL-qfpn4)的后代构建遗传群体进行BSA性状定位。将来自30个TFPN和PNPP高的个体的DNA混合成为MFP混池。同样,将30个TFPN和PNPP低的个体DNA混合形成LFP混池。为了找到与性状相关的关键基因,对这两个混池进行了高通量全基因组测序。测序共获得33.28 G的原始数据,质控后高质量数据占比为93.44%, MFP和LFP分别有98.34%(208,615,358)和98.67%(213,395,128)的reads比对到参考基因组上。比对上的reads在参考基因组中的平均覆盖度为95.44%,平均测序深度为26×。
采用ED算法分析MFP和LFP之间显著SNP/Indel性状关联区域。关联阈值为ED=1.0时,在RHLs确定的qFPN4的物理位置(Chr.4: 41,754,458 - 445,173 bp),在Chr.4: 39,889,438 - 454,442,849 bp处发现了一个具有显著信号的基因组区域(图5)。该区域的变异为候选基因的筛选提供了有用的变异信息。
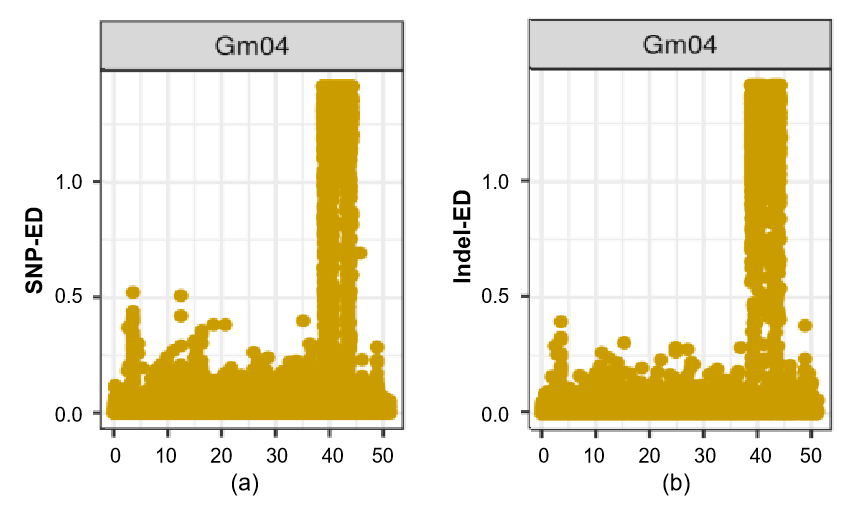
图3. ED相关值在4号染色体上的分布。注:彩色点代表每个SNP (a)或Indel (b)的ED值。
4. 候选基因的鉴定与验证
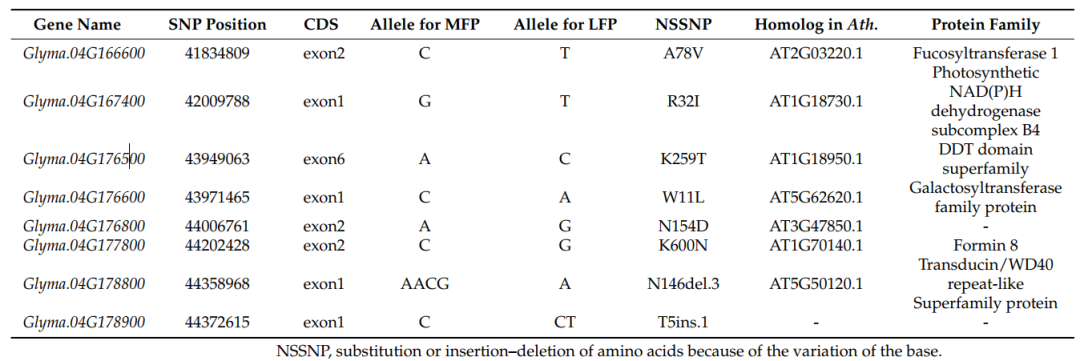
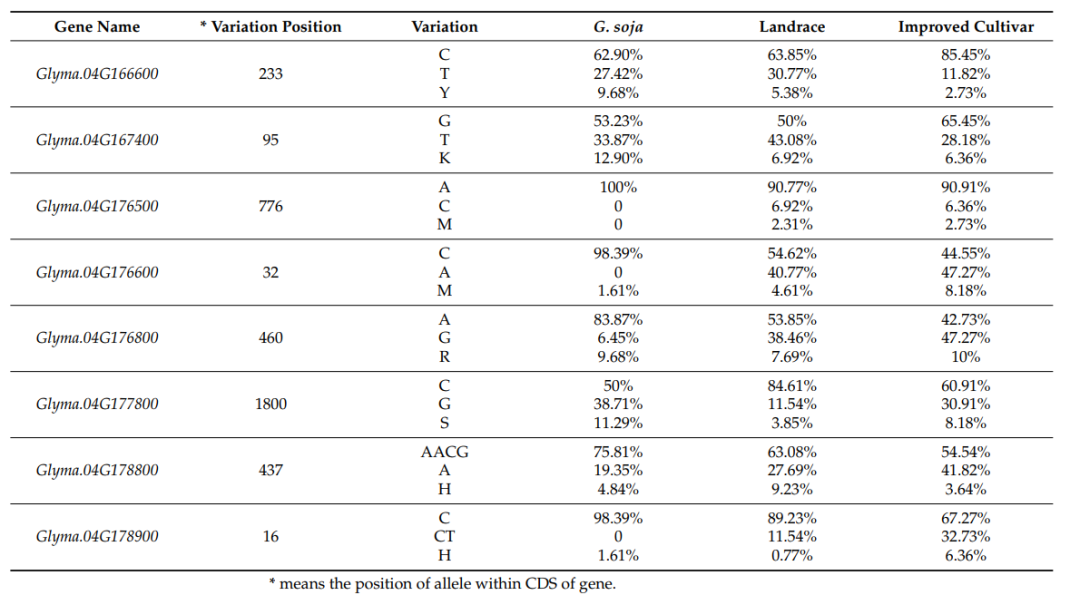
基于SNP/ indel的BSA分析和QTL定位结果(图3和图5)识别了一个约2.62 M的交叉区域,该区域位于Gm04: 41,754,458 - 444,375,173 bp。根据Williams 82这个参考基因组序列可知该区域包含127个基因,其中8个基因在两个区段之间存在SNP或InDel的非同义突变(表3)。特别是Glyma.04G178900基因上的单碱基插入(C:CT)导致5氨基转移造成蛋白功能缺乏。
表3在参考Williams82序列中预测qFPN4位点~2.62 Mb区域内的基因。
为了验证性状关联的关键基因,从不同发育阶段收集的不同组织(包括根、茎、叶、芽、发育中的豆荚和种子)的来探究基因的表达模式。结果显示在不同组织中有不同的表达模式,其中Glyma.04G176400在茎、叶和子叶中表达量最高,表明Glyma.04G176400在营养生长期起着重要作用。其余基因Glyma.04G176600和Glyma.04G178900在花期(Flower5)和早期荚果(Pod_seed1、Pod_seed2、Pod_seed3、Pod1、Pod2和Pod3)相对于其他组织高度表达(图6)。这表明这两个基因可能是参与调控花和荚果发育的关键基因。
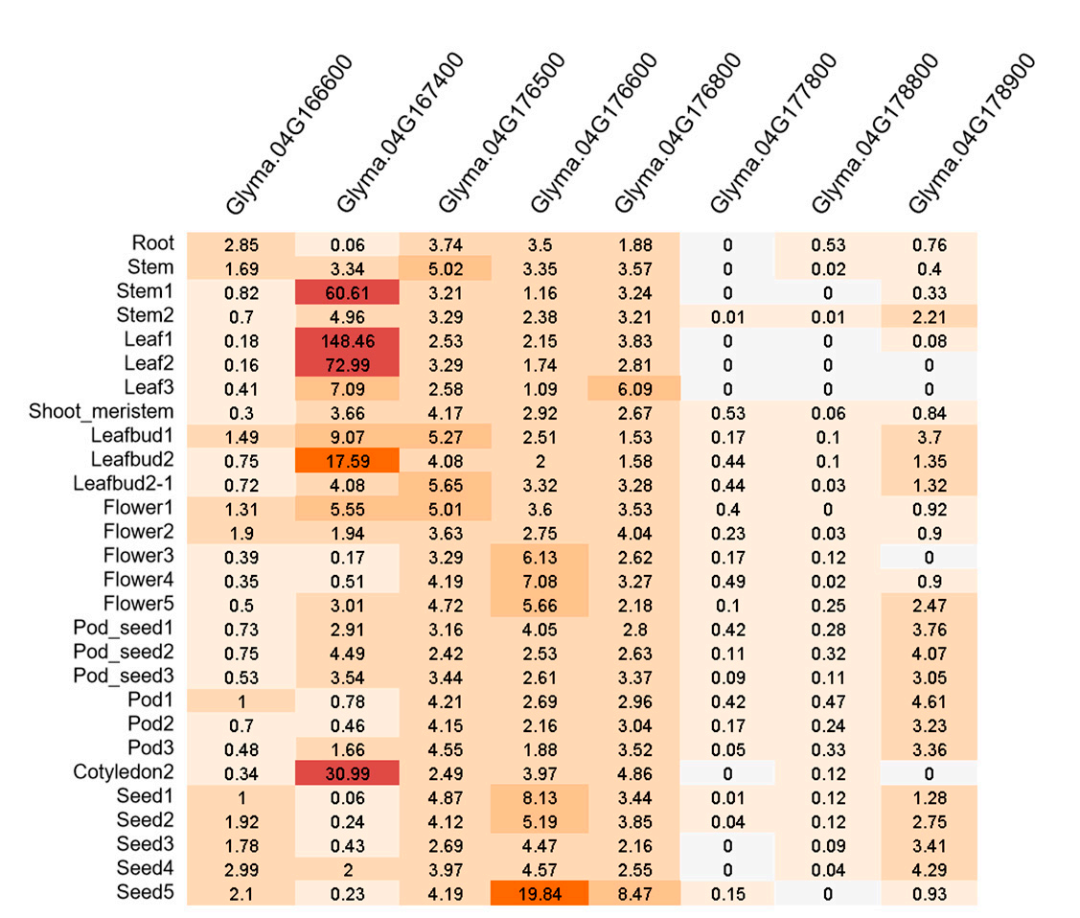
图4. 8个基因在不同组织中的表达模式及相对水平。
5.从遗传变异角度对上述定位基因验证
对302份具有代表性的大豆品种(包括已测序的野生大豆、地方大豆和栽培品种)进行了遗传变异分析。如图5和表4所示,基因型分析显示了8个候选基因在群体中这些非同义snp或Indels的分布和频率的差异。这8个基因的等位基因可能在大豆驯化和发育过程中对花和荚数的发育起作用,表明这些基因能够满足人类对更高大豆产量的需要。
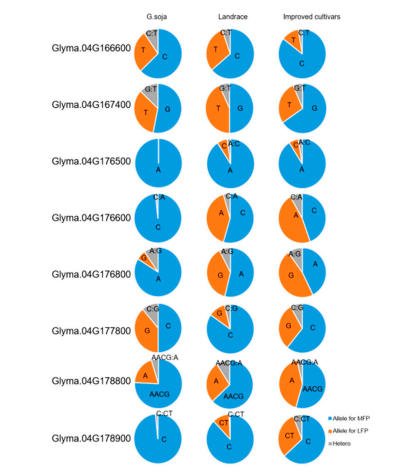
图5. 大豆、长白及改良品种中8个基因的等位基因频率。
表4 群体中8个基因的变异信息和百分比
结论
本研究发现一个与TFPN和PNPP性状关联的关键QTL且这两个性状是由一个单显性基因控制的,其中qFPN4等位基因控制低TFPN和PNPP,而qfpn4控制高TFPN和PNPP。关键QTL-qFPN4是一个~2.62 Mb的区间(Gm04: 41,754,458-44,375,173)。在这一区间内,有8个基因携带非同义突变的SNP和/或InDels。根据基因表达模式分析可知Glyma.04G176600和Glyma.04G178900为关键候选基因。此外,本研究还开发了一个与该QTL-qFPN4紧密连接的InDel标记C1-5,可以通过标记辅助选择促进大豆的改良。本研究结果证实了qFPN4对大豆产量的影响,丰富了大豆改良的候选QTL。
文章索引:
Sun, X.; Sun, X.; Pan, X.;Zhang, H.; Wang, Y.; Ren, H.; Wang, F. Identification and Fine Mapping of a Quantitative Trait Locus Controlling the Total Flower and Pod Numbers in Soybean. Agronomy 2022, 12, 790. https://doi.org/10.3390/ agronomy12040790