2022-07-04

《Nucleic Acids Research》
影响因子:19.16
上周,派森诺生物与上海师范大学合作,于国际著名期刊《Nucleic Acids Research》发表了拟南芥叶绿体中基因转录翻译新机制的研究成果。
研究背景
在大肠杆菌中,转录-翻译偶联是由NusG介导的。尽管叶绿体是内共生原核生物的后代,但叶绿体中这种耦合的机制仍不清楚。在此,我们揭示了叶绿体中通过AtNusG进行的转录-翻译偶联机制。
研究结果表明,AtNusG介导的叶绿体中转录和翻译之间的耦合确保了植物生长的光合作用能力的快速建立和对环境变化的反应。因此,我们的研究揭示了叶绿体和大肠杆菌之间转录-翻译耦合的保守机制,它可能代表了叶绿体基因表达的调节机制。这项研究为高等植物中叶绿体基因表达的基本机制提供了新的见解。
研究路线
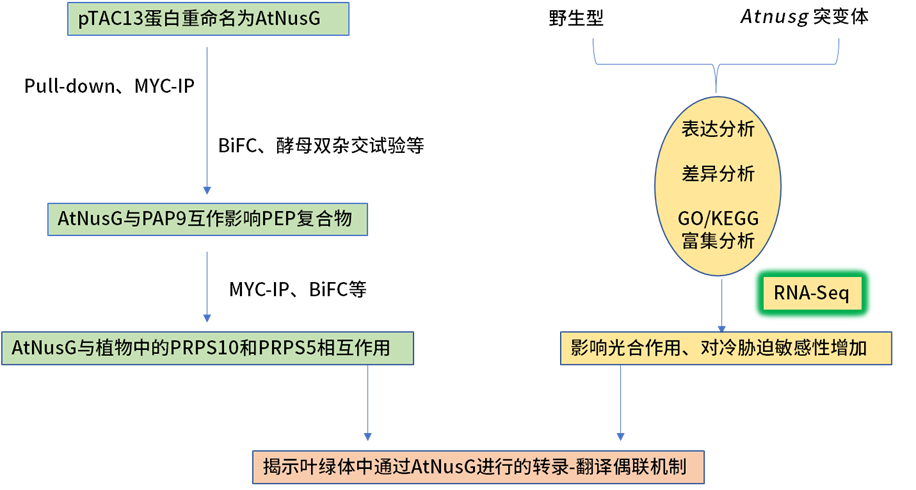
研究内容
1.拟南芥中AtNusG与叶绿体PEP复合物相关
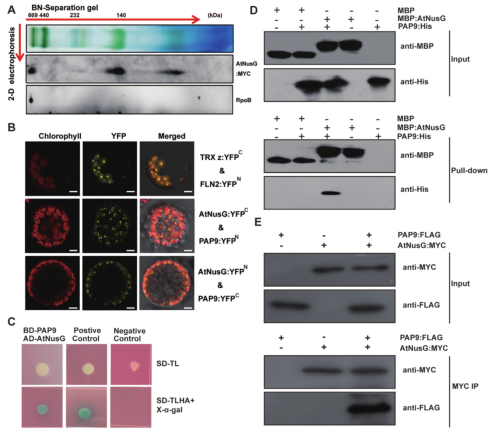
通过实验验证,pTAC13活性和结构与大肠杆菌NusG相似,故我们将pTAC13蛋白重命名为AtNusG。并通过SDS-PAGE、免疫印迹分析和BiFC等实验验证了AtNusG是通过与PAP9相互作用而与PEP复合物相关的。
2.AtNusG与叶绿体核糖体结合,并影响光合作用
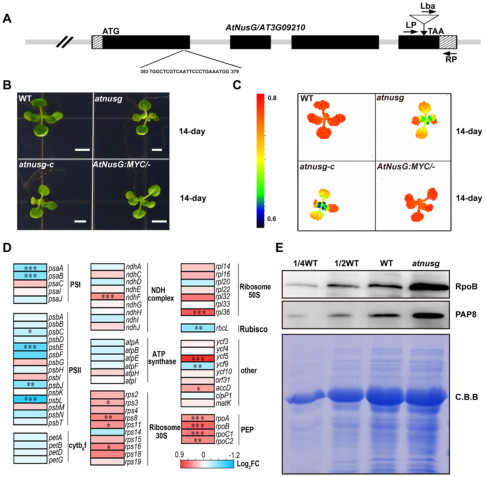
WB和BiFC显示, AtNusG与植物中的PRPS10和PRPS5相互作用,这也表明AtNusG与植物中的叶绿体翻译机制密切相关。且敲除AtNusG基因可以延迟拟南芥叶绿体暗向光转变过程中的发育,并影响拟南芥叶绿体类囊体膜系统的形成,进一步影响光合作用。
3.敲除AtNusG改变了叶绿体的转录谱和叶绿体编码的光合蛋白
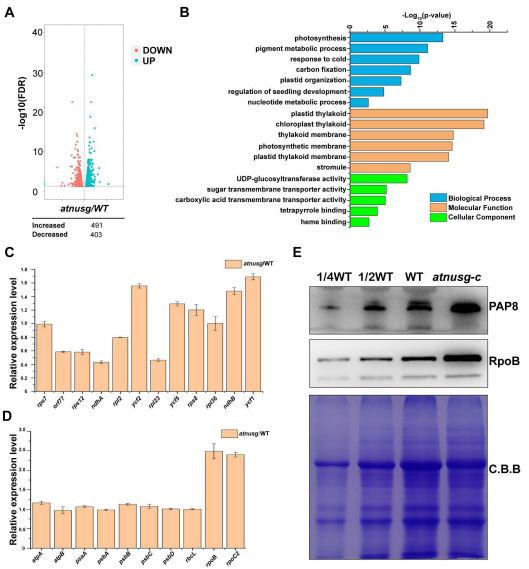
为了研究AtNusG突变对基因表达的影响,我们进行了转录组测序,比较了AtNusG突变体和野生型植物基因表达的区别。结果表明,与野生型相比,atnusg有894个基因表达显著差异(P < 0.05)。其中403个基因表达下调,491个基因表达上调。GO富集分析显示,这些基因参与RNA光合作用、色素代谢过程和质体组织等。与光合作用相关的质体基因psbA、psbB、psbC和psbD的差异变化不明显,而其他转录本如rpoB和rpoC2的转录本水平增加,RT-qPCR验证实验与转录组结果一致。
此外,我们的RNA测序分析显示,在nusg突变体中,这些pep依赖的叶绿体转录本的编码序列(例如psbA、psbB、psbC和psbD)中没有发生插入/缺失突变。
4.atnusg突变体的叶绿体翻译受损
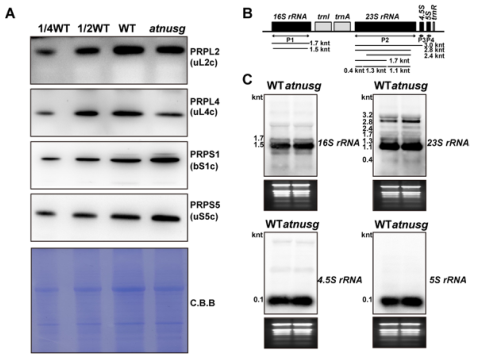
免疫印迹分析显示,与野生型植物相比,atnusg突变体中PRPL2 (uL2c)和PRPL4 (uL4c)的积累减少,而PRPS1 (bS1c)和PRPS5 (uS5c)的数量没有明显变化。RNA凝胶印迹分析显示,atnusg和atnusg- C突变体的23S rRN A、16S rRN A、4.5S rRN A和5S rRN A的成熟形态与野生型相比均未发生明显变化。这些数据表明,敲除AtNusG会损害这些质体编码的光合基因的叶绿体翻译。
5.AtNusG的缺失诱导了对冷胁迫的敏感性
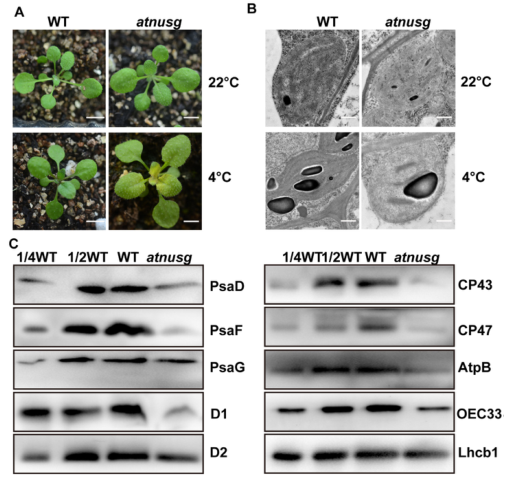
据报道,质体核糖体活性缺陷的植物比野生型植物对低温处理更敏感。我们对atnusg突变体和野生型做了一系列冷胁迫实验。结果显示,AtNusG失活后,幼叶叶绿体发育受到了严重的损害。与野生型相比,atnusg突变体的核糖体蛋白数量也显著减少。这些结果也表明,敲除AtNusG基因可以增强植物对冷胁迫的敏感性,类似于质体核糖体活性或核糖体生物发生缺陷的突变体,这与AtNusG突变体中叶绿体翻译能力受损的结论一致。
总 结
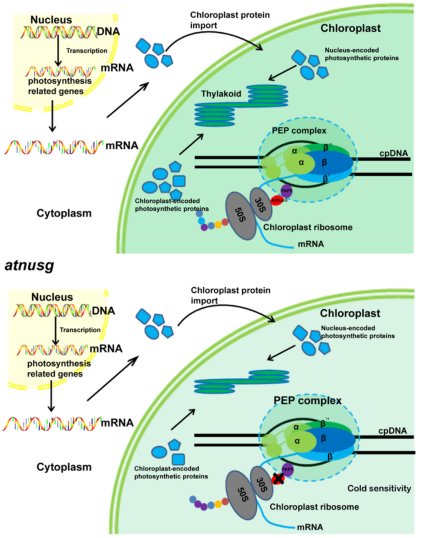
AtNusG在叶绿体中的作用模型
在文章的最后,作者提出了AtNusG在叶绿体中的作用模型。该模型显示,AtNusG耦合了叶绿体PEP复合体和叶绿体核糖体,以保证叶绿体编码的光合蛋白的快速合成。在atnusg突变体中,解耦降低了这些光合蛋白的翻译效率,不能满足叶绿体快速发育和在新生幼叶中建立光合作用能力的需要,而冷应激加剧了对叶绿体发育的影响。
本研究的RNA测序和分析工作由上海派森诺生物科技有限公司完成。
原文索引:https://doi.org/10.1093/nar/gkac501


